Мини-обзор генома бактерии Enterococcus mundtii
Чингариева Алия
Студентка первого курса факультета биоинженерии и биоинформатики МГУ им. М. В. Ломоносова,
2023 год- Введение
- Материалы и методы
-
Результаты
- Гистограмма длин белков E. mundtii
- Распределение генов белков и разных типов РНК по репликонам
- Распределение генов РНК в геноме
- Исследование межгенных промежутков E. mundtii
- Распределение генов на прямой и обратной цепях
- Сопроводительные материалы
- Литература и дополнительные источники
Введение
Таксономия
Царство(regnum) – Bacteria
Отдел(phylum) – Baciliota
Класс(class) – Bacilli
Порядок(order) – Lactobacillales
Семейство(family) – Enterococcaceae
Род(genus) – Enterococcus
Вид(species) – E. mundtii
Enterococcus mundtii – жёлто-пигментный микроорганизм, иногда встречающийся при инфекциях человека. Принадлежит к роду Enterococcus, все представители которого являются грамположительными, неспорообразующими, каталаза-отрицательными факультативными анаэробами.
E. mundtii обладает низкой устойчивостью к антибиотикам, в отличие от некоторых других видов рода Enterococcus; у E. mundtii не было выявлено генов, связанных с островом патогенности и профагами.
Потенциально может быть использован для ферментации молока в качестве дополнительной культуры (филогенетически Enterococcus связаны с группой молочнокислых бактерий), однако ни одной авторитетной лабораторией не были проведены достаточные исследования этой потенциальной сферы использования.
Материалы и методы
Исходные данные (информация о протеоме и нуклеотидной последовательности) были взяты из библиотеки данных NCBI.
Вычислительные исследования на основе этих данных производились с помощью ресурса Google Sheets. С помощью него была составлена гистограмма длин белков.
На основании данных из геномной таблицы были выявлены расположенные на “+”-цепи ДНК кольцевой хромосомы E. mundtii белок-кодирующие последовательности, найдены интервалы между ними и на основании длин этих интервалов выявлено перекрывание этих CDS. По этим данным построена гистограмма.
С помощью Google Sheets также была составлена диаграмма распределения генов белков и разных типов РНК по репликонам, оставлен график соотношения типов РНК в геноме, определено количество генов на прямой и обратной цепях, выявлено соотношение.
Результаты
3.1 Гистограмма длин белков E. mundtii
На основании данных о белках, представленных в геномной таблице E. mundtii, была построена гистограмма длин белков, отражающие особенности распределения белков из протеома по длине (числу аминокислотных остатков).
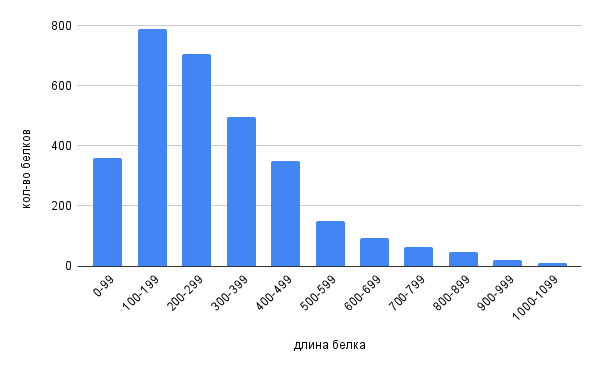
Рис. 1. Гистограмма длин белков на промежутках от 0 до 1099 аминокислотных остатков.
Наибольшее количество белков приходится на промежуток от 100 до 199, затем от 200 до 299, на третьем месте промежуток от 300 до 399 аминокислотных остатков.
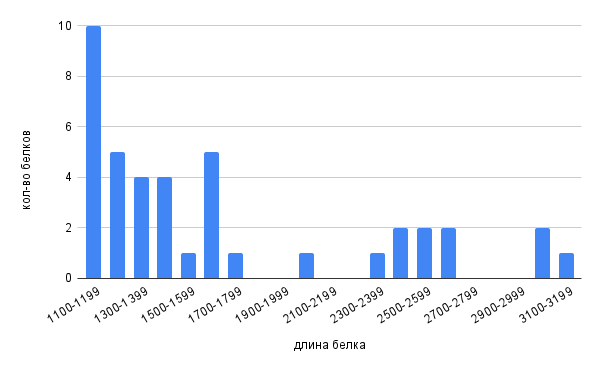
Рис. 2. Гистограмма длин белков на промежутках от 1100 до 3199 аминокислотных остатков.
Можно заметить, что с увеличением длины кол-во белков соответствующей длины уменьшается – наблюдается тенденция на спад. Тем не менее, четкой закономерности нет – части промежутков не соответствует ни один белок, таким как, например, 1900-1999 или 2100-2199.
Таблица 1. Белки с наибольшей длиной.
| name | product_length |
|---|---|
| QWxxN domain | 3091 |
| QWxxN domain | 3073 |
| QWxxN domain | 2641 |
| putative mucin/carbohydrate-binding domain-containing protein | 2586 |
| QWxxN domain | 3165 |
| isopeptide-forming domain-containing fimbrial protein | 2552 |
| QWxxN domain | 2632 |
3.2 Распределение генов белков и разных типов РНК по репликонам
Таблица 2. Распределение по репликонам.
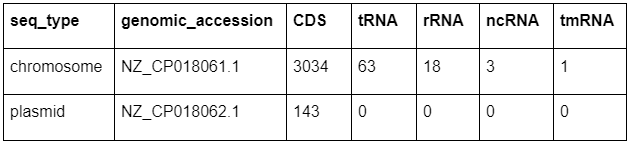
В столбце seq_type, где указан тип последовательности, встречается два значения: chromosome (хромосомная) и plasmid (плазмидная). Для каждого из этих значений было подсчитано кол-во встречающихся участков кодирующей области(CDS), а также кол-во встречающихся видов РНК.
Можно заметить, что плазмидного типа последовательности значение по каждому из видов РНК равно нулю. Отсутствие РНК в плазмиде может указывать на то, что плазмида не содержит генетической информации или что процесс изоляции плазмиды был неполным или неудачным, что менее вероятно, на мой взгляд, чем первый вариант.
3.3 Распределение генов РНК в геноме
В протеоме бактерии есть 85 генов, кодирующих РНК:
1.транспортная – 63 гена;
2.рибосомная – 18 генов;
3.некодирующая – 3 гена;
4.транспортно-матричная – 1 ген.
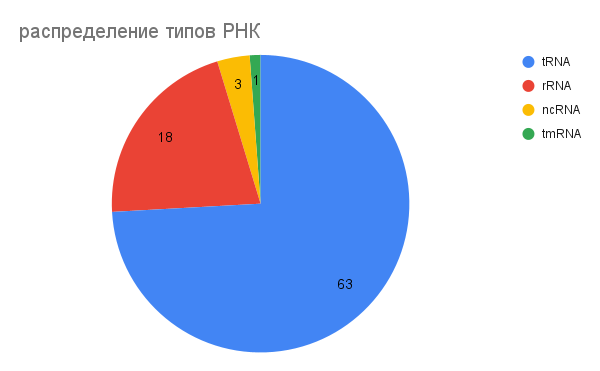
Рис. 3. Распределение типов РНК в хромосоме.
3.4 Исследование межгенных промежутков E. mundtii
На основании данных, приведенных в таблице генома бактерии, были построены гистограммы длин промежутков между соседними неперекрывающимися участками кодирующей области(CDS) на прямой (“+”) цепи.
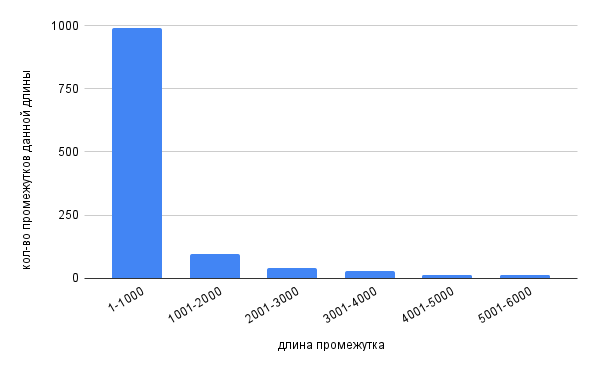
Рис. 4. Гистограмма длин расстояний до 6000 пар нуклеотидов между генами. Наибольшее кол-во промежутков соответствующей длины приходится на промежуток от 1 до 1000 пар нуклеотидов. Это число соответственно равно 991. Таким образом, абсолютное большинство приходится именно на этот промежуток. Далее наблюдается тенденция на спад.
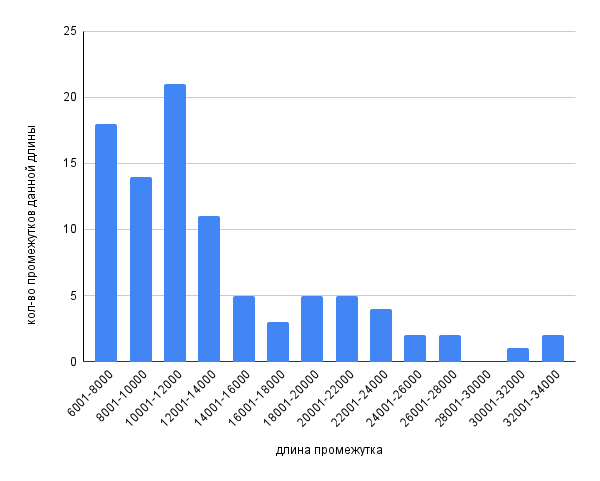
Рис. 5. Гистограмма длин расстояний от 6001 до 34000 пар нуклеотидов между генами.
На данных промежутках значение кол-ва подобных расстояний не превышает 25. Не было найдено ни одного промежутка длиной от 28001 до 30000 пар нуклеотидов.
Свыше 34000 было найдено два промежутка:
1. Белок-кодирующий CDS, расстояние которого до следующего гена равно 61798 парам нуклеотидов;
2. Белок-кодирующий CDS, расстояние которого до следующего гена равно 42842 парам нуклеотидов.
3.5 Распределение генов на прямой и обратной цепях
Были собраны данные о количестве генов на прямой и обратной цепях.
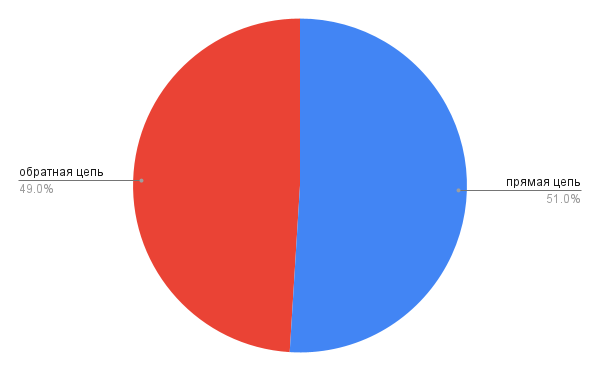
Рис. 6. Распределение генов на прямой и обратной цепи. Как можно наблюдать, разница в соотношение генов незначительная. На прямую (“+”) цепь приходится 51% всех генов E. mundtii, а на обратную (“-”) – 49%.
Сопроводительные материалы
1. Таблица генома из библиотеки NCBI.
2. Нуклеотидная последовательность бактерии в формате fasta.
3. Таблица Google Sheets.
Литература и дополнительные источники
1. Collins M, Farrow J, Jones D. 1986. Enterococcus mundtii sp. nov. Int. J. Syst. Bact.
2. Enterococcus mundtii sp. nov. Matthew D. Collins, John A. E. Farrow, Dorothy Jones.
3. The ecology, epidemiology and virulence of Enterococcus. Katie Fisher, Carol Phillips.
4. Endophthalmitis Caused by Enterococcus mundtii Tomomi Higashide, Mami Takahashi, Akira Kobayashi, Shinji Ohkubo, Mayumi Sakurai, Yutaka Shirao, Toshihiro Tamura and Kazuhisa Sugiyama.
5. Isolation of Enterococcus mundtii from normally sterile body sites in two patients. A. Kaufhold and P. Ferrieri.
6. Genomic comparative analysis of the environmental Enterococcus mundtii against enterococcal representative species. Guillermo D Repizo, Martín Espariz, Víctor S Blancato, Cristian A Suárez, Luis Esteban, Christian Magni.