Практикум 11
Гомология и множественное выравнивание
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Выбор семейства доменов из Pfam для анализа
На первом этапе использовался список всех семейств из Pfam (©Алексеевский АВ), на нем был применен фильтр и помимо прочих найдено следующее семейство: альфа-домен рибонуклеотидредуктазы. На втором этапе поиска была посещена страница в Pfam с описанием выбранного семейства доменов.
2. Краткое описание семейства
При выполнении задания обнаружилось, что нашла я сначала страничку семейства в InterPro, решено было найти ее в Pfam-legacy (хотя Pfam website в процессе отключения и переправки своих данных в InterPro). Здесь приводится описание семейства, в основном взятое из Pfam-legacy, с возможными дополнениями из InterPro.
Формальные данные:
- AC семейства (PF-номер): PF17975 (IPR040763)
- ID: RNR_Alpha (RNR_alpha_hel)
- Название: Ribonucleotide reductase alpha domain
- Число последовательностей в seed: 33
- Число последовательностей в full: 393
- Число доменных архитектур: 13*
*по данным InterPro число доменных архитектур - 30.

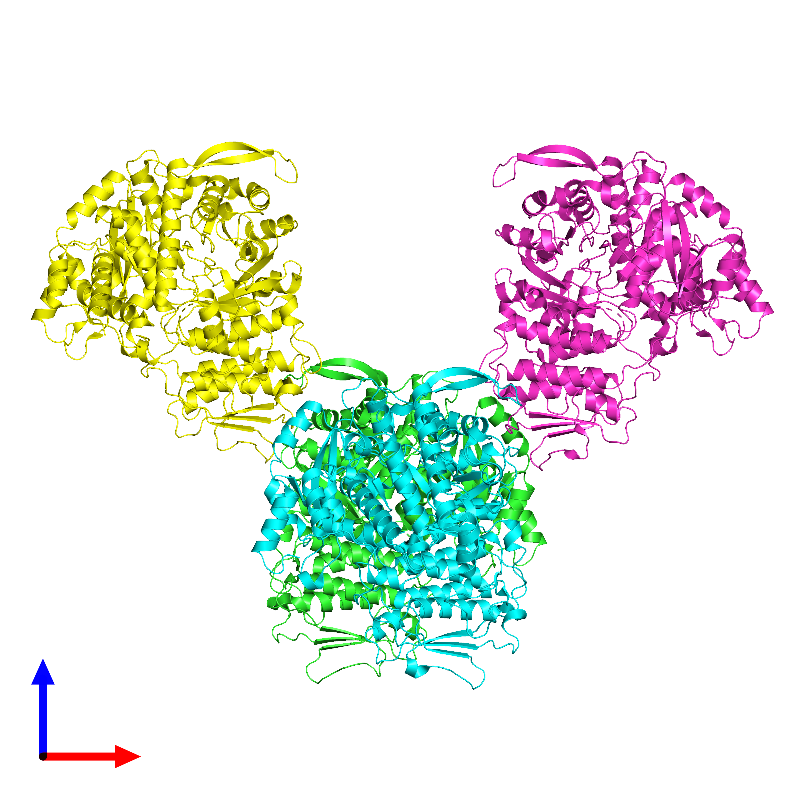
Члены семейства включают рибинуклеотидредуктаз, которые катализируют восстановление дезоксирибонуклеотидов из соответствующих рибонуклеотидов. Этот процесс обеспечивает предшественников для синтеза ДНК. Всего выделяют три класса редуктаз, этот домен можно найти у представителей II класса. RNR этого класса используют кофермент B12 (AdoCbl). Кристаллическая структура состоит из четырех цепей: A,B,C,D (рис 2, рис 3). В состав RNR_Alpha в основном входят фрагменты последовательностей белков бактерий и вирусов.
3. Описание выравнивания seed с точки зрения гомологичности всех последовательностей или их подмножества
//при нажатии на рисунки к этому заданию можно посмотреть на их увеличенный формат
1 этап.
Выраванивание seed со страницы семейства в Pfam в fasta-формате.
2 этап.
Создан файл Jalview-проект с выравниванием
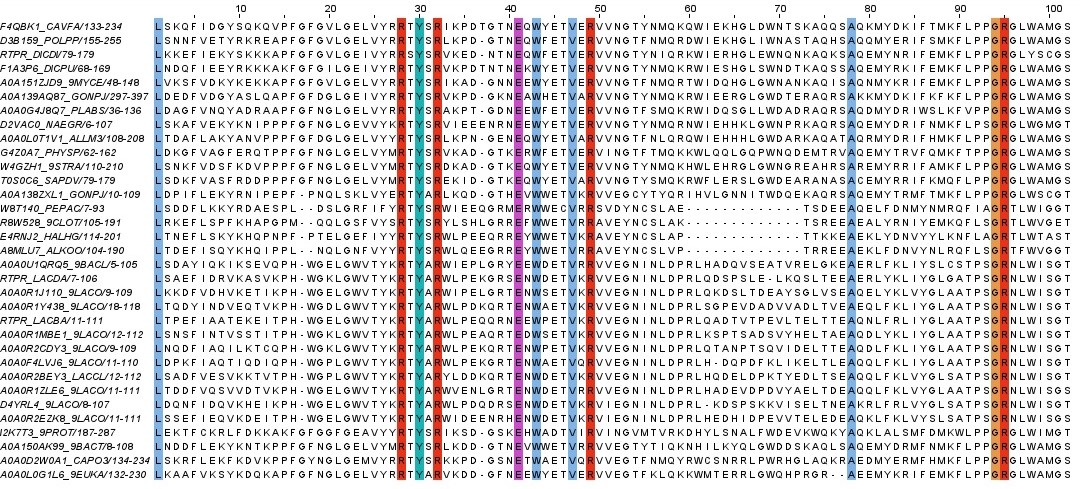
всех последовательностей seed, окрашенное по проценту идентичности. Также проведено окрашивание Clustal
при разных порогах идентичности, результаты представлены на рисунках 4-6.
3 этап.
Окрашивание Clustal было сделано по порогу консервативности 100, таким образом были получены границы потенциальных
достоверных блоков. Интересно, что все окрашенные столбцы, кроме последнего, проходят по порогу идентичности
в 100%. Затем было выполенено условие по гэпам -
на рисунке 7 можно увидеть блоки, которые остались после исключения групп с гэпами. Таким образом имеется три
максимальных достоверных блока, включающих все последовательности,
на колонках: 28-32, 41-49, 78-98*. Также можно назвать достоверным
блоком 1 колонку, состоящую из лейцинов.
*95, если считать за границы колонки, проходящие по порогу идентичности в 100%
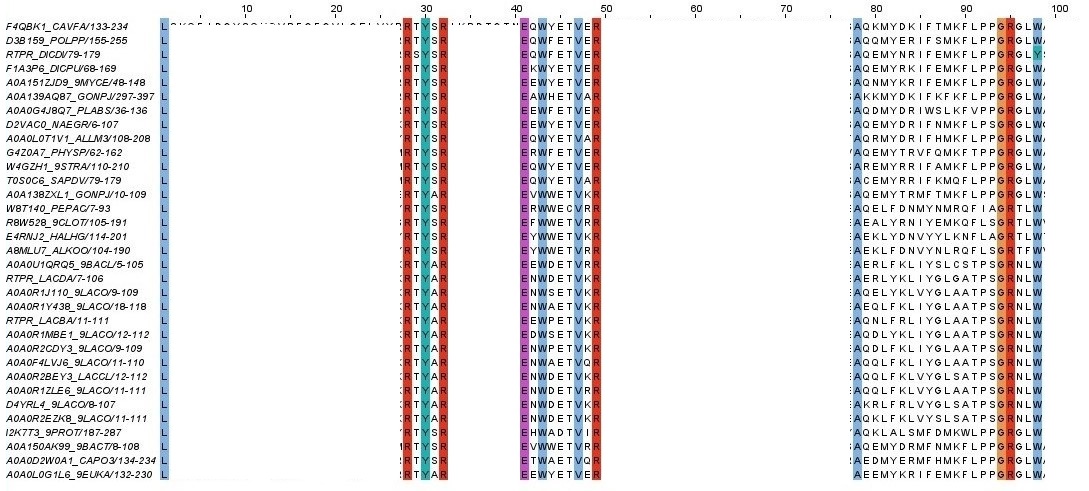
4 этап.
Было выбрано взять подмножество последовательностей, состоящее из всех исходных кроме четырех с инделем
в колонках 60-72. Оно было выделено в отдельное окно, окрашено Clustal с наивысшим порогом консервативности.
Пустых колонок не было, поэтому и удалять их не пришлось. Jalview-project
с итоговым результатом. На рисунке 8 представлены блоки, оставшиеся после исключения групп с гэпами.
Казалось бы получилось как и в 3 этапе три максимальных достоверных блока в колонках: 26-32, 41-53, 78-101.
Но блок для колон 26-32 останется достоверными даже если добавить три из убранных последовательностей, как и блок
для колонок 78-101 может принять еще две последовательности, следовательно, они не являются максимальными.
Таким образом единственный максимальный достоверный блок расположен в колонках 41-53.
Получилось, что достоверные блоки всего множетсва остаются достоверными подблоками подмножества последовательностей,
но не все максимальные достоверные блоки остаются такими при переходе от всей группы seed к его части.
5/6 этап.
Участки без достоверных подблоков следует искать там, где нет достоверных блоков как для всего множества seed, так
и для рассматривавшегося на 4 этапе подмножества. Получается первичная оценка подходящих колонок:
2-25, 33-40, 54-77, 102. Из общего для seed выравнивания выделим колонки 54-77 в отдельное окно в Jalview.
При быстрой проверке не удалось найти участка выравнивания seed без достоверных подблоков, содержащих хотя бы 3 колонки.
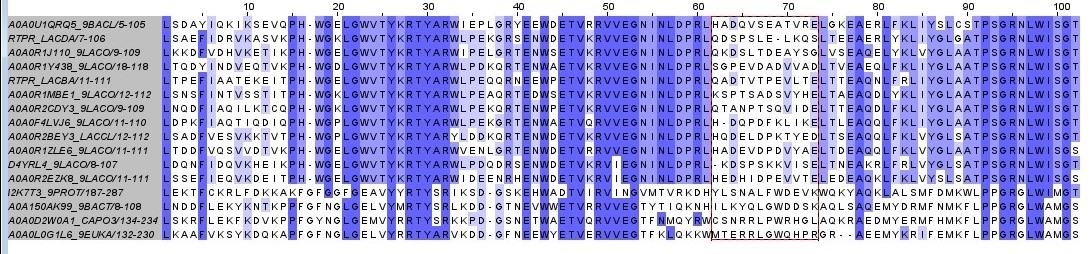
Поэтому решено было найти блок, не содержащий все последовательности, в котором маловероятно, что выравнивание
отражает ход эволюции. Найденный блок состоял из 16 последовательностей с 62 по 73 позиции (рисунок 9).
Проект Jalview
с указанным блоком показывает, что колонок с консервативностью выше 30% в ней нет и
из-за достаточно хаотичного расположения аминокислотных остатков можно предположить, что выравнивание в этом блоке
не является эволюционным.
7 этап.
В итоге, выравнивание отражает гомологию последовательностей достаточно достоверно. На большой части последовательностей
обнаруживаются достоверные блоки, а небольшие участки с достоверными блоками малых размеров могут быть
связаны как с ошибками множественного выравнивания, так и с тем, что соответствующий участок не выполняет важную функцию и
на нем накапливаются локальные мутации.



4. Домены, входящие в состав белков с разной доменной архитектурой, достоверно различаются?
Доменные архитектуры были выбраны из предложенных в Pfam Legacy.
Первая доменная архитектура:
RNR_Alpha (PF17975). Состоит только из представителей выбранного семейства доменов.
Вторая доменная архитектура:
ATP-cone (PF03477), RNR_Alpha (PF17975), Ribonuc_red_lgC (PF02867).
Присутствуют участки, принадлежащие другим доменам.
Запроса белков с нужным доменом в UniProt: (xref:pfam-PF17975).
Таблица в формате excel
с информацией об AC, ID, длине последовательности и доменной архитектуре белков. Листы DArch1 и DArch2 посвящены спискам белков с соответствующими
доменными архитектурами.
Список источников
- Lundin, D.; Berggren, G.; Logan, D.T.; Sjöberg, B.-M. The Origin and Evolution of Ribonucleotide Reduction. Life 2015, 5, 604-636. https://doi.org/10.3390/life5010604
- Peter Reichard ,From RNA to DNA, Why So Many Ribonucleotide Reductases?.Science260,1773-1777(1993).DOI:10.1126/science.8511586
//а также все гиперссылки, присутствующие в смысловой части практикума