Практикум 3
Сравнительный анализ канонической ДНК и стеблей тРНК
Автор старался, но не может гарантировать отсутствие биологических ошибок.
Задание 1
С помощью инструментов пакета 3DNA были построены модели структур A-,B- и Z-формы ДНК: gatc-a.pdb, gatc-b.pdb, gatc-c.pdb. Дуплексы ДНК представляют собой 5 раз повторенную последовательность "gatc".
Задание 2
Было решено сравнить сгенерированную структуру A-форма дуплекса ДНК со структурой той же формы, полученной экспериментальными данным: 3v9d (рисунок 1). Обе структуры, как и ожидалось, похожи. Правда, в экспериментальной структуре включены молекулы воды из окружения, которые отсуствуют в сгенерированном pdb-файле.
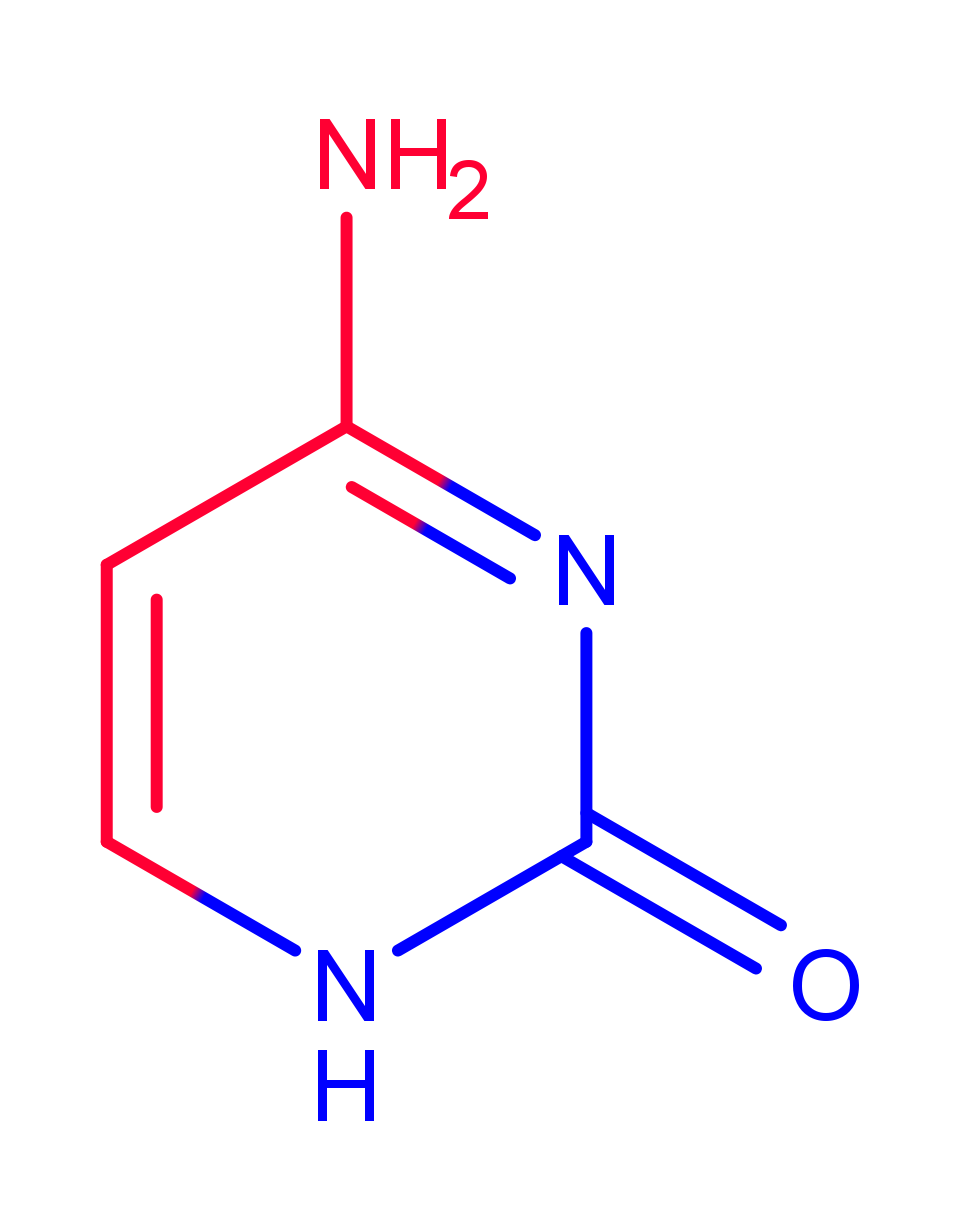
Далее было произведено определение направления обращения атомов азотистого основания - цитозина (рисунок 2). Рассматривалась экспериментальная структура 3v9d. Резюме:
- В сторону большой бороздки обращены атомы с10.c4, c10.n4, c10.c5, c10.c6;
- В сторону малой бороздки обращены атомы c10.n1, c10.c2, c10.o2, c10.n3.
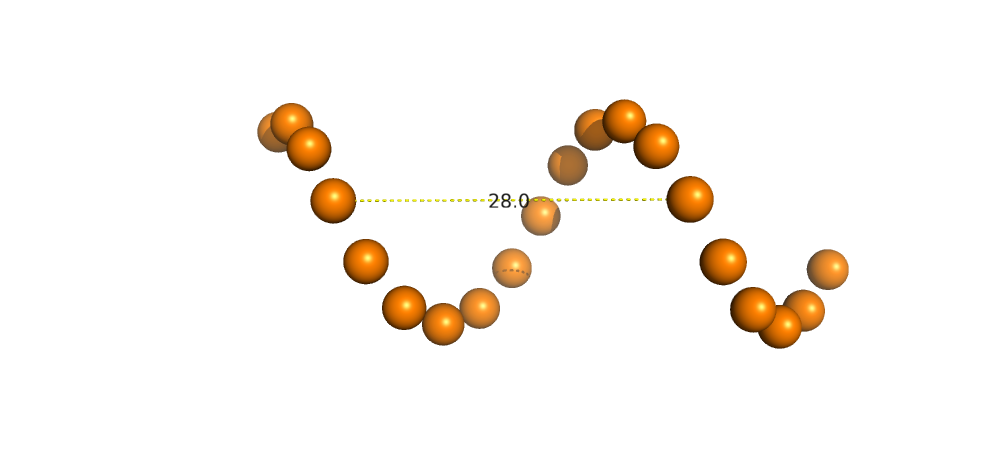
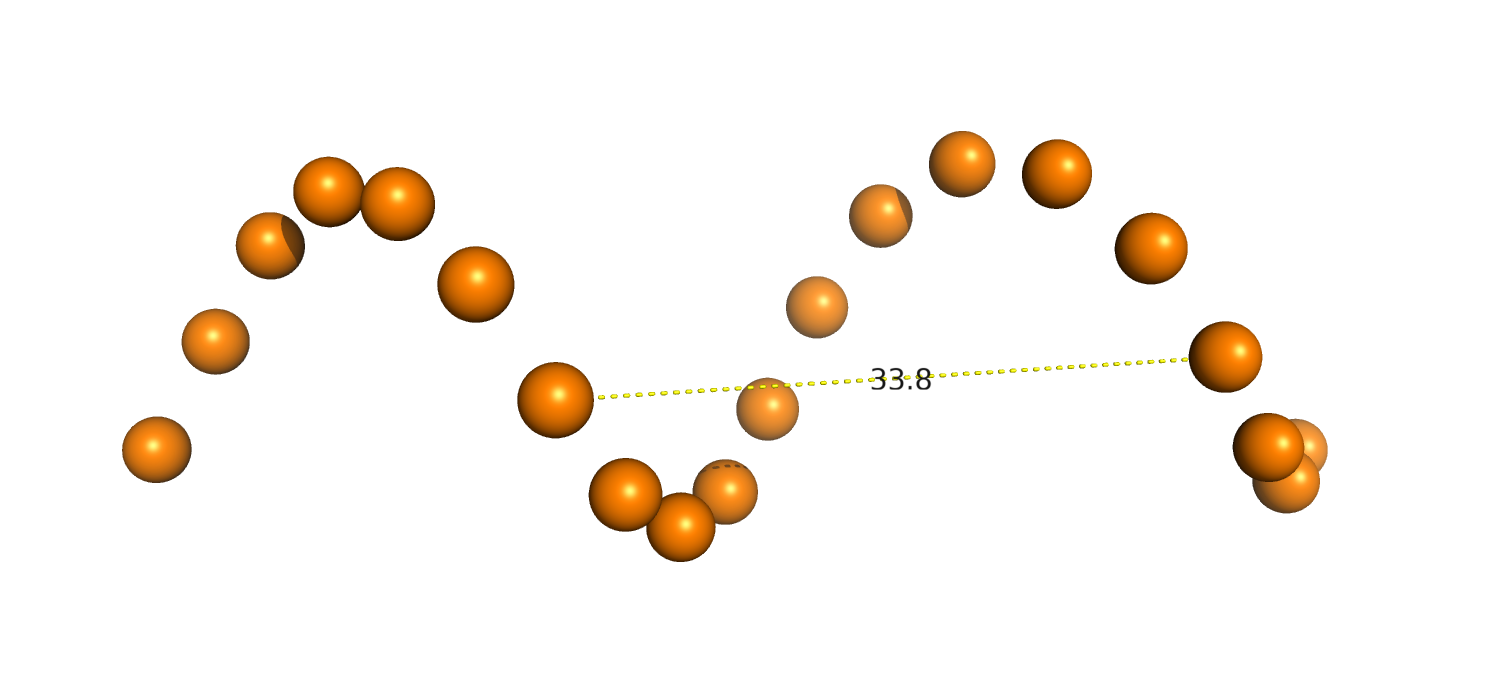
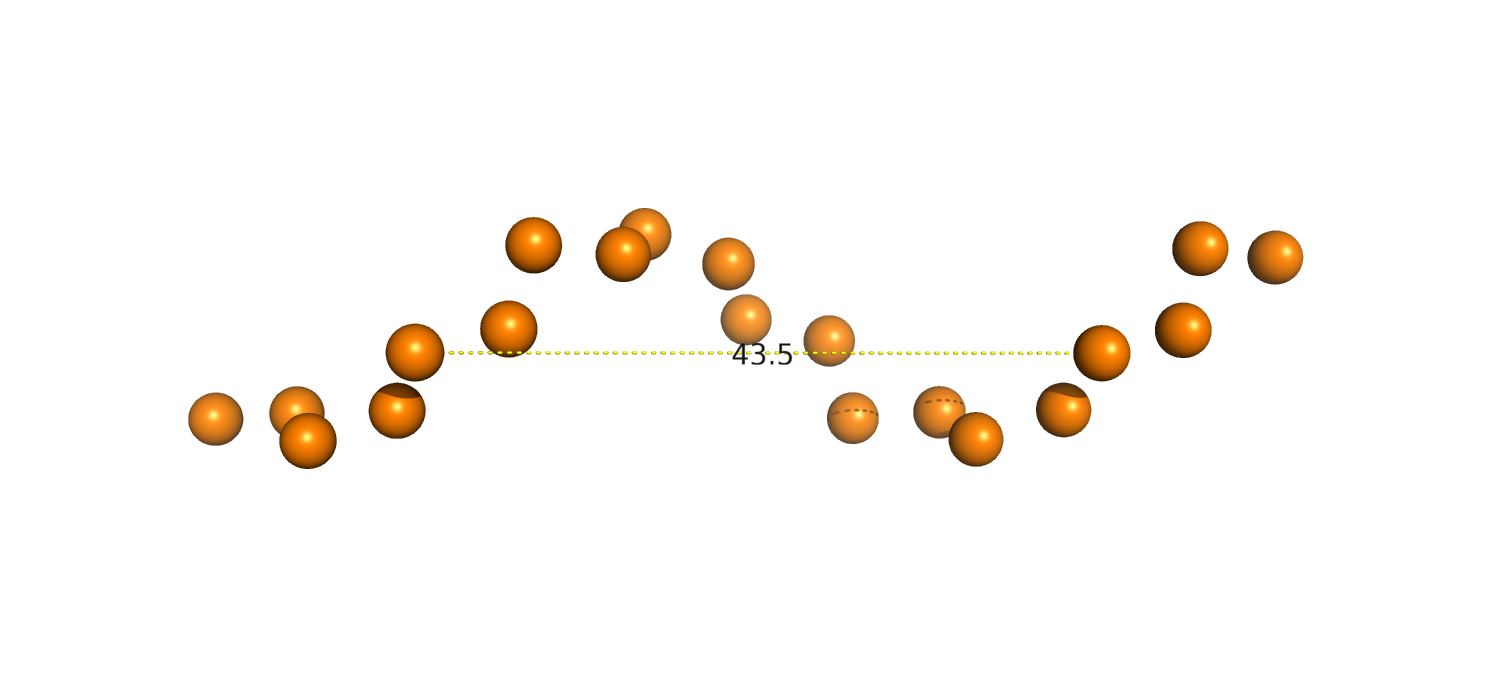
| Форма ДНК | Тип спирали | Шаг спирали (Å) | Число оснований на виток |
Ширина большой бороздки | Ширина малой бороздки |
|---|---|---|---|---|---|
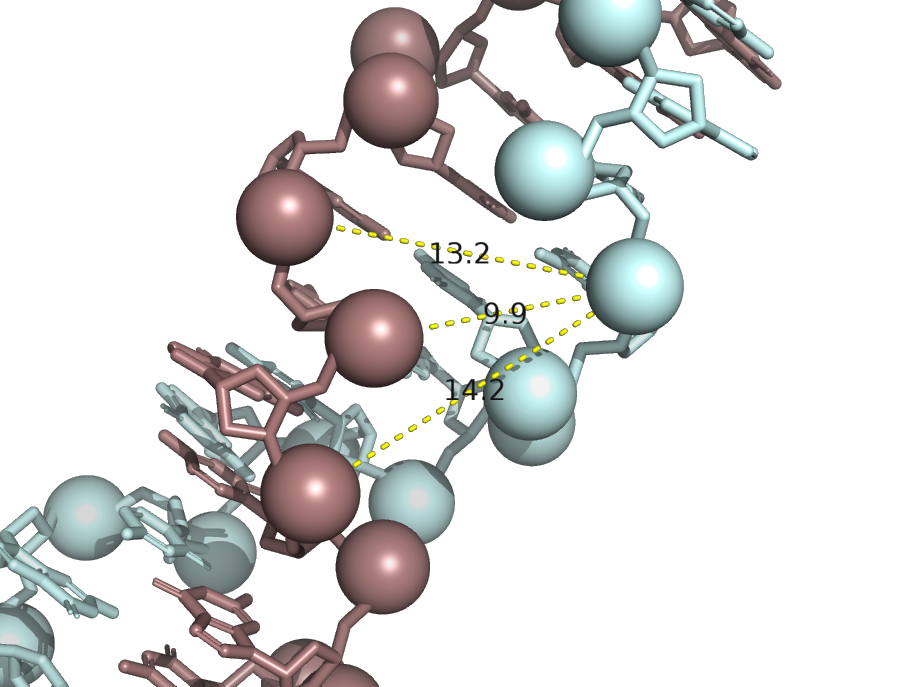
| A-форма | правая | 28.0 | 11 | 16.8 (A/DC`12/P-B/DT`31/P) |
8.0 (A/DC`12/P-B/DT`23/P) |
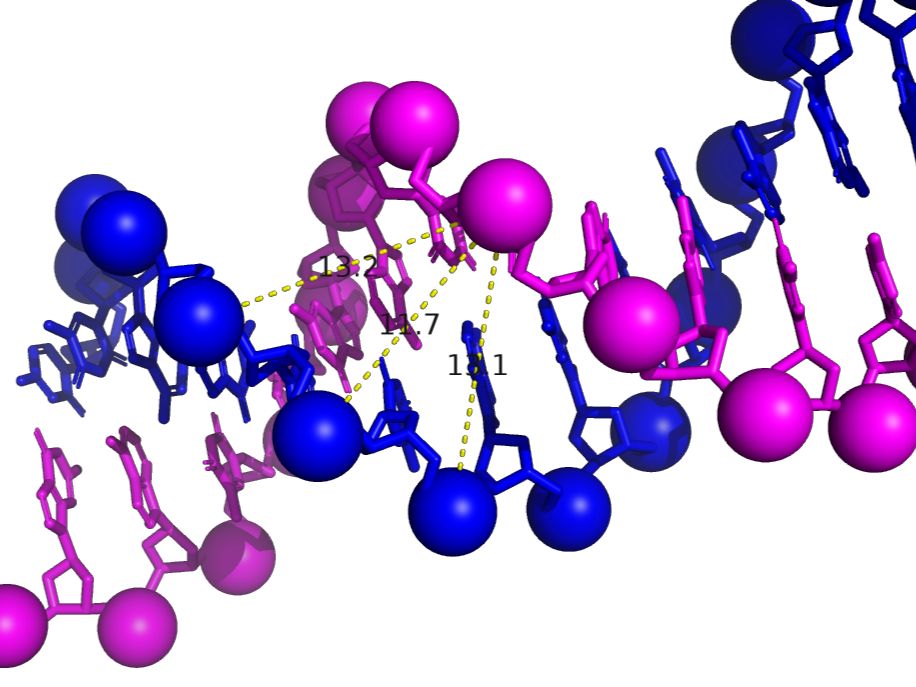
| B-форма | правая | 33.8 | 10 | 17.8 (A/DG`9/P-B/DG`29/P) |
11.7 (A/DG`9/P-B/DC`36/P) |
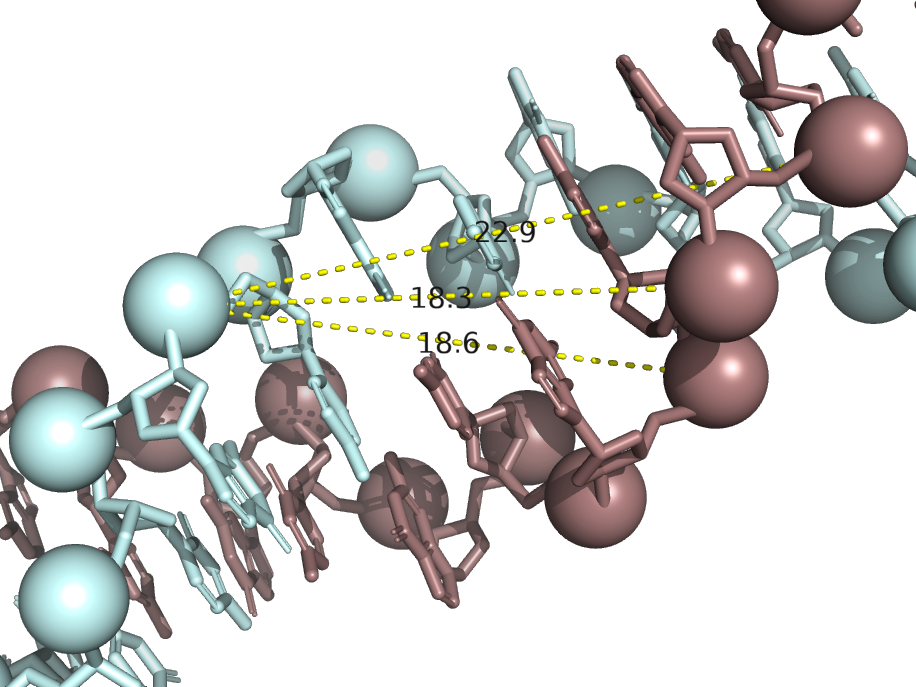
| Z-форма | левая | 43.5 | 12 | 18.3 (A/DC`10/P-B/DC`28/P) |
9.9 (A/DC`10/P-B/DG`35/P) |
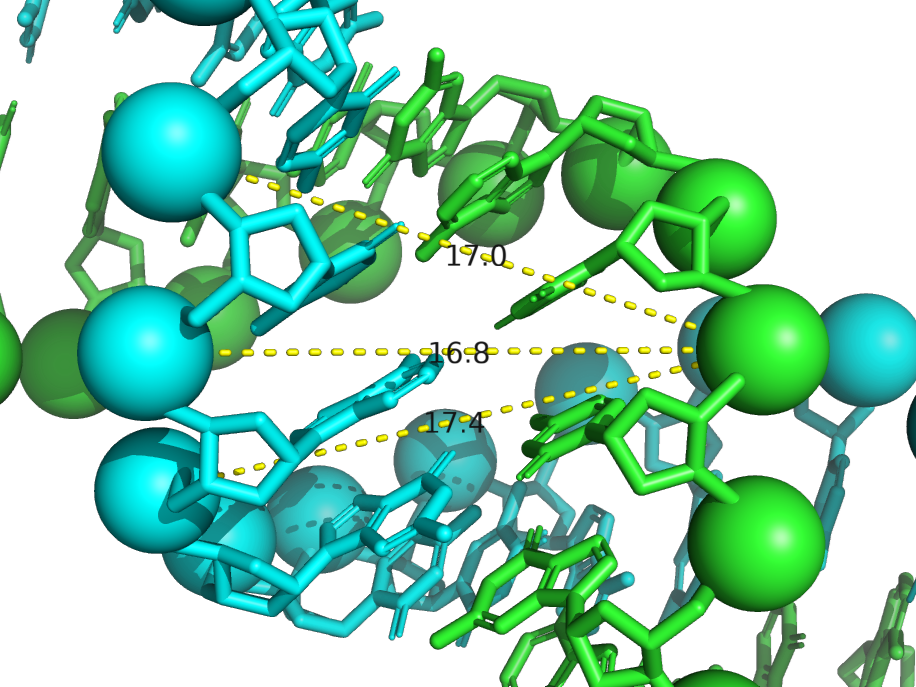
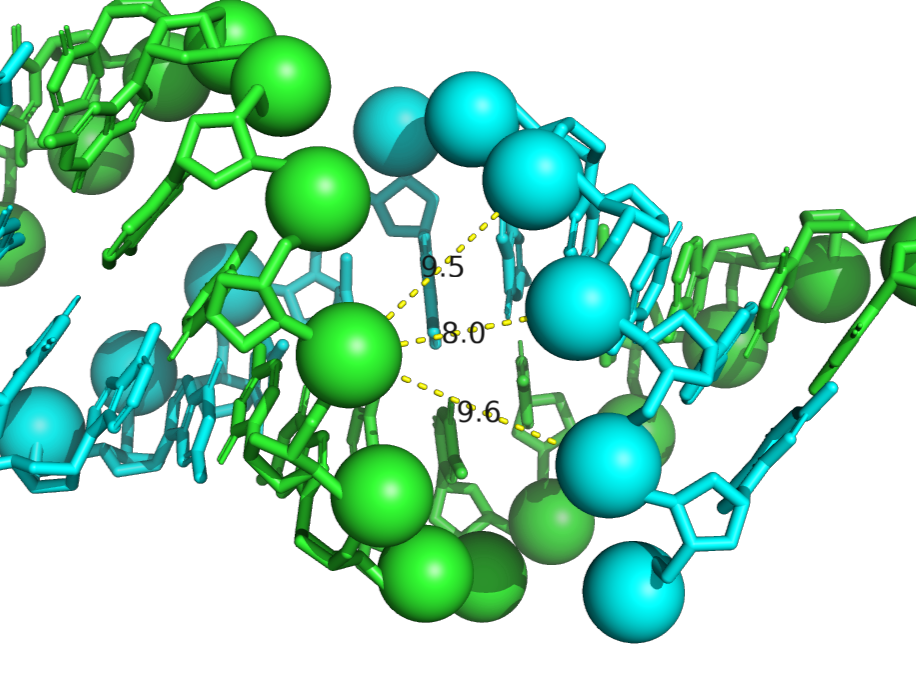
Задание 3
Ссылка на файл: 1FFY. Ссылка на аналогичные файлы для сгенерированных структур ДНК: A-форма, B-форма, Z-форма. Расчеты проводились при помощи программы на Python.
Торсионные углы
| Тип НК | α | β | γ | δ | ε | ζ | χ |
|---|---|---|---|---|---|---|---|
| тРНК | -30.2 | 35.08 | 39.83 | 85.46 | -131.99 | -52.33 | -123.71 |
| A-форма | -51.7 | 174.8 | 41.7 | 79.08 | -147.79 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.64 | 31.14 | 143.34 | -140.8 | -160.5 | -97.99 |
| Z-форма | -54.42 | 21.12 | -61.47 | 116.25 | -100.44 | 0.43 | -47.8 |
Значения торсионных углов были найдены в соответствующих файлах и их средние значения определены при помощи программы на Python. Результаты представлены в таблице 2. В той же программе было рассчитано, что тяжи в тРНК больше всего похожи на A-форму ДНК.
Водородные связи
wget "https://files.rcsb.org/download/1FFY.pdb" -O "1FFY.pdb"
remediator --old 1FFY.pdb > 1FFY_old.pdb
find_pair -t 1FFY_old.pdb stdout | analyze
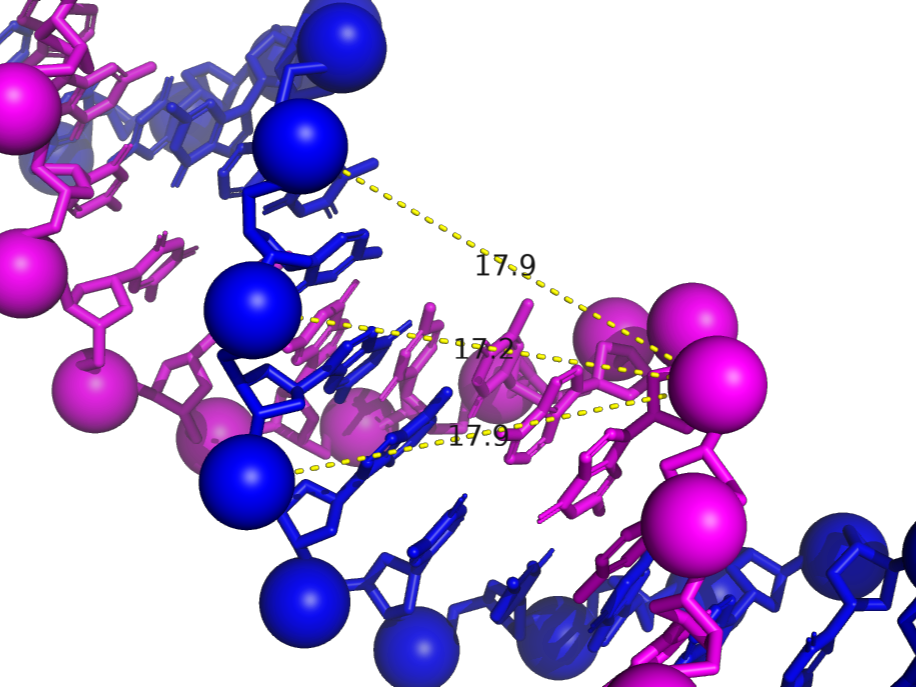
Структура водородных связей между основаниями определялась по представленным слева данным из файла 1FFY_old.out. Нуклеотиды образующие стебли (stems) во вторичной структуре заданной тРНК: 1-7 и 72-66, 49-53 и 65-61, 38-44 и 32-26, 10-13 и 25-22. Неканонические пары оснований в структуре тРНК получены по данным справа: 5U-68G, 49G-65U, 50G-64C, 54U-58A, 55U-18G, 36U-33U, 38A-32C,44A-26G, 14A-8U, 15G-48C, 16G-17U. Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру: 54U-58A, 55U-18G, 36U-33U, 14A-8U, 15G-48C, 16G-17U, 19G-56C.
Strand I Strand II Helix
1 (0.003) ....>T:...1_:[..G]G-----C[..C]:..72_:T<.... (0.003) |
2 (0.008) ....>T:...2_:[..G]G-----C[..C]:..71_:T<.... (0.005) |
3 (0.007) ....>T:...3_:[..G]G-----C[..C]:..70_:T<.... (0.004) |
4 (0.009) ....>T:...4_:[..C]C-----G[..G]:..69_:T<.... (0.007) |
5 (0.007) ....>T:...5_:[..U]U-*---G[..G]:..68_:T<.... (0.009) |
6 (0.006) ....>T:...6_:[..U]U-----A[..A]:..67_:T<.... (0.004) |
7 (0.008) ....>T:...7_:[..G]G-----C[..C]:..66_:T<.... (0.002) |
8 (0.006) ....>T:..49_:[..G]G-*---U[..U]:..65_:T<.... (0.005) |
9 (0.005) ....>T:..50_:[..G]G-----C[..C]:..64_:T<.... (0.002) |
10 (0.003) ....>T:..51_:[..U]U-----A[..A]:..63_:T<.... (0.001) |
11 (0.004) ....>T:..52_:[..G]G-----C[..C]:..62_:T<.... (0.004) |
12 (0.007) ....>T:..53_:[..G]G-----C[..C]:..61_:T<.... (0.002) |
13 (0.005) ....>T:..54_:[..U]U-**--A[..A]:..58_:T<.... (0.007) |
14 (0.008) ....>T:..55_:[..U]U-**+-G[..G]:..18_:T<.... (0.011) x
15 (0.011) ....>T:..36_:[..U]U-**+-U[..U]:..33_:T<.... (0.005) |
16 (0.013) ....>T:..38_:[..A]A-**--C[..C]:..32_:T<.... (0.003) |
17 (0.012) ....>T:..39_:[..G]G-----C[..C]:..31_:T<.... (0.007) |
18 (0.004) ....>T:..40_:[..G]G-----C[..C]:..30_:T<.... (0.004) |
19 (0.004) ....>T:..41_:[..G]G-----C[..C]:..29_:T<.... (0.009) |
20 (0.007) ....>T:..42_:[..U]U-----A[..A]:..28_:T<.... (0.008) |
21 (0.007) ....>T:..43_:[..G]G-----C[..C]:..27_:T<.... (0.005) |
22 (0.009) ....>T:..44_:[..A]A-**--G[..G]:..26_:T<.... (0.011) |
23 (0.005) ....>T:..10_:[..G]G-----C[..C]:..25_:T<.... (0.005) |
24 (0.005) ....>T:..11_:[..C]C-----G[..G]:..24_:T<.... (0.010) |
25 (0.004) ....>T:..12_:[..U]U-----A[..A]:..23_:T<.... (0.008) |
26 (0.006) ....>T:..13_:[..C]C-----G[..G]:..22_:T<.... (0.004) |
27 (0.003) ....>T:..14_:[..A]A-**--U[..U]:...8_:T<.... (0.007) |
28 (0.006) ....>T:..15_:[..G]G-**+-C[..C]:..48_:T<.... (0.008) x
29 (0.005) ....>T:..16_:[..G]G-**--U[..U]:..17_:T<.... (0.003) +
30 (0.008) ....>T:..19_:[..G]G-----C[..C]:..56_:T<.... (0.004) +
Detailed H-bond information: atom-name pair and length [ O N]
1 G-----C [3] O6 - N4 2.96 N1 - N3 2.96 N2 - O2 2.86
2 G-----C [3] O6 - N4 3.14 N1 - N3 2.96 N2 - O2 2.66
3 G-----C [3] O6 - N4 2.98 N1 - N3 2.95 N2 - O2 2.82
4 C-----G [3] O2 - N2 2.70 N3 - N1 2.99 N4 - O6 3.14
5 U-*---G [2] O2 - N1 2.74 N3 - O6 2.91
6 U-----A [2] N3 - N1 2.94 O4 - N6 3.14
7 G-----C [3] O6 - N4 2.56 N1 - N3 2.76 N2 - O2 2.88
8 G-*---U [2] O6 - N3 2.94 N1 - O2 2.69
9 G-----C [2] N1 - N3 3.41 N2 - O2 3.01
10 U-----A [2] N3 - N1 2.71 O4 - N6 2.80
11 G-----C [3] O6 - N4 2.83 N1 - N3 2.81 N2 - O2 2.70
12 G-----C [3] O6 - N4 2.84 N1 - N3 2.83 N2 - O2 2.67
13 U-**--A [2] O2 - N6 2.52 N3 - N7 3.10
14 U-**+-G [1] O2 - N1 2.91
15 U-**+-U [2] N3 - O2 3.03 O4 * O4' 3.09
16 A-**--C [1] N6 - O2 2.92
17 G-----C [3] O6 - N4 2.87 N1 - N3 2.88 N2 - O2 2.76
18 G-----C [3] O6 - N4 3.26 N1 - N3 3.19 N2 - O2 3.00
19 G-----C [3] O6 - N4 3.20 N1 - N3 3.09 N2 - O2 2.95
20 U-----A [2] N3 - N1 2.74 O4 - N6 2.85
21 G-----C [3] O6 - N4 3.11 N1 - N3 2.92 N2 - O2 2.76
22 A-**--G [2] N6 - O6 2.71 N1 - N1 2.79
23 G-----C [3] O6 - N4 2.80 N1 - N3 2.83 N2 - O2 2.79
24 C-----G [3] O2 - N2 2.83 N3 - N1 2.92 N4 - O6 2.90
25 U-----A [2] N3 - N1 2.97 O4 - N6 2.93
26 C-----G [3] O2 - N2 2.79 N3 - N1 2.98 N4 - O6 3.03
27 A-**--U [2] N7 - N3 3.08 N6 - O2 2.82
28 G-**+-C [2] N1 - O2 2.53 N2 - N3 2.64
29 G-**--U [2] N7 - N3 3.69 O6 * O4 2.66
30 G-----C [3] O6 - N4 2.65 N1 - N3 2.67 N2 - O2 2.57