Практикум 8
Нуклеотидный BLAST
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Разные варианты BLAST для фрагмента ДНК

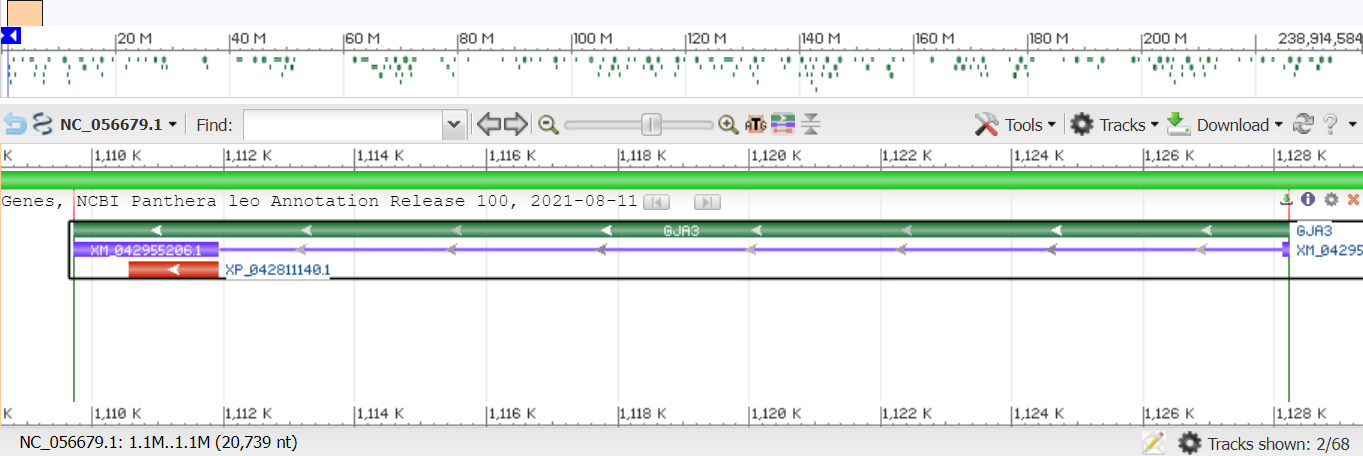
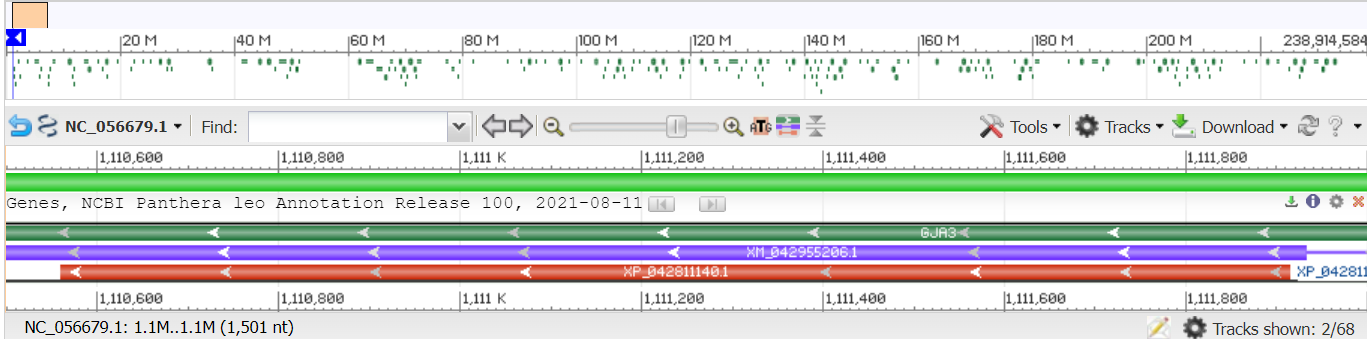
В геноме Panthera leo из 7 практикума был взят участок, внутри которого содержится ген GJA3, кодирующий белок альфа-3 щелевого соединения (gap junction alpha-3 protein). Идентификатор нуклеотидной записи, откуда был взят фрагмент (GeneID): 122229570. Координаты GJA3: complement(1110560..1111915), соотвественно длина - 1355 п.н. Изначально был выбран фрагмент от 1108600 до 1129338 (длина - 20738 п.н.), содержащий не только CDS, но и весь ген с мРНК (ссылка на NCBI Graphics). На рисунке 2 показана схема расположения CDS на выбран фрагменте (показан красным), также видно участок, кодирующий мРНК (фиолетовый) и весь ген (зеленый). Другие белок-кодирующие области в выбранный фрагмент не вошли. Далее при поиске гомологичных последовательностей алгоритмами BLAST было обнаружено, что чрезмерно крупный фрагмент только понижает процент покрытия, но не влияет на список находок. Решено было сократить исследуемый участок и взять фрагмент от 1110500 до 1112000 (длина - 1501 п.н.) - представлен на рисунке 3. FASTA-файлы с последовательностями первоначального и выбранного фрагмента.
В качестве целевого таксона, в котором будет осуществляться поиск BLAST, была выбрана группа Unidentata - клада чешуйчатых рептилий, объединяющая Scinciformata, Laterata, Toxicofera, среди представителей есть змеи, игуаны, поясохвосты и другие. На рисунке 4 можно посмотреть на одного из их представителей - ящерицу, названную в честь фантастического дракона Смауга. Львы относятся к млекопитающим (Mammalia), Unidentata - к пресмыкающимся (Reptilia), оба класса относятся к типу хордовые (Chordata). Далее выполнялся поиск гомологичных последовательностей с использованием различных методов BLAST. Для запусков всех из них использовался в качестве последовательности запроса fasta-файл, поиск вёлся по коллекции данных для организмов из группы Unidentata (taxid:1329950), в остальном сохранялись параметры по умолчанию, если иное не указано.

-
blastn
Program version: BLASTN 2.14.1+.
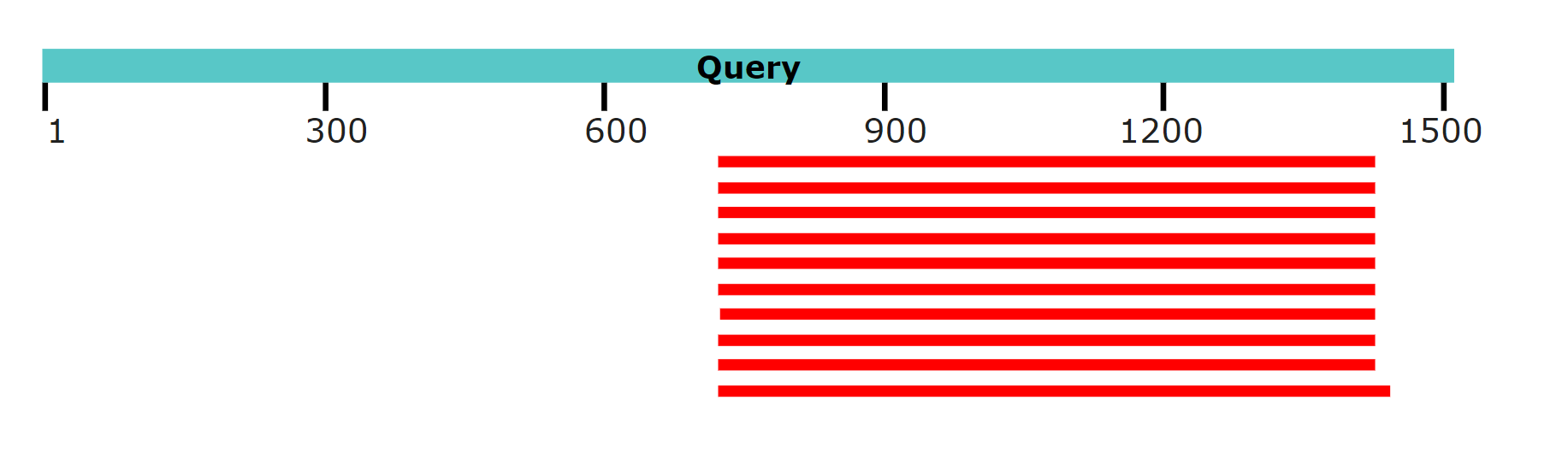
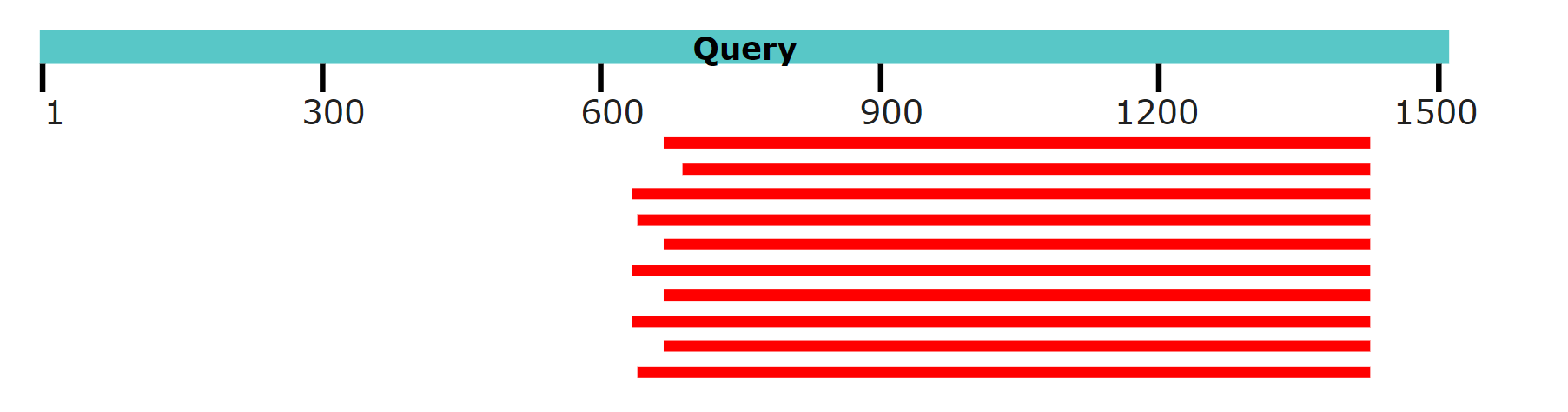
Поиск проводился при трех разных длинах слов: 7, 11, 15. При длине затравки 7: находок 734. При длине затравки 11: находок 659. При длине затравки 15: находок 254. Получается, что число находок падает с увеличением длины затравки, потому что совпадать должны уже более крупные участки. Наиболее схожая последовательность в любом случае принадлежала Sceloporus undulatus (предполагаемо кодирует мРНК альфа-3 щелевого соединения), с e-value в 4e-146. Совпадение результатов в первых строчках по e-value возможно указывает на значительное влияние длины затравки только на выравнивание менее схожих последовательности. Графическое представление результатов на рисунке 5.
blastn - поиск по гену в нуклеотидной базе данных. Алгоритм следует использовать для поиска гомологичных последовательностей среди не близкородственных видов. -
megablast
Program version: BLASTN 2.14.1+.
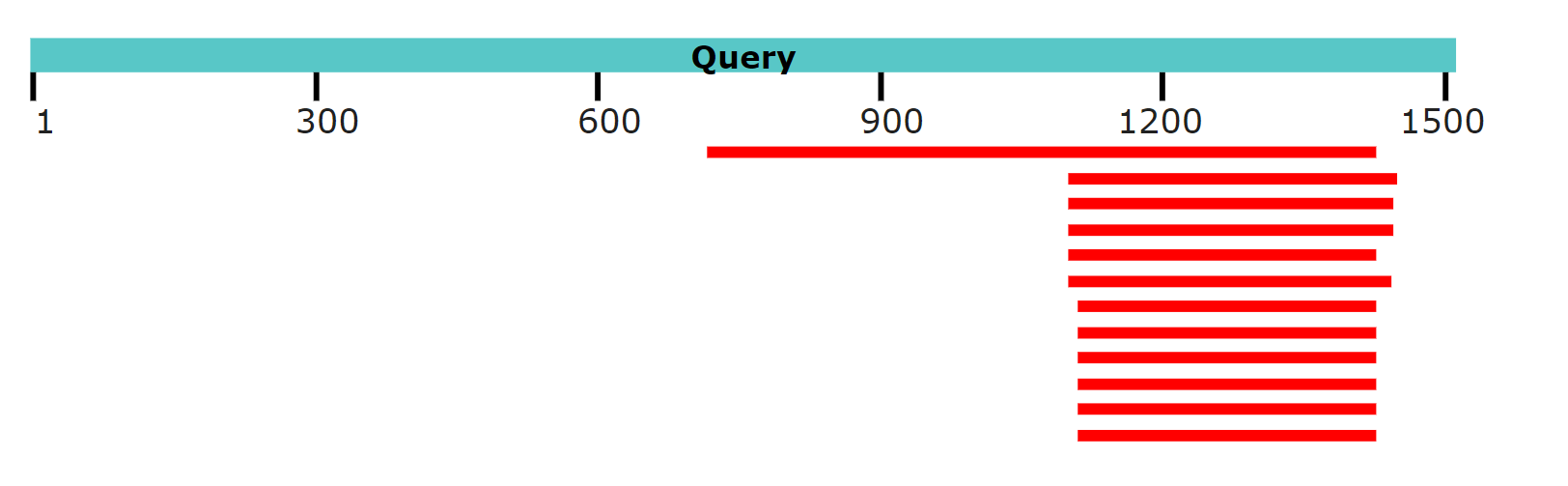
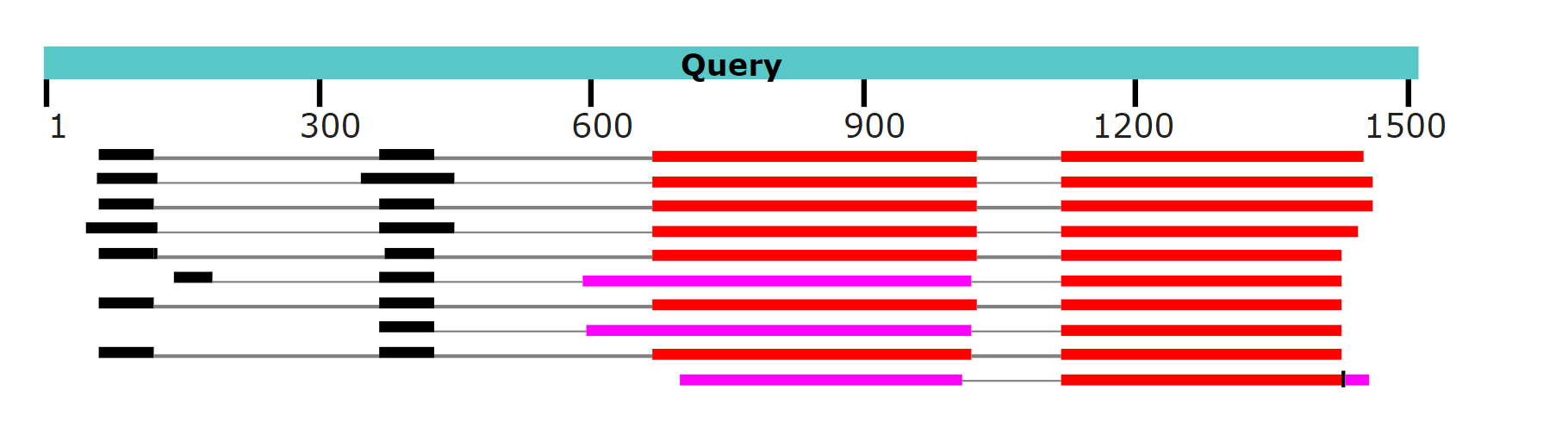
При длине затравки 16: находок 197, наиболее схожая последовательность совпадала с blastn, но e-value вырос до 5e-112. При длине затравки 28: находок 12 и больше всего наблюдалось сходство с участком генома Hemicordylus capensis (тоже предполагаемо кодирует мРНК альфа-3 щелевого соединения), e-value = 8e-110. Графическое представление результатов на рисунке 6.
Участки совпадения получаются намного меньше по длине чем при blastn. megablast - более точный поиск по гену в нуклеотидной базе данных. Программа может находить последовательности с высоким процентом идентичности среди близкородственных видов. -
blastx
Program version: BLASTX 2.14.1+.
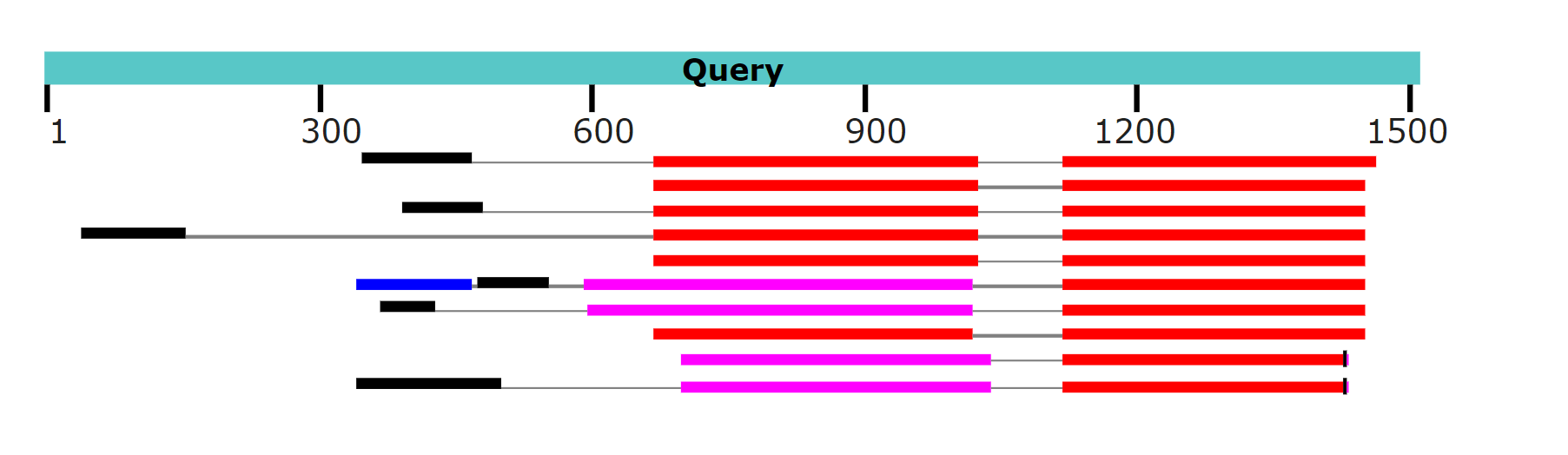
При длине затравки 5: находок 571. При максимальной длине затравки в 6 число находок падает всего на две до 569. Наиболее схожая последовательность принадлежала Anolis carolinensis и уже представляла собой не мРНК, а сам белок альфа-3 щелевого соединения, e-value = 2e-138. Также программа распознала консервативный домен connexin (белки, кодирующие щелевые соединения). Графическое представление результатов на рисунках 7 и 8.Участки выравнивания увеличиваются по сравнению с выравнвианиями относительно геномных последовательностей. Результаты намекают на предпочтительность поиска гомологов по белковым базам данных для кодирующих последовательностей. blastx - поиск по гену в белковой базе данных. Режим находит белки со схожими функциями по отношению к продукту трансляции кодирующей последовательности гена.
-
tblastx
Program version: TBLASTX 2.14.1+.
Поиск велся при длине затравки 3 (по умолчанию), но разным базам данных. При поиске по коллекции нуклеотидов (nucleotide collection, по умолчанию): 813 находок. Общий объем базы для Unidentata: 1,206,757 последовательностей (6,366,500,533 букв). Больше всего наблюдалось сходство с участком генома Lacerta agilis (предполагаемо кодирует мРНК альфа-3 щелевого соединения), e-value = 1e-131. Из интереса был также произвден поиск по репрезентативным геномам RefSeq (refseq_representative_genomes): 199 находок. Общий объем базы для Unidentata: 313,290 последовательностей (27,884,400,230 букв). Больше всего наблюдалось сходство с участком генома Podarcis muralis (последовательность - участок 4 хромосомы), e-value = 2e-125. Графическое представление результатов на рисунках 9 и 10.
В общем, получается, что более качественные с точки зрения выравнивания находки можно обнаружить в коллекции нуклеотидов, которая предлагается по умолчанию. tblastx - поиск по гену в транслированной нуклеотидной базе данных. Алгоритм можно использовать если известен ген ранне изученного белка и хочется проверить присутствуют ли его гомологи в сборке генома, где не предсказаны гены.

2. Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу
После многодневных страданий, связанных с установкой blast+, удалось получить локальный BLAST на своем ноутбуке. Далее была проведена индексация последовательности генома P. leo:
$ makeblastdb -in 'P.leo_genomic.fasta' -dbtype 'nucl'Были скачены последовательности рРНК E. coli из преложенного файла. В файле содержались следующие рРНК:
- 16S рРНК - часть малой субъединицы рибосомы прокариот (аналог 18S для эукариот). 3'-конец участвует в инициации трансляции и связывании с мРНК. Также обеспечивает связывание большой и малой субъединиц рибосомы и стабилизацию кодон-антикодонового взаимодействия в А-сайте.
- 23S рРНК - часть большой субъединицы рибосомы прокариот, образующая пептидил-трансферазный центр (аналог 28S для эукариот). Зачастую антибиотики ингибирующие трансляцию связываются именно с этим сайтом. Считается, что последовательность 23S рРНК более изменчива, чем у других рРНК.
$ blastn -task blastn -query 16S_rRNA.txt -db P.leo_genomic.fasta -out ~/blast16.out -evalue 0.05
$ blastn -task blastn -query 23S_rRNA.txt -db P.leo_genomic.fasta -out ~/blast23.out -evalue 0.05Файлы с результатами BLAST для 16S и 23S рРНК.
Размер затравки был оставлен 11 (по умолчанию для blastn), evalue опущено до 0.05 (как в веб-версии). Остальные параметры запуска касались исключительно расположения и названия файлов. Для анализа результатов показалась удобнее выдача для 16S и 23S в формате таблицы (добавление параметра -outfmt со значением 7):
$ blastn -task blastn -query 16S_rRNA.txt -db P.leo_genomic.fasta -out blast16.out -evalue 0.05 -outfmt 7
$ blastn -task blastn -query 23S_rRNA.txt -db P.leo_genomic.fasta -out blast23.out -evalue 0.05 -outfmt 7Для обоих последовательностей рРНК удалось найти гомологов.
Для 16S рРНК число находок = 7, для всех есть аннотация и все соответствуют участкам 18S рРНК генома P. leo. Идентификаторы гомологичных 18S рРНК (по порядку следования выравниваний): XR_006196888.1, XR_006196890.1, XR_006196891.1, XR_006196892.1, XR_006196893.1, XR_006196895.1, XR_006196894.1. Не смотря на малую длину покрытия в 43 п.н. (при длине последовательности 16S рРНК для E. coli в 1556 п.н.), мне эти находки не кажутся случайными, потому что все гомологичные участки соотвествуют рРНК и участок выравнивания у E. coli был постоянным (1494..1536).
Для 23S рРНК число находок = 31. Из значимых и приятных находок - 28S рРНК (их идентификаторы: XR_006196897.1, XR_006196898.1, XR_006196902.1, XR_006196899.1, XR_006196901.1, XR_006196896.1). Примечательно, что находок для 28S рРНК по четыре на каждую и каждый раз картировался иной фрагмент последовательности E.coli на иной фрагмент генома P. leo, причем порядок следования фрагментов соблюдался. На мой взгляд, это подтверждает, гомологию эукариотических рРНК с данной прокариотической. Из интересного: для XR_006196899.1 найденные гомологичные последовательности были перевернуты во всех четырех случаях (возможно у львов произошла инверсия для данной рРНК). Остальные 7 находок не представляют особого смысла (3 фрагмента не имели аннотации, 3 аннотировались на белок XM_042916655.1, 1 на белок XP_042772540.1).
Список источников
//а также все гиперссылки, присутствующие в смысловой части практикума