Практикум 6
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для сравнения результатов предсказания трансмембранных участков по последовательности (с помощью DeepTMHMM) и по
трехмерной структуре (база данных OPM) в β-листовом белке сначала выберем белок, в трансмембранной части
которого находятся β-листы. Сделаем это в базе данных OPM
(orientations of proteins in membranes) с помощью поиска по уровням классификации.
Был выбран белок 1k24 - outer membrane adhesin/invasin OpcA
(адгезин/инвазин наружной мембраны OpcA). Белок из грам-отрицательной бактерии, вызывающей менингококковую
инфекцию
(Neisseria meningitidis).
Идентификатор UniProt: Q51227_NEIME. Белок считается
лиганд-связывающим и играет важную роль во взаимодействиях с эпителиальными и эндотелиальными клетками.
В таблице 1 представлены координаты трансмембранных участков моего белка. Скачаем из UniProt
последовательность белка и при ее сравнении с представленной
в OPM увидим, что последовательность белка в OPM укорочена (отстутствует первый десяток нуклеотидов).
Поэтому в итоге в DeepTMHMM на вход отправилась последовательность из OPM.
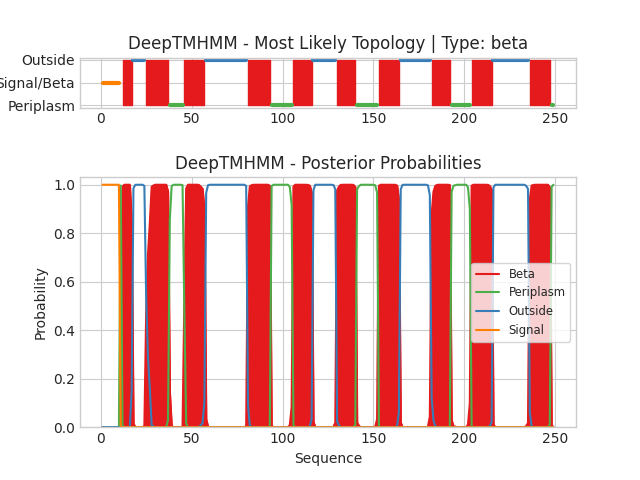
Запустим DeepTMHMM для выбранного белка. Графическое изображение результатов работы программы представлено на рисунке 1. Файл с результатами в текстовом виде.
| Номер | Координаты по базе данных OPM | Координаты по DeepTMHMM |
|---|---|---|
| 1 | 9-17 | 12-17 |
| 2 | 33-42 | 25-37 |
| 3 | 49-57 | 46-57 |
| 4 | 89-98 | 81-93 |
| 5 | 108-117 | 106-116 |
| 6 | 136-147 | 130-140 |
| 7 | 153-165 | 153-164 |
| 8 | 189-199 | 182-192 |
| 9 | 206-215 | 204-215 |
| 10 | 244-252 | 236-247 |
Результаты полученные двумя методами частично совпадают. Общее количество замеченных β-тяжей одинаково, во всех случаях они перекрываются, но их координаты отличаются.
2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для сравнения результатов предсказаний трансмембранных участков по последовательности и по AlphaFold-модели структуры в α-спиральном белке мне был выдан белок Y4364_PSEA8. Белок UPF0761 membrane protein PLES_43641 (UPF0761 мембранный белок PLES_43641) из грамотрицательной бактерии Pseudomonas aeruginosa (strain LESB58). Сведения о функции белка крайне ограничены - мембранный белок.
| Номер | Координаты по Alpha-Fold | Координаты по DeepTMHMM |
|---|---|---|
| 1 | 23-44 | 33-53 |
| 2 | 92-116 | 93-112 |
| 3 | 129-153 | 133-156 |
| 4 | 168-194 | 174-194 |
| 5 | 201-226 | 205-226 |
| 6 | 239-263 | 239-259 |
Запустим DeepTMHMM для выданного белка. Графическое изображение результатов работы программы представлено на рисунке 2. Файл с результатами в текстовом виде.
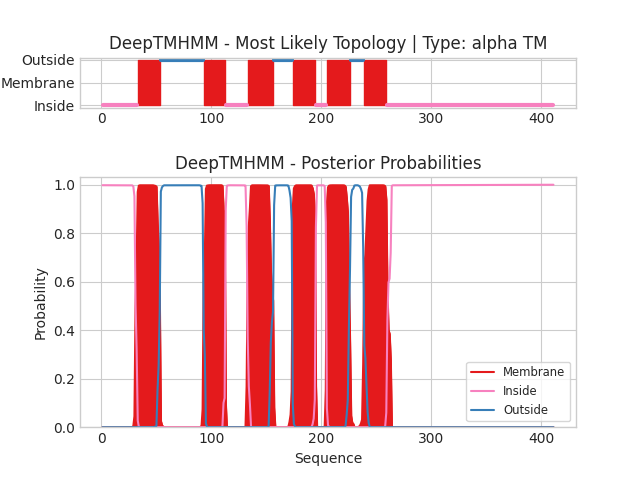
Поскольку это белок из SwissProt, для него существует предсказание трехмерной структуры, полученное с помощью AlphaFold. Загрузим этот файл. Выберем сервер PPM 3.0. Type of membrane выеберем gram-negative bacteria inner membrane, данные для параметра "Topology (N-ter)" возьмем из предсказания DeepTMHMM - in, остальные параметры оставим по умолчанию. Полученная модель, выданная программой информация о координатах трансмембранных участков в белке. Обе программы предсказали одинаковое количество трансмембранных участков. В целом, участки совпадают и отличаются погрешностями в определении их границ (получается, что координаты по DeepTMHMM слегка подрезают трансмембранные участки).