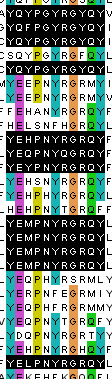Практикум 10
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Поиск консервативных мотивов в выравнивании
Для выполнения задания был выбран домен
PF00030 - Beta/Gamma crystallin.
Всего белков с этим доменом 21k, из них из swissprot 107.
Crystallin - водорастворимый структурный белок, который содержится в хрусталике и роговице
глаза, обеспечивая прозрачность структуры (также был обнаружен в других областях, таких как сердце,
и в агрессивных опухолях рака молочной железы).
Поскольку было показано, что повреждение хрусталика может способствовать регенерации нервов,
кристаллин стал предметом исследований в области нервной системы.
На данный момент было продемонстрировано, что кристаллин β b2 (crybb2) может способствовать
развитию нейронных отростков.
Согласно описанию в pfam,
выравнивание включает в себя два ключевых greek мотива, поскольку сходство между ними очень мало,
посмотрим как это отразится на поиске сигналов.
Вырванивание seed
содержит 74 последовательности.
Далее работа с выравниванием шла в Jalview, удалять идентичные последовательности не пришлось,
даже при пороге redundancy в 80% они не обнаруживались.
По внешней оценке информационного содержания при покраске Clustal c identity порогом в 50 и 60%
был выбран участок 63-73 как консервативный.
Паттерн этого мотива: Y[E,Q].P[N,G]Y.G[R,Y]QY.
В результате поиска по этому паттерну во всем выравнивании получилось 14 находок.
Результат не слишком хороший, но зато все мотивы были обнаружены в одних и тех же столбцах,
участок выравнивания представлен на рисунке 1.
Выбранный паттерн в формате prosite: Y-[EQ]-x-P-[NG]-Y-x-G-[RY]-Q-Y.
Выполним поиск по этому паттерну в базе данных SwissProt в
PROSITE. Получено
68 находок
в 65 последовательностях. Белки, в которых паттерн обнаружился дважды:
CRGA_MOUSE,
CRGA_RAT,
CRGS1_CHIID.
Для них домен Crystall встречается в последовательности не раз. В целом, мне кажется, паттерн достаточно
точно описывает искомый мотив.
2. Поиск мотива, специфичного для одной клады филогенетического дерева
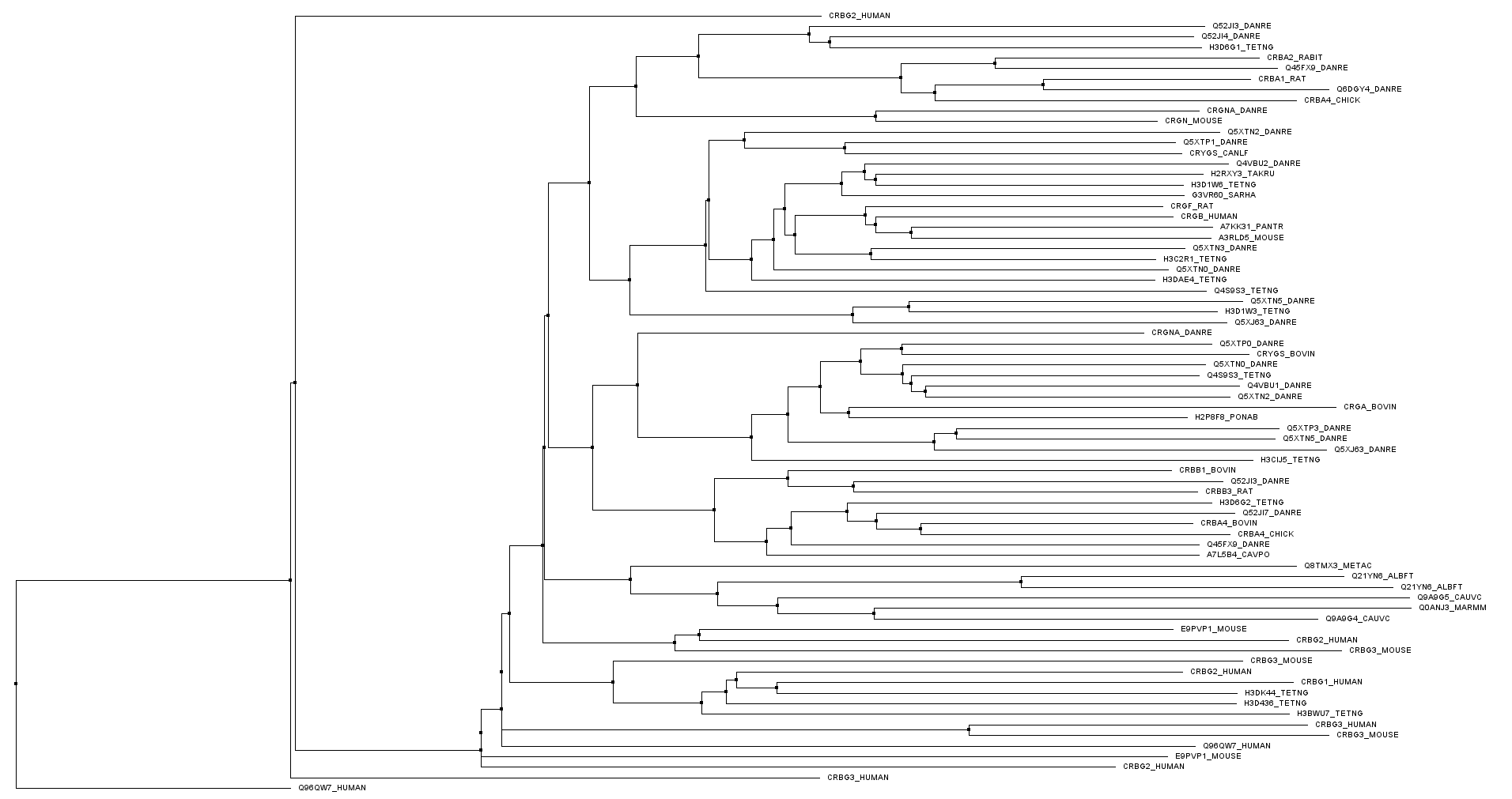
В том же выравнивании найдем мотив, специфичный для одной клады филогенетического дерева.
Построили в Jalview филогенетическое дерево методом Neighbour joining - рисунок 2.
Выделим ветвь, отрезающую одну кладу и поместим в отдельное окно ее
выравнивание: Q5XJ63_DANRE/3-84, Q5XTN5_DANRE/3-81, Q5XTP3_DANRE/4-83.
В качестве консервативного мотива выбран в этой кладе участок 4-11: FYEDRNFQ (рисунок 3).

Поиск этого мотива во всем выравнивании дал 4 находки. "Лишняя" находка была в Q5XTN2_DANRE/5-84, поэтому, на мой вгляд, можно говорить о том, что мотив встречается во всех последовательностях клады и не встречается больше нигде в выравнивании - мотив специфичен для клады.
3. PSI-BLAST
Для выполнения задания случайно был выбран идентификатор (P74518). Это фактор, способствующий гибернации рибосом (HPF_SYNY3), - белок из некоторого штамма PCC 6803 бактерии Synechocystis sp. Требуется для димеризации активных 70S-рибосом в 100S-рибосомы в стационарной фазе; 100S-рибосомы трансляционно неактивны и иногда присутствуют во время экспоненциального роста. На странице белкового BLAST в NCBI, был внесен выбранный AC в окошко, выбран PSI-BLAST и поиск по банку Swiss-Prot. После каждого запуска заполнялась строка таблицы 1. Стабилизация наступила уже на третьей итерации (контрольно проведена четвертая). E-value худшей правильной находки изначально было достаточно маленьким (e-value снижать не стала). Большинство полученных белков - ribosome hibernation promotion factor, но встречаются и исключения. В целом, семейство относительно обособлено.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value находки | Идентификатор лучшей находки ниже порога | E-value находки |
|---|---|---|---|---|---|
| 1 | 24 | P33987.1 | 3e-05 | - | - |
| 2 | 28 | P9WMA8.1 | 4e-06 | - | - |
| 3 | 28 | P24694.1 | 2e-20 | - | - |
| 4 | 28 | P24694.1 | 2e-20 | - | - |