Практикум 8
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Краткое описание одного сигнала, закодированного в геноме
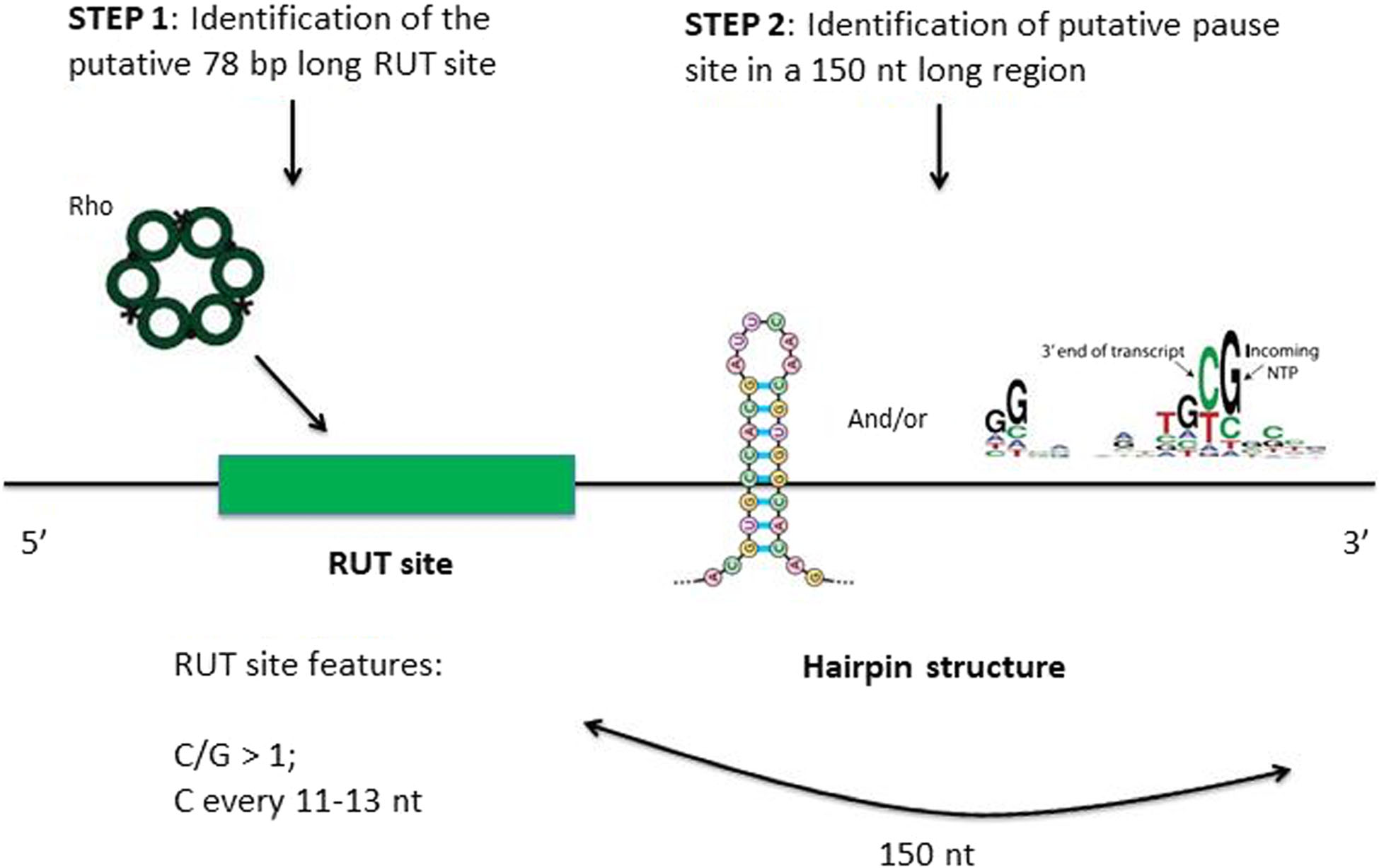
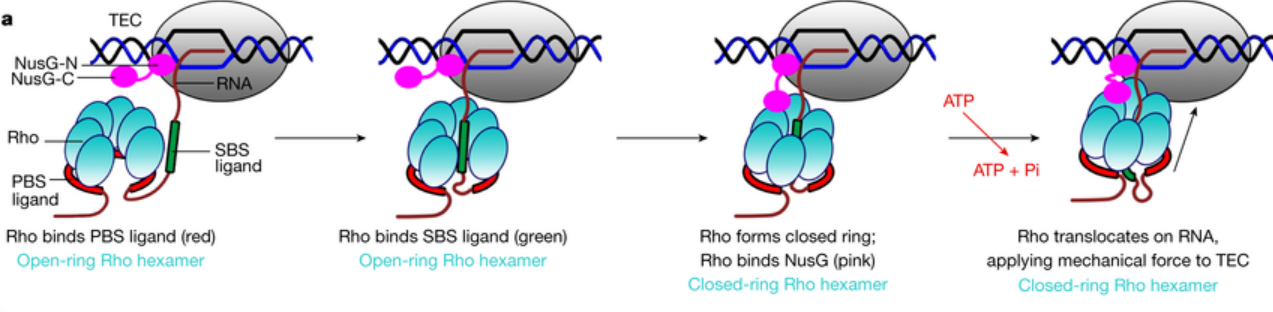
Для описания был выбран сигнал rho-зависимой терминации транскрипции у бактерий. Его носителем считается rut-сайт. Сигнал адерсован для крупной РНК-хеликазы - rho-белка. Считается, что при данном варианте терминации rho-белок сначала связывается с цитозин-богатым rut-сайтом (rho utilization site) на РНК, затем, взаимодействуя с РНК-полимеразой, вызывает терминацию транскрипции (Di Salvo et al). Однако, существует мнение, что rho-белок связывает РНК-полимеразу на ранней стадии элонгации, образуя комплекс, предшествующий терминации. Оброзование этого комплекса позоволяет rho-белку вести поиск rut-сайта и впоследствии удержать комплекс в неактивном состоянии до его диссоциации (Hao et al). Еще один вариант представлен на рисунке 1.
Личная гипотеза. Сигнал участвует в регуляции одного из важнеших биологических процессов, поэтому должен быть достаточно сильным. Однако, сила сигнала от rut-сайта будет зависеть от многих факторов, в частности, от длины и состава последовательности, структуры РНК, влияния других факторов транскрипции.
Описание алгоритма предсказания сайта rut по статье di Salvo 2019. Ученые представили алгоритм предсказания rho-зависимых терминаторов - RhoTermPredict. Программа узнает эти сайты на основе раннее предложенного консенсунсного мотива общего для всех rho-зависимых терминаторов. Она ищет rut-сайт длиной в 78 нуклеотидов с высоким содержанием цитозина, за которым следует предполагаемый сайт остановки РНК-полимеразы. Для этого происходит сканирование последовательности окнами длины 78 нуклеотидов до тех пор, пока C/G содержание окна не превысит пороговое значение в 1 и цитозины при этом должны быть распределены равномерно (через каждый 11-13 нуклеотидов). Затем рассматривается окно в 128 нуклеотидов для максимизации C/G содержания в rut-сайте. Таким образом, находится предполагаемый rut-сайт в участке длиной 128 нуклеотидов.
Далее ищется участок, где останавливается РНК-полимераза. Предполагалось, что этот сайт представляет собой либо
шпильку, либо присутствовал особый элемент
Последовательность rut-сайта в hlyA. Последовательность мРНК гена гемолизина А E. coli. Воспользуемся программой, указанной в статье, чтобы попытаться найти сигнал rho-зависимой терминации в выбранной последовательности. Выдача программы.
Список литературы к первому заданию
- Di Salvo, Marco & Puccio, Simone & Peano, Clelia & Lacour, Stéphan & Alifano, Pietro. (2019). RhoTermPredict: An algorithm for predicting Rho-dependent transcription terminators based on Escherichia coli, Bacillus subtilis and Salmonella enterica databases. BMC Bioinformatics. 20. 117. 10.1186/s12859-019-2704-x.
- Hao, Zhitai & Svetlov, Vladimir & Nudler, Evgeny. (2021). Rho-dependent transcription termination: a revisionist view. Transcription. 12. 1-11. 10.1080/21541264.2021.1991773.
- Molodtsov, Vadim & Wang, Chengyuan & Firlar, Emre & Kaelber, Jason & Ebright, Richard. (2023). Structural basis of Rho-dependent transcription termination. Nature. 614. 10.1038/s41586-022-05658-1.
2. Поиск последовательностей представителей для одного из сигналов в геноме, построение PWM и оценка результатов поиска по этой PWM новых сайтов
Для выполнения задания была выбрана окрестность ATG кодона (последовательность Козак) - старта транскрипции в геноме человека (список генов). Воспользуемся скриптом, заимствованным у Начатого Влада. На вход ему подается только аннотированная таблица генов человека. Матрица строилась по участкам из 7 нуклеотидов до старт-кодона и 3 нуклеотидов после него. Выходные данные программы:
- Материал обучения - выравнивание (без гэпов) последовательностей сигнала для построения PWM
- Материал тестирования - последовательности, в которых ожидается наличие сигнала
- Материал негативного контроля - последовательности, в которых не должно быть сигнала
- PWM - позиционная весовая матрица, построенная на материале обучения (таблица 1)
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.676 | -0.676 | -0.126 | -0.089 | 0.05 | -0.089 | -0.34 | 1.218 | -5.691 | -5.691 | -0.34 | -0.052 | -0.819 |
| C | 0.59 | 0.312 | 0.347 | 0.071 | -0.248 | 0.717 | 0.892 | -5.327 | -5.327 | -5.327 | -0.381 | 0.643 | 0.275 |
| G | 0.198 | 0.741 | 0.312 | 0.563 | 0.786 | -0.312 | 0.115 | -5.327 | -5.327 | 1.582 | 0.851 | -0.248 | 0.717 |
| T | -0.249 | -0.745 | -0.551 | -0.676 | -1.305 | -0.612 | -1.593 | -5.691 | 1.218 | -5.691 | -0.551 | -0.551 | -0.494 |
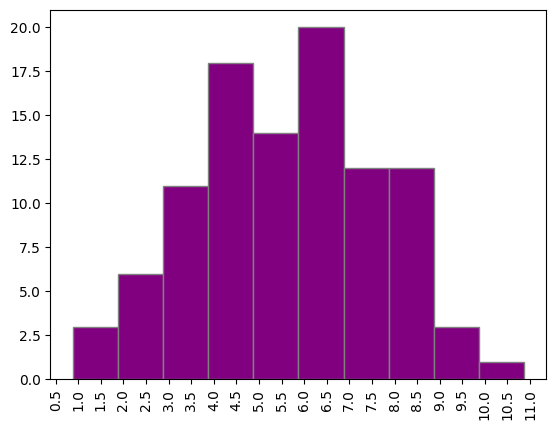
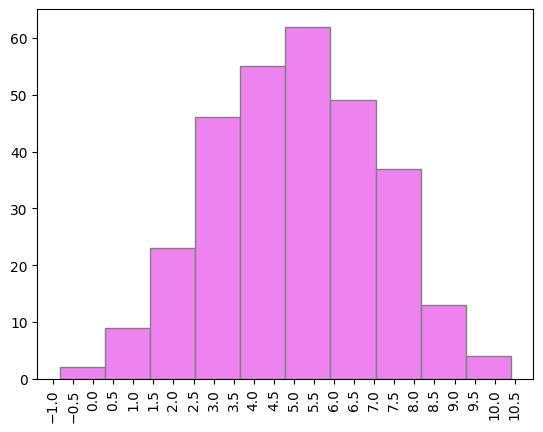
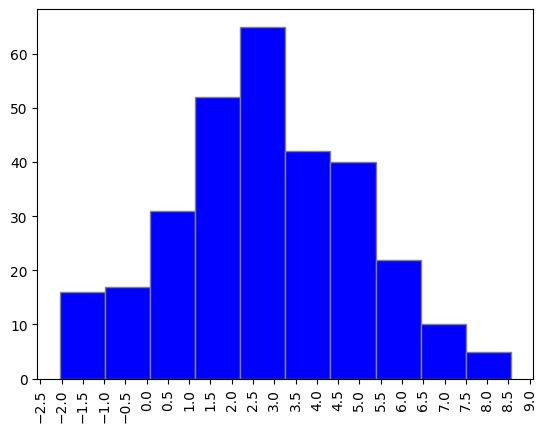
Для всех последовательностей из трех выборок были вычислены веса, гистограммы весов представлены на рисунках 3-5. По гистограммам определим порог веса, выше которого находка считается правильной, равным 3,5. При таких и больших весах начинает обнаруживаться большое количество находок в тестовой выборке, при этом отсеивается большая часть негативной выборки.
С учетом порогового значения в 3,5 построим таблицу результатов проверки - таблица 2. Обучающая выборка в основном состояла из последовательностей, в которых сигнал присутствовал, в положительном тесте действительно получалось выявлять сигнал намного чаще, чем в отрицательном тесте.
| Обучающая выборка | Тестовая выборка | Негативная выборка | |
|---|---|---|---|
| Сигнал (+) | 83 (83%) | 230 (77%) | 111 (37%) |
| Сигнал (-) | 17 (17%) | 70 (23%) | 189 (63%) |
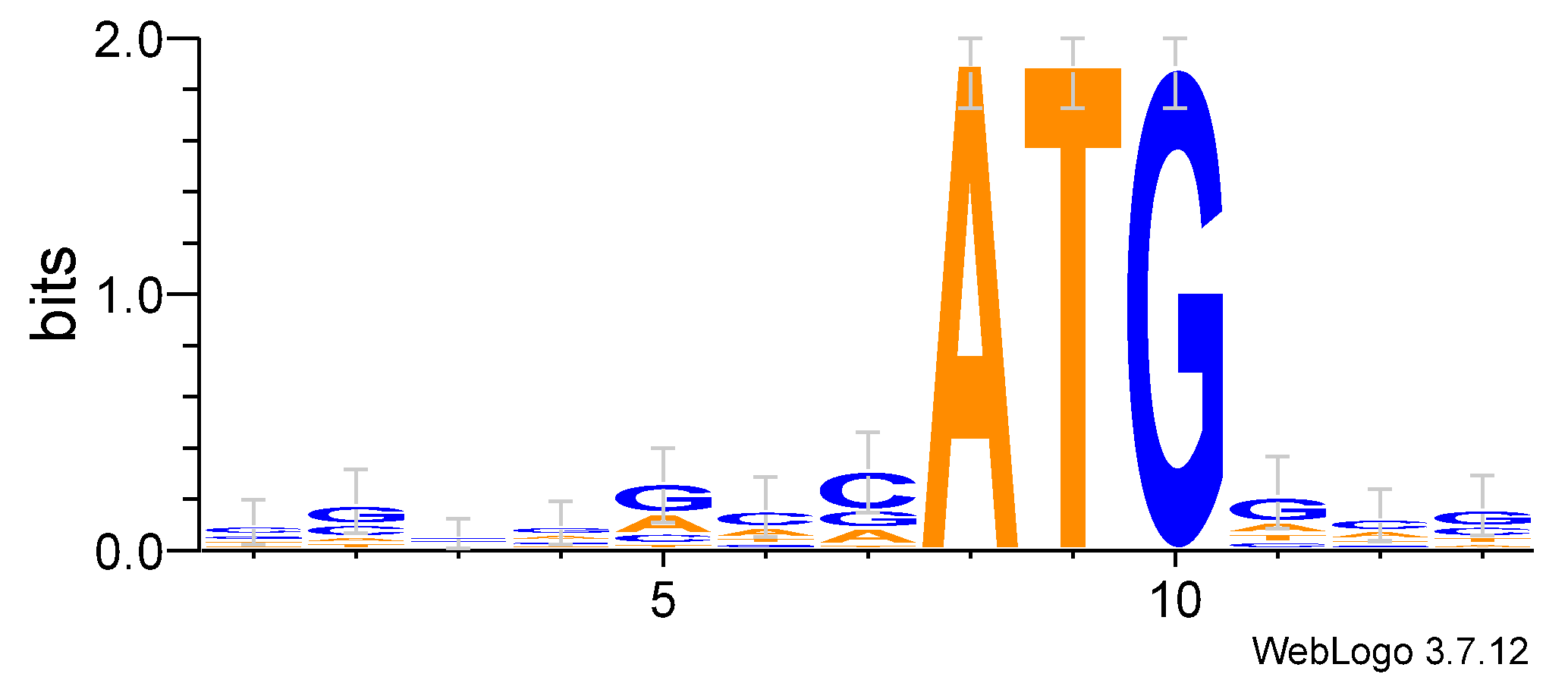
Вычислим IC (информационное содержание) выравнивания. Подадим на вход скрипту Влада Начатого нашу обучающую выборку и получим матрицу IC (таблица 3). Для построения LOGO используем сервис WegLOGO 3, которому тоже вход подаем обучающую выборку - рисунок 3. Видим по IC и LOGO, что окрестность ATG кодона достаточно консервативна и обладает некоторым, хотя и слабовыраженным, но консенсусом.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.146 | -0.146 | -0.047 | -0.034 | 0.022 | -0.034 | -0.103 | 1.761 | 0.0 | 0.0 | -0.103 | -0.021 | -0.154 |
| C | 0.315 | 0.126 | 0.145 | 0.022 | -0.057 | 0.435 | 0.643 | 0.0 | 0.0 | 0.0 | -0.077 | 0.362 | 0.107 |
| G | 0.072 | 0.46 | 0.126 | 0.292 | 0.51 | -0.068 | 0.038 | 0.0 | 0.0 | 2.286 | 0.589 | -0.057 | 0.435 |
| T | -0.083 | -0.151 | -0.135 | -0.146 | -0.151 | -0.141 | -0.138 | 0.0 | 1.761 | 0.0 | -0.135 | -0.135 | -0.128 |