Практикум 9. Поиск de novo сигналов в ДНК
Автор старался, но не может гарантировать отсутствие биологических ошибок.
В практикуме будет происходить поиск сигнала посадки сигма-субъединицы РНК-полимеразы в промоторах генов генома бактерии Mycobacterium tuberculosis. Бактерия известная, возбудитель туберкулёза, интересно ее исследовать.
Подготовка данных
Был скачен fasta-файл с хромосомой M. tuberculosis из GenBank, а также аннотация - gff-файл. Теперь необходимо найти опероны, ведь, по определению, сайты связывания σ-субъединицы есть только в промоторах перед оперонами. Используем сервис Operon-mapper, подадим ему наш fasta-файл и список генов. Выходной файл: список оперонов. Для дальнейшего анализа потребовался скрипт (заимствован у Георгия Муравьева). На вход подавались список оперонов и геном M. tuberculosis. Промотором считалась область в 100 нуклеотидов перед началом оперона. Cгенерировались три выборки:
Поиск с помощью MEME
Воспользуемся локальным MEME на kodomo. Запускаем следующей командой:
meme train.fasta -dna -nmotifs 3 -minw 6
В результате получилась папка meme_out, хранящая файлы выдачи, в том числе html-файл. Программе было задано найти 3 мотива и она их нашла: рисунки 1-3. Для дальнейшей проверки выбираю первый мотив, потому что для него e-value лучше (5.2e-10). Его консенсусная последовательность: CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC.
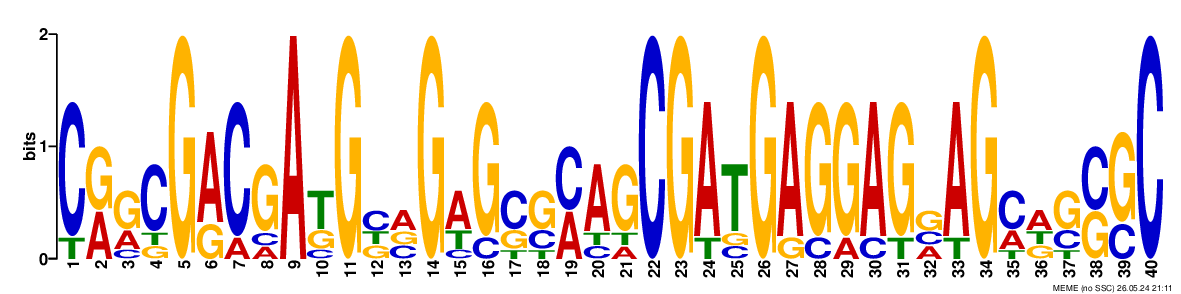
Поиск с помощью FIMO
Выполним поиск с помощью FIMO среди всех отобранных промоторов (положительный контроль - тестовая выборка), и в выборке для негативного контроля. Для FIMO тоже будем запускать консольную версию на kodomo:
fimo --norc -motif CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC -thresh 0.001 meme_out/meme.txt test.fasta
fimo --norc -motif CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC -thresh 0.001 meme_out/meme.txt neg.fasta
Выдача снова получилась в виде двух папок fimo_out. HTML-файлы для
положительного и
негативного контролей.
Для положительного контроля получили 2390 находок, для негативного - 263 (порог = 0.001).
Выглядит очень плохо, потому число всех отобранных промоторов - 4080, размер негативной выборки - 270.
Поэтому решено было запустит FIMO для какого-нибудь другого мотива - второго:
fimo --norc -motif TCTTGCCTTTDACWTCBAYCA -thresh 0.001 meme_out/meme.txt test.fasta
fimo --norc -motif TCTTGCCTTTDACWTCBAYCA -thresh 0.001 meme_out/meme.txt neg.fasta
Получили еще две папки fimo_out. HTML-файлы для
положительного и
негативного контролей.
К сожалению, тут дело только немного лучше:
для положительного контроля получили 265 находок, для негативного - 14 (порог = 0.001).
Результат стал "вернее" для негативной выборки - число находок уменьшилось.
P.S. Для третьего мотива (положительные и негативные) ситуация для промоторов тоже была печальная: 41 и 0 находок соотвественно.

