Поиск гипотетических гомологов изучаемого белка в разных банках
Таблица 1. Результаты поиска гипотетических гомологов белка XXXX_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P40406.1 | 3BMX_A | NP_388047.1 |
| E-value | 0.0 | 0.0 | 0.0 |
| Вес (в битах) | 1320 | 1320 | 1320 |
| Процент идентичности | 100% | 100% | 100% |
2. Число находок с E-value < 10–10 |
134 | 21 | 7351 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 184 | 28 | 10331 |
| Accession | A4G7V9.1 | 2NU9_A | YP_003160439.1 |
| E-value | 0.86 | 0.99 | 0.97 |
| Вес (в битах) | 35.4 | 32.3 | 41.6 |
| % идентичности | 37% | 34% | 21% |
| % сходства | 61% | 46% | 42% |
| Длина выравнивания | 49 | 76 | 217 |
| Координаты выравнивания (от-до, в запросе и в находке) | 517-565; 132-176 | 224-286; 40-112 | 117-318; 668-874 |
| Число гэпов | 4 | 16 | 25 |
Мне удалось найти исходный белок в Swiss-Prot и его структуру 3BMX в банке PDB. Разумеется в банке PDB структур намного меньше белков, так как не для всех описанных белков есть смоделированные структуры. В Non-redundant protein sequences ищется по всем базам, поэтому результатов в сотни раз больше.
Swiss-Prot последняя находка E-value = 8.4
PDB последняя находка E-value = 7.9
Non-redundant protein sequences последняя находка E-value = 9.8
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Задача - найти лучшего гомолога ybbD_bacsu, филогенетически как можно более далекого.
Были проверены следующие таксоны:
- 'Eukaryota' (другое царство);
- 'Actinobacteria' (другой отдел того же царства бактерий);
- 'Clostridia' (другой класс того же отдела Firmicutes);
- 'Lactobacillales' (другой порядок того же класса Bacilli);
- 'Listeriaceae' (другое семейство того же порядка Bacillales);
- 'Geobacillus' (другой род того же семейства Bacillaceae);
- 'Bacillus anthracis' (другой вид того же рода).
Таблица 1. Результаты поиска гипотетических гомологов изучаемого белка с фильтром по таксонам
| Eukaryota | Actinobacteria | Clostridia | Lactobacillales | Listeriaceae | Geobacillus | Bacillus anthracis | |
Результаты с фильтром таксонов. |
|||||||
| Номер находки в списке описаний | 1 | 1 | 1 | Не найдено | Не найдено | Не найдено | Не найдено |
| Accession | Q5BCC6.1 | Q7WUL3.1 | P14002.2 | . | . | . | . |
| E-value | 4e-24 | 1e-42 | 1e-13 | . | . | . | . |
| Вес (в битах) | 109 | 161 | 72.4 | . | . | . | . |
| % идентичности | 23% | 26% | 26% | . | . | . | . |
| % сходства | 41% | 44% | 44% | . | . | . | . |
| Длина выравнивания | 625 | 553 | 330 | . | . | . | . |
| Координаты выравнивания (от-до, в запросе и в находке) | 38-618; 52-585 | 42-582; 26-528 | 136-459; 68-339 | . | . | . | . |
| Число гэпов | 135 | 62 | 64 | . | . | . | . |
BLAST двух последовательностей
Было выполнено парное выравнивание с помощью BLAST белка ybbd_bacsu и гомолога из царства Eukaryota, сначала с порогом на E-value 10, затем с порогом 0.01. Получены карты локального сходства для двух выравниеваний (Рис.1-2).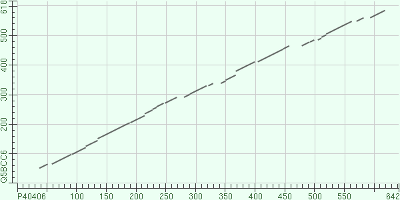

Рис.1-2. Карты локального сходства двух парных выравниваний белка ybbd_bacsu и его гомолога из царства Eukaryota с порогом на E-value 10 и 0.01 соответственно.
Как можно заметить, изменение E-value никак не повлияло на карту локального сходства.
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com