Составление репрезентативной выборки гомологов белка ybbD_BACSU с помощью BLAST
Для составление репрезентативной выборки гомологов белка был произведен поиск с помощью BLAST среди большинствва филумов прокариот и среди эукариот. Такой поиск не ограничивает число результатов, но облегчает работу с более чем 4 тысячами хитов. Для поиска по каждому филуму прокариот (по базе RefSeq) в поле Organism исключались эукариоты и включался соответствющий филум бактерий. Порог по e-value бфл установлен везде одинаковый - 10е-5 (такой высокий, потому что выдается колоссальное количество хитов, столько и не нужно). Далее из результатов выбиралось несколько наиболее идентичных последовательностей. Потом из них составлялось дерево в GenBank. Результаты поиска можно посмотреть в таблице 1, а последовательности отобранных белков в фаста-формате можно скачать здесь.
Результаты поиска с помощью BLAST
| Группа | Ограничения по таксонам | Порог по e-value | Максимальное количество хитов | Число взятых в выборку | Идентификаторы в RefSeq |
| a-proteobacteria | a-proteobacteria | 10e-5 | 607 | 1 | YP_001832004.1 |
| b-proteobacteria | b-proteobacteria | 10e-5 | 322 | 2 | ZP_02355965.1, ZP_02463814.1 |
| d-proteobacteria | d-proteobacteria | 10e-5 | 97 | 1 | ZP_21237458.1 |
| g-proteobacteria | g-proteobacteria | 10e-5 | 1775 | 5 | ZP_23550178.1, ZP_15940755.1, YP_341561.1, ZP_08408924.1, ZP_10279984.1 |
| cyanobacteria | cyanobacteria | 10e-5 | 144 | 2 | ZP_05025562.1, YP_478540.1 |
| chlorobi group | chlorobi group | 10e-5 | 1374 | 4 | ZP_20983629.1, YP_006433559.1, YP_004238974.1, YP_006426314.1 |
| Actinobacteria | Actinibacteria | 10e-5 | 1023 | 3 | ZP_03298131.1, YP_643411.1, ZP_08287378.1 |
| Spirochaetes | Spirochaetes | 10e-5 | 216 | 2 | YP_005061787.1, ZP_09659728.1 |
| Tenericutes | Tenericutes | 10e-5 | 3 | 1 | YP_001620280.1 |
| Chlamydiae | Chlamydiae | 10e-5 | 25 | 1 | ZP_06299899.1 |
| Euryarchaeota | Euryarchaeota | 10e-5 | 114 | 1 | YP_004785994.1 |
| Остальные | exclude (Eukarya, Proteobacteria, Euryarchaeota, Chlamidiae, Tenericutes, Spirochaetes, Actinibacteria, Chlorobo group, Cyanobacteria, Tenericutes) | 10e-5 | 262 | 3 | YP_003322188.1, ZP_08554226.1, ZP_06392296.1 |
Табл.1. Таблица параметров поиска BLAST и отобранных в результате белков.
На рисунке 1 представлена репрезентативность выборки по прокариотам.

Рис.1. Распределение выбранных белков по таксонам прокариот.
Встречаемость белка у эукариот
Для того, чтобы оценить встречаемость белка ybbD_BACSU у эукариот, был запущен поиск BLAST по Eukaryota. Т.к. хитов вышло немного, сразу все результаты были отправлены в GenBank, составлено дерево и выбрано 11 последовательностей из разных таксонов. Их таксономическую связь можно увидеть на рисунке 2. Последовательности этих белков в фаста-формате можно скачать здесь.
Рис.2. Распределение выбранных белков по таксонам эукариот.
В таблице 2 можно ознакомиться со всеми отобранными организмами, у которых обнаружен гомолог белка ybbD_BACSU.
| Домен | Филум | Организм | Белок |
| Archaea | Euryarchaeota | Haloarcula hispanica ATCC 33960 | YP_004785994.1 |
| Bacteria | Actinobacteria | Collinsella stercoris DSM 13279 | ZP_03298131.1 |
| Actinobacteria | Rubrobacter xylanophilus DSM 9941 | YP_643411.1 | |
| Actinobacteria | Streptomyces griseoaurantiacus M045 | ZP_08287378.1 | |
| Bacteroidetes/Chlorobi group | Fulvivirga imtechensis AK7 | ZP_20983629.1 | |
| Bacteroidetes/Chlorobi group | Flexibacter litoralis DSM 6794 | YP_006433559.1 | |
| Bacteroidetes/Chlorobi group | Weeksella virosa DSM 16922 | YP_004238974.1 | |
| Bacteroidetes/Chlorobi group | Ornithobacterium rhinotracheale DSM 15997 | YP_006426314.1 | |
| Chlamydiae/Verrucomicrobia group | Parachlamydia acanthamoebae str. Hall's coccus | ZP_06299899.1 | |
| Cyanobacteria | Coleofasciculus chthonoplastes PCC 7420 | ZP_05025562.1 | |
| Cyanobacteria | Synechococcus sp. JA-2-3B'a(2-13) | YP_478540.1 | |
| Firmicutes | Bacillus subtilis subsp. subtilis str. 168 | ybbD_BACSU | |
| Proteobacteria | Beijerinckia indica subsp. indica ATCC 9039 | YP_001832004.1 | |
| Proteobacteria | Burkholderia oklahomensis EO147 | ZP_02355965.1 | |
| Proteobacteria | Burkholderia thailandensis MSMB43 | ZP_02463814.1 | |
| Proteobacteria | Cystobacter fuscus DSM 2262 | ZP_21237458.1 | |
| Proteobacteria | Morganella morganii SC01 | ZP_23550178.1 | |
| Proteobacteria | Morganella morganii subsp. morganii KT | ZP_15940755.1 | |
| Proteobacteria | Pseudoalteromonas haloplanktis TAC125 | YP_341561.1 | |
| Proteobacteria | Pseudoalteromonas haloplanktis ANT/505 | ZP_08408924.1 | |
| Proteobacteria | Pseudoalteromonas arctica A 37-1-2 | ZP_10279984.1 | |
| Spirochaetes | Sphaerochaeta pleomorpha str. Grapes | YP_005061787.1 | |
| Spirochaetes | Leptonema illini DSM 21528 | ZP_09659728.1 | |
| Synergistetes | Dethiosulfovibrio peptidovorans DSM 11002 | ZP_06392296.1 | |
| Tenerecutes | Acholeplasma laidlawii PG-8A | YP_001620280.1 | |
| unclassified Bacteria | Thermobaculum terrenum ATCC BAA-798 | YP_003322188.1 | |
| unclassified Bacteria | Haloplasma contractile SSD-17B | ZP_08554226.1 | |
| Eukaryota | Archaeplastida | Zea mays | NP_001146552.1 |
| Archaeplastida | Fragaria vesca subsp. vesca | XP_004301317.1 | |
| Archaeplastida | Glycine max | XP_003554030.1 | |
| Chromalveolata | Phytophthora infestans T30-4 | XP_002907074.1 | |
| Opisthokonta | Trichoplax adhaerens | XP_002118719.1 | |
| Opisthokonta | Puccinia graminis f. sp. tritici CRL 75-36-700-3 | XP_003307054.2 | |
| Opisthokonta | Myceliophthora thermophila ATCC 42464 | XP_003665722.1 | |
| Opisthokonta | Sordaria macrospora k-hell | XP_003352972.1 | |
| Opisthokonta | Penicillium chrysogenum Wisconsin 54-1255 | XP_002568350.1 | |
| Opisthokonta | Aspergillus oryzae RIB40 | XP_001820553.2 | |
| Opisthokonta | Sclerotinia sclerotiorum 1980 | XP_001591716.1 |
Табл.2. Встречаемость белка в различных таксонах.
Множественное выравнивание гомологов белка ybbD_BACSU
Было проведено множественное выравнивание гомологов белка при помощь программы
Muscle. Её результаты можно видеть на рисунках 3-4.

Рис.3. Выравнивание гомологов белка ybbd_BACSU из прокариот.

Рис.4. Выравнивание гомологов белка ybbd_BACSU из эукариот. Использовано окрашивание Clustalx. ДЛя того, чтобы развернуть рисунки, просто откройте их в новой вкладке.
Из этих рисунков можно заметить, что лучше выравниваются последовательности прокариот между собой, эукариотические же сохранили же несколько важных блоков, но с большей изменчивостью в составе.
Результаты анализа множественного выравнивания гомологов белка ybbD_BACSU
Для анализа были добавлены дополнительные строки аннотации: Secondary, Ligand, Blocks.SECONDARY: отражает нахождение альфа-спиралей и бета-листов в структуре 3BMX белка ybbD_BACSU.
LIGAND: отражает, те АК, которые связывают лиганды - ионы натрия, находящий рядом с ними ацетат-ион, а также исследуемый ранее лиганд P4G.
BLOCKS: отражает блоки на участках выравнивания.
Получившийся результат можно рассмотреть на рисунке 5.

Рис.5. Выравнивание со всеми последовательностями как из прокариот, так из эукариот. Добавлены новые строки аннотации. Окрашивание АК: His, Arg, Lys - розовый; Glu, Asp - голубой; Ala, Leu, Ile, Met, Val - серый; Phe, Trp, Tyr - синий; Asn, Gln - темно-зеленый; Cys - желтый; Pro - салатовый. Окрашивание по консервативности с порогом 30%.
После анализа множественного выравнивания стало ясно, что лучше (с большим процентом идентичности) выравнялись прокариотические последовательности.
Но абсолютно все сиквенсы имееют несколько участков, которые мало отличаются друг от друга, такие участки были объединены в блоки
(см. рис.5). Трудно угадать какую-то зависимость элементов вторичной структуры от консервативных участков, но с уверенностью можно сказать,
что аминокислоты, связывающие ионы натрия и ацетат-ион, находятся в блоках, а это значит, что они
несут функциональну нагрузку и необходимы для выполнения белком своих функций. Посмотреть, как связан лиганд в структуре 3BMX белка ybbD_BACSU можно
на рисунках 6-7.

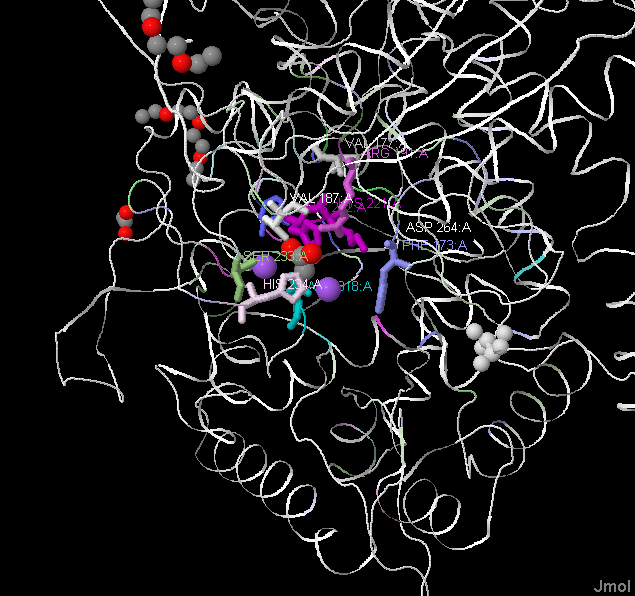
Рис.6-7. Связывание лигандов Na и ацетат-иона.
HIS222 и LYS221 строго консервативны у всех, у прокариот консервативны также Ser233, Val187, Arg191, у эукариот они заменяются на Тhr (Gly,Glu,Asn), Gly (Arg) и Glu соответственно. Phe173 рандомно заменяется на тирозин, лейцин и метионин. Val177 может заменяться на изолейцин, аланин или тирозин. Из этого можно сделать вывод, что лиганд-связывающие аминокислоты консервативны и заменяются только на схожие по радикалу АК. (Взаимозаменяемость тирозина и АК с гидрофобными радикалами можно объяснить тем, что тирозин своим фенольным кольцом может обеспечивать те же гидрофобные взаимодействия).
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com