Реконструкция деревьев по нуклеотидным последовательностям. Деревья, содержащие паралоги.
Построение дерева по нуклеотидным последовательностям
| Бактерия | Сокращенное название | AC | Координаты 16S рРНК | Цепь |
| Bacillus subtilis | BACSU | AF008220 | 485-2034 | Прямая |
| Clostridium tetani | CLOTE | AЕ015927 | 8715-10223 | Обратная |
| Enterococcus faecalis | ENTFA | AЕ016830 | 1018187-1019708 | Прямая |
| Finegoldia magna | FINM2 | AP008971 | 611796-613319 | Прямая |
| Lactococcus lactis | LACLM | AМ406671 | 511423-512971 | Прямая |
| Staphylococcus epidermidis | STAES | AE015929 | 1722288-1723841 | Обратная |
| Streptococcus pyogenes | STRP1 | AЕ004092 | 17169-18503 | Прямая |
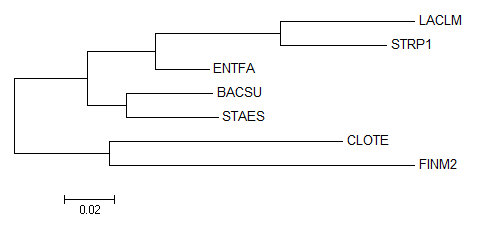
Рис.1. Дерево, построенное на основе нуклеотидных последовательностей 16S рРНК.
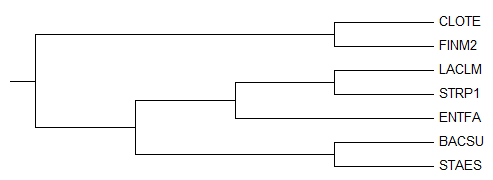
Рис.2. Идеальное дерево.
Можно заметить, что деревья обсолютно одинаковые, что говорит о том, что некодирующие нуклеотидные последовательности рРНК лучше отражают родство организмов, чем белковые.
Построение и анализ дерева, содержащего паралоги
Из файла proteo.fasta были отобраны все гомологи белка CLPX_BACSU с e-value меньше 0.001, далее из них были отобраны только принадлежащие изучаемым бактериям:CLPX_BACSU CLPX_STAES CLPX_CLOTE CLPX_ENTFA CLPX_LACLM B0S2N5_FINM2 CLPX_STRP1 J7M389_STRP1 HSLU_ENTFA CLPY_BACSU HSLU_STAES Q890L5_CLOTE CLPC_BACSU CLPE_BACSU A2RIW9_LACLM Q899V4_CLOTE CLPC_STAES B0S3X9_FINM2 B0S0E3_FINM2 Q99XR9_STRP1 J7MBF9_STRP1 J7M6I1_STRP1 Q9A200_STRP1 Q82YZ7_ENTFA Q899H3_CLOTE B0S3J0_FINM2 Q891B9_CLOTE Q837W9_ENTFAПоследовательности этих белков были выровняны с помощью JalView и программы muscle, затем составлено дерево в программе MEGA алгоритмом Maximum Likelihood.
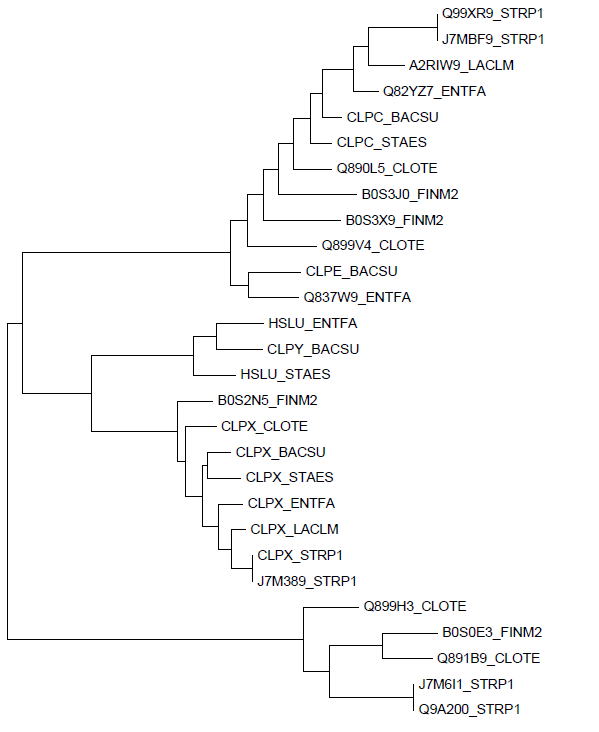
Рис.2. Дерево гомологов белка CLPX_BACSU.
Паралоги Q899H3_CLOTE и Q891B9_CLOTE или B0S3X9_FINM2 и B0S3J0_FINM2.
Ортологи, например, CLPX_BACSU и CLPX_STAES.
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com