Таксономия выбранных бактерий
| Bacillus subtilis | BACSU | Firmicutes, Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Firmicutes, Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Firmicutes, Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | FINM2 | Firmicutes, Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Lactococcus lactis | LACLM | Firmicutes, Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Staphylococcus epidermidis | STAES | Firmicutes, Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pyogenes | STRP1 | Firmicutes, Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
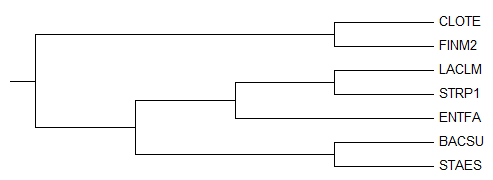
Рис.1.Филогенетическое дерево отобранных бактерий.
Реконсрукция дерева белков
Для работы выбрано семейство факторов инициации трансляции 2 (IF2). С помощью команды seqret
были скачены последовательности всех IF2 выбранных бактерий. Затем с помощью JalView и программы muscle
эти последовательности были выровнены, окрашены по проценту идентичности и представлены в блочной форме
с шириной блока 100 нуклеотидов(Рис.2).

Рис.1.Привильное филогенетическое дерево отобранных бактерий.

Рис.2. Выравнивание факторов инициации трансляции 2 выбранных бактерий с окраской по проценту идентичности.
Затем 4 различными способами в JalView были построены филогенетические деревья последовательностей (Рис.3-6).
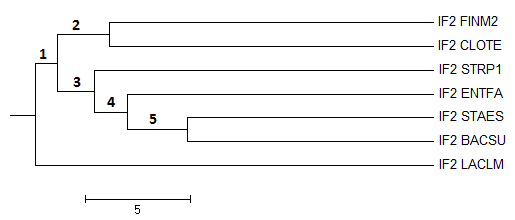
Рис.3. Дерево, созданное с помощью алгоритма Average Distance Using % Identity. Ветви 2,3,5 есть в правильном дереве - это 1,2,4 соответственно.
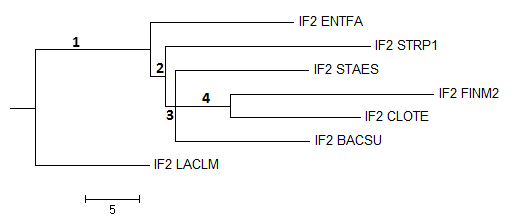
Рис.4.Дерево, созданное с помощью алгоритма Neighbour Joining Using % Identity Ветвь 4 есть на правильном дереве - это 1 ветвь.
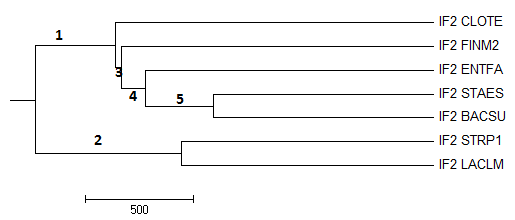
Рис.5. Дерево, созданное с помощью алгоритма Average Distance Using BLOSUM62 Ветвь 2, 5 есть на правильном дереве - это 5, 4 соответственно.
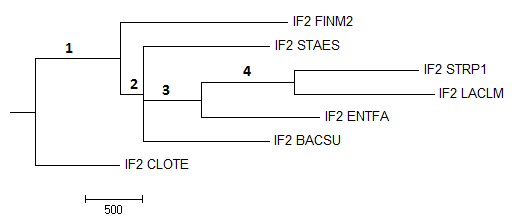
Рис.6. Дерево, созданное с помощью алгоритма Neighbour Joining Using BLOSUM62 Ветвь 2,3,4 есть на правильном дереве - это 2, 3, 5 соответственно.
Далее с помощью MEGA, используя алгоритм Maximum Parsimony, было построено ещё одно дерево этих
последовательностей. Верное укоренение позволило составить дерево идентичное правильному (Рис.7).
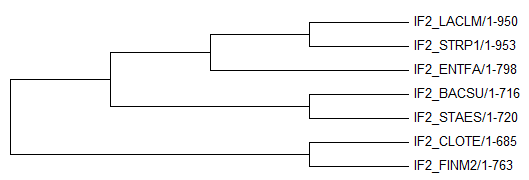
Рис.7. MEGA дерево последовательностей IF2 отобранных бактерий.
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com