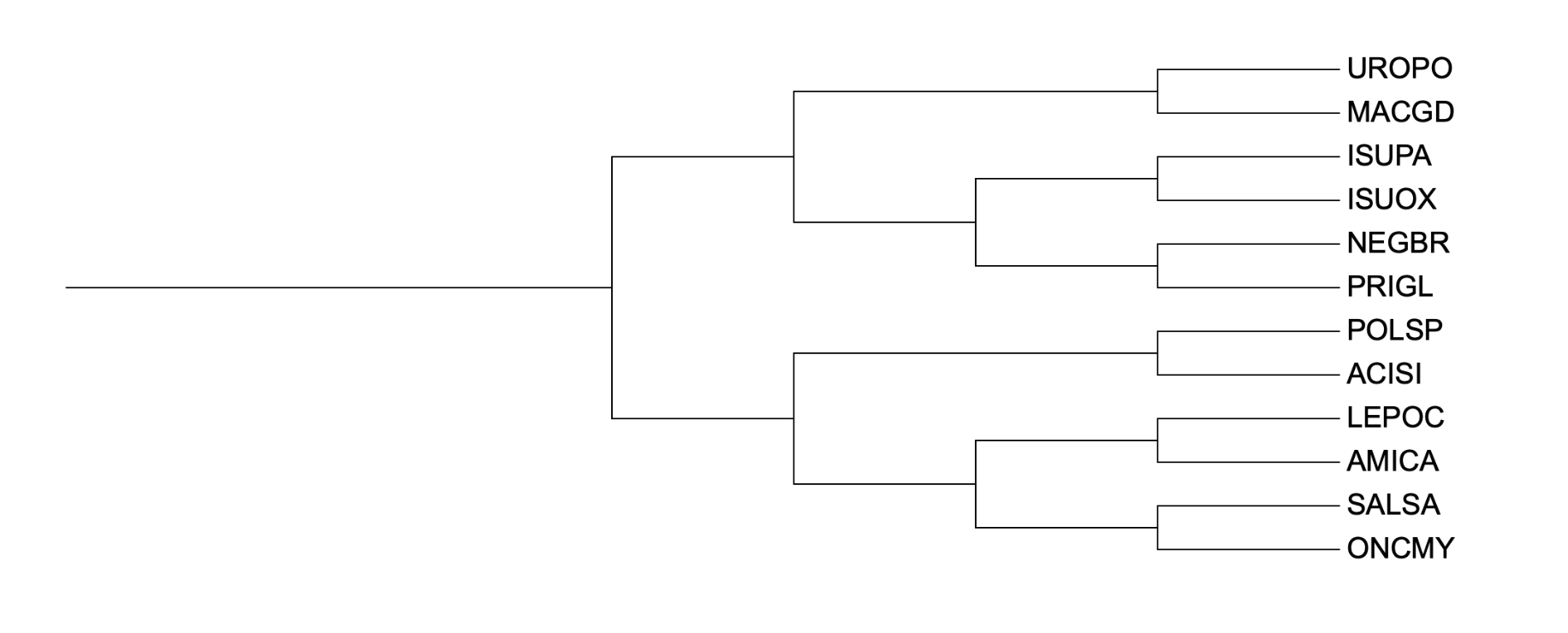
Для реконструкции филогенетического дерева использовались нуклеотидные последовательности гена 12S р-РНК митохондрий. Дерево было построено с использованием программы FastME.
В ходе анализа выяснилось, что у некоторых организмов, представленных в дереве по цитохрому B, отсутствуют полные митохондриальные последовательности 12S р-РНК.
Поэтому пришлось заменить их на близкородственные виды с доступными полными геномами. Внесенные замены:
Latimeria chalumnae (LATCHA) – Внешняя группа
Acipenser sinensis (ACISI) – Без изменений
Polyodon spathula (POLSP) – Без изменений
Amia calva (AMICA) – Без изменений
Lepisosteus platostomus (LEPPL) – Замена Lepisosteus oculatus (LEPOC)
Oncorhynchus mykiss (ONCMY) – Без изменений
Salmo salar (SALSA) – Без изменений
Maculabatis gerrardi (MACGD) – Без изменений
Hemiscyllium dahlia (HIDAL) – Замена Urogymnus polylepis (UROPO)
Isurus paucus (ISUPA) – Без изменений
Isurus oxyrinchus (ISUOX) – Без изменений
Negaprion brevirostris (NEGBR) – Без изменений
Prionace glauca (PRIGL) – Без изменений
Таким образом, в новой реконструкции учитывались доступные полные последовательности 12S рРНК, что позволило получить более точное дерево.

Следующим шагом была проведена реконструкция дерева (рис.3) на основе выравнивания нуклеотидных последовательностей гена 12S рРНК .
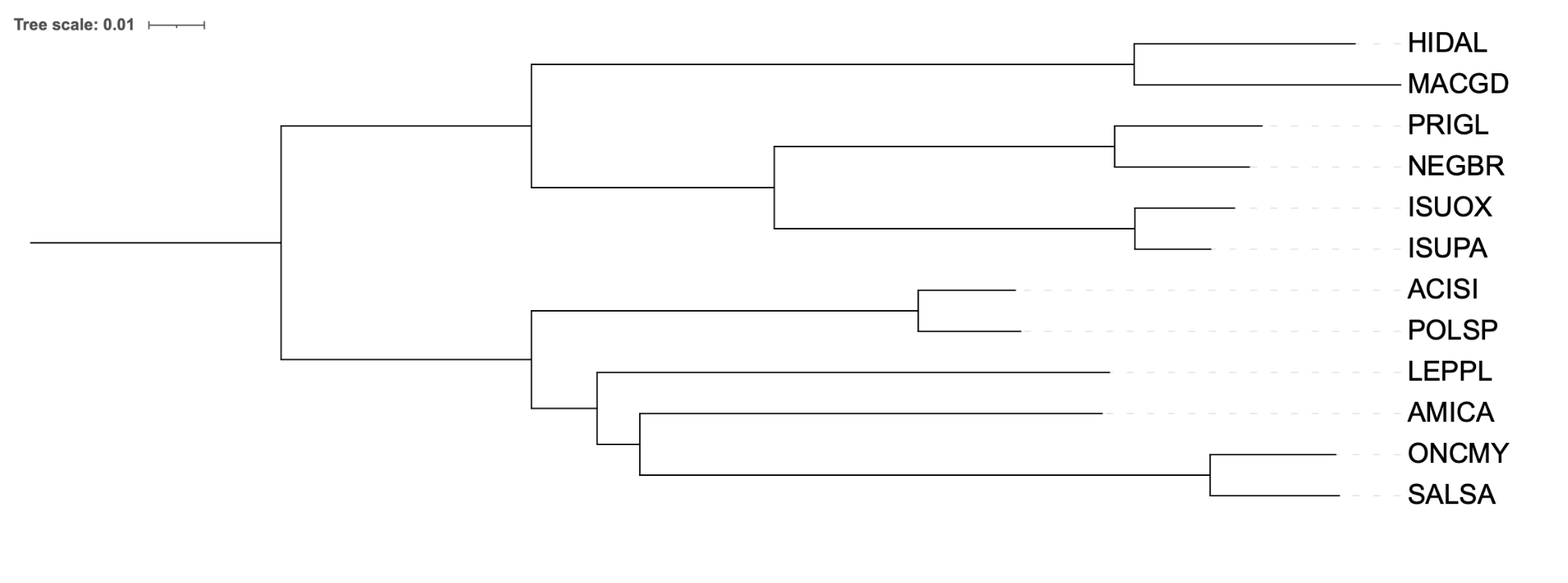
В таксономическом дереве есть клада состава (((SALSA,ONCMY),(LEPOC,AMICA)),(POLSP,ACISI)); (LEPOC,(AMICA,((POLSP,ACISI),(SALSA,ONCMY))));, которой нет в реконструкции моделью p-distance, вместо неё есть противоречащая ей клада состава ((POLSP,ACISI),(LEPPL(AMICA,(SALSA,ONCMY))));
Для дополнительной проверки филогенетических связей было произведено укоренение дерева во внешнюю группу (Latimeria chalumnae, LATCHA) (рис.4). Данное укоренение подтвердило ранее полученные клады и позволило выявить стабильность основных групп. Однако некоторые ветви иллюстрируют отличную от таксономической структуру. Данное дерево построено по цитохромам, а не по таксономии

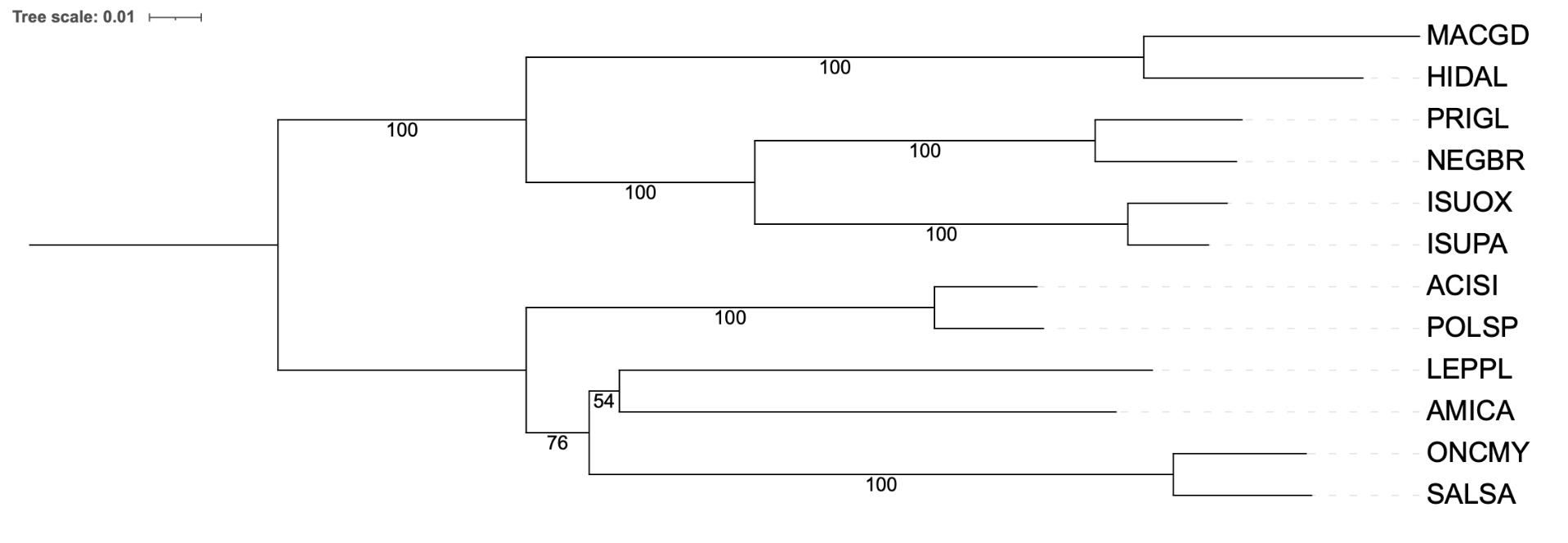
В заключительной части был проведен бутстреп-анализ (рис.5), необходимый для оценки статистической поддержки каждой ветви дерева. Большинство группировок показали высокую статистическую поддержку (100%). При этом были обнаружены ветви с умеренной поддержкой (например, кластер AMICA-LEPPL, 54%), что может указывать на меньшую надежность этих группировок и необходимость более глубокого анализа. Помимо этого, нельзя не отметить, что неправильно реконструированные ветви имеют более низкую поддержку
Бутстреп-анализ необходим для оценки надежности топологии и выполнялся при помощи программы FastME v2.1.6 (модель p-distance), и задании 100 реплик (-b 100). На полученном дереве (рис. 5) большинство ветвей оказалось поддержано на 100 процентов, что свидетельствует о высокой стабильности основных клад. При этом именно те участки, где наша реконструкция расходится с классической таксономией, оказались отмечены более низкими значениями поддержки. Это наглядно демонстрирует: «ошибочные» или менее надёжные ветви в ходе бутстрепа проявляют себя именно снижением процентной поддержки, подтверждая тем самым обоснованность такого критерия качества топологии.
Проведенный комплексный анализ показал, что реконструкция филогенетических деревьев зависит от выбранного гена и метода построения. Дерево по цитохрому B показывает общие тенденции, однако из-за вынужденной замены некоторых видов наблюдаются расхождения с таксономическим деревом. Дерево по 12S рРНК, построенное с помощью FastME (p-distance), демонстрирует высокую степень согласованности с таксономией NCBI и подтверждает важность выбора адекватной внешней группы для корректного укоренения. Бутстреп-анализ подтверждает надежность основных группировок и выявляет менее надежные ветви, которые требуют дополнительных данных или более сложных моделей эволюции для уточнения их положения.