Реконструкция и визуализация
После получения списка находок были собраны в один файл fasta-файлы соответствующих последовательностей. Затем было проведено множественное выравнивание с помощью программы muscle. Полученное выравнивание было преобразовано в формат .phy (при помощи скрипта), и на его основе построено филогенетическое дерево с использованием программы FastME, модели MtREV и 100 бутстреп-реплик. Действия повторяют схему, использованную в предыдущих практикумах.
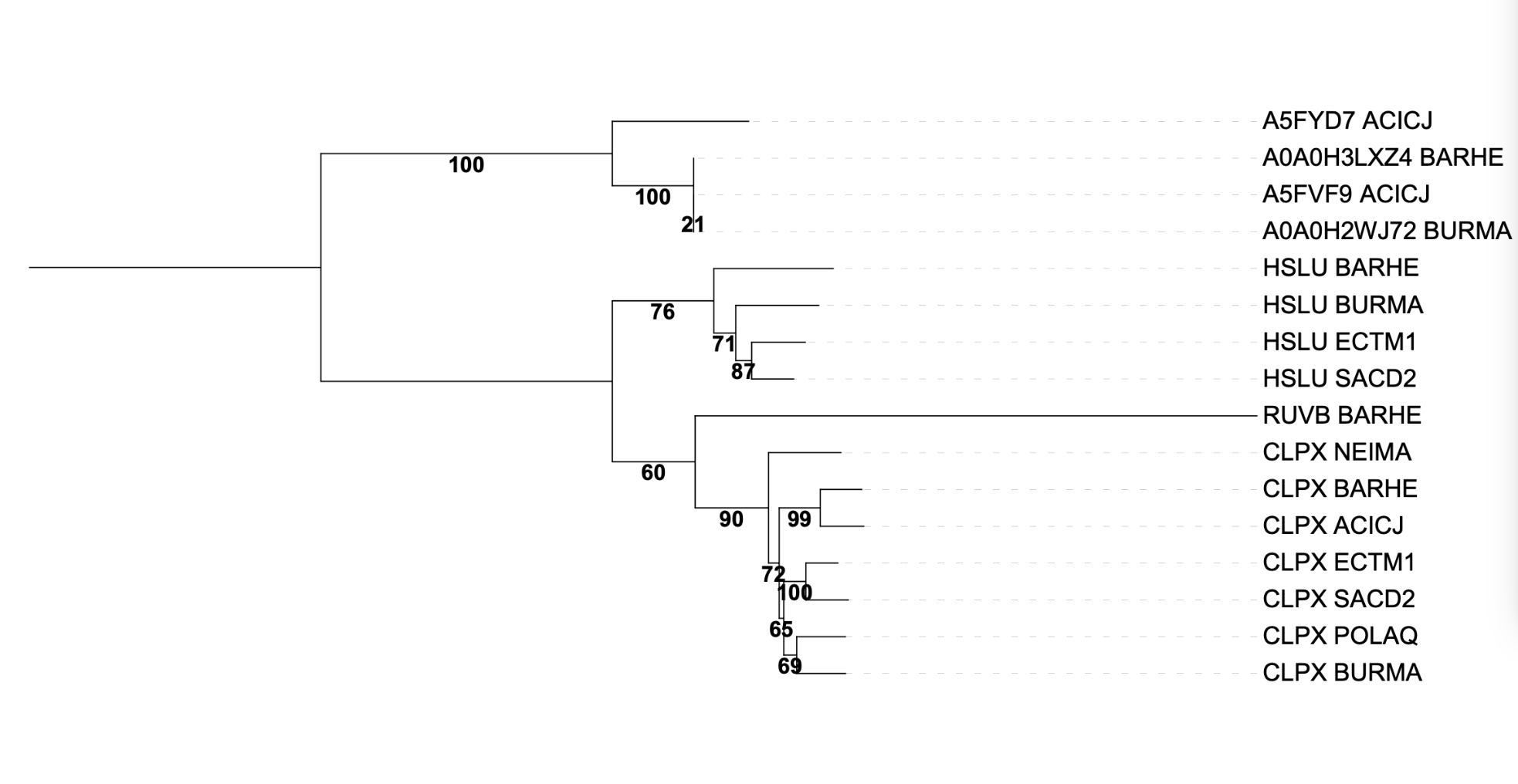
На рисунке представлено дерево, на котором разными цветами выделены ортологические группы (наборы попарно ортологических белков, в каждой группе не менее четырёх последовательностей). По результатам анализа были выделены две группы белков: группа белка CLPX и группа белка HSLU. На дереве наглядно показаны примеры как ортологичных, так и паралогичных пар.
Пары ортологов:
CLPX_BURMA и CLPX_POLAC
CLPX_ECTM1 и CLPX_SACD2
CLPX_BARHE и CLPX_ACICJ
HSLU_ECTM1 и HSLU_SACD2
Пары паралогов:
CLPX_BARHE и HSLU_BARHE
CLPX_ECTM1 и HSLU_ECTM1
CLPX_SACD2 и HSLU_SACD2
CLPX_BURMA и HSLU_BURMA
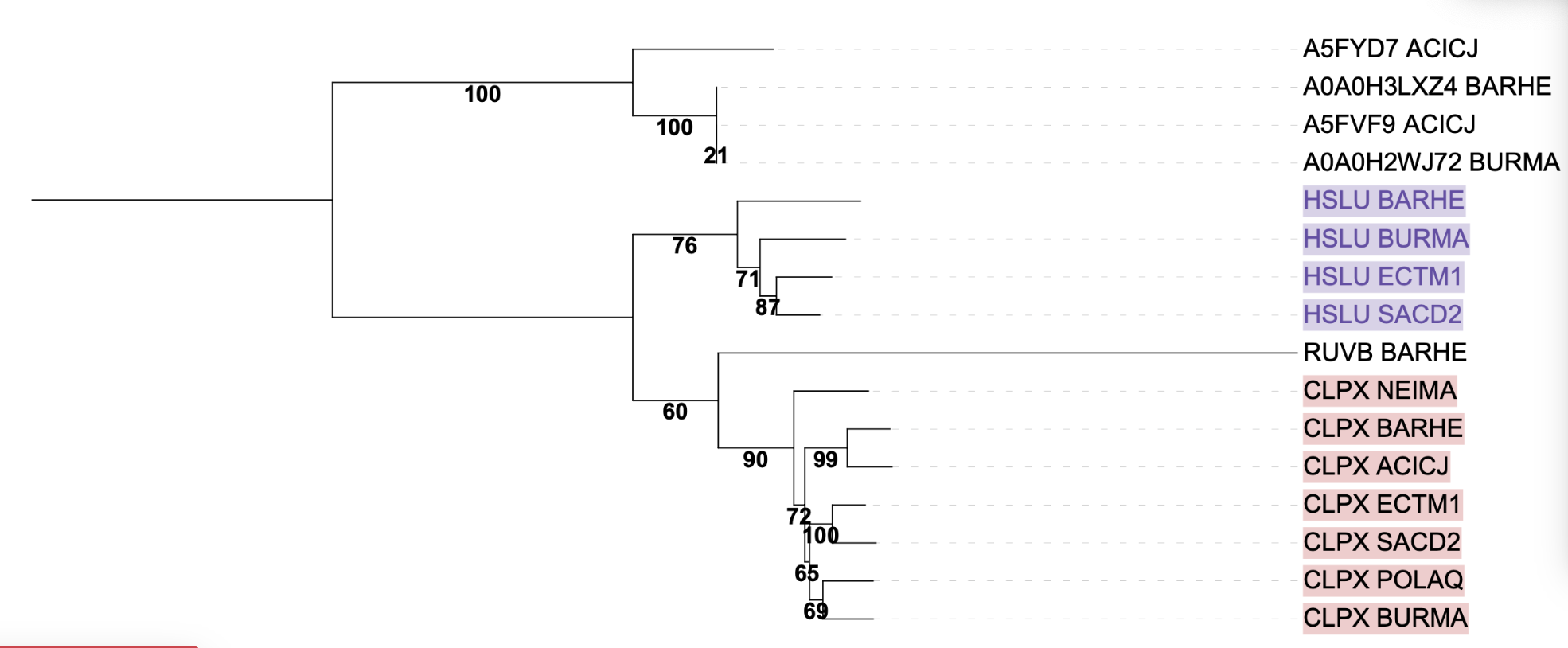
На Рис. 4 показано дерево с схлопнутыми (collapsed) ортологическими группами. Обе группы содержат последовательности большинства бактерий, что позволяет оценить соответствие реконструкции групп ортологов с филогенией бактерий (сравнение с эталонным деревом):
Утверждение о том, что обе группы (CLPX и HSLU) содержат последовательности всех 7 выбранных бактерий, является неточным. Группа CLPX действительно включает по одной последовательности от каждой из 7 бактерий, что позволяет использовать её для оценки соответствия реконструкции филогенетическим отношениям. Однако группа HSLU представлена только последовательностями от 4 бактерий (BARHE, BURMA, ECTM1, SACD2) и, следовательно, не охватывает все организмы, что ограничивает её применимость для полноценного филогенетического сравнения.
- CLPX: реконструкция данной группы не полностью соответствует филогении
- HSLU: полностью соответствует рекомендованной филогении.