
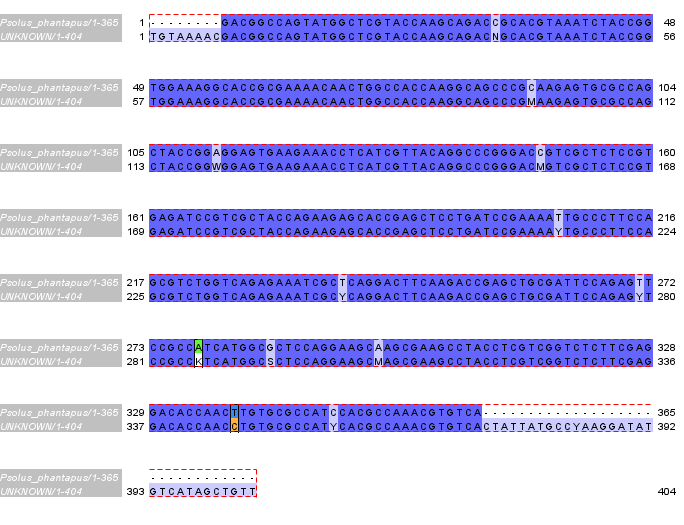
Для определения принадлежности секвенированной последовательности использовался сервис BLAST для поиска нуклеотидных последовательностей. В качестве запроса была взята собранная последовательность, поиск проводился blastn в базе Nucleotide с параметрами по умолчанию. Результат запроса доступен по ссылке. Лучшая находка принадлежит виду Psolus phantapus. Для подтверждения видовой принадлежности было сделано выравнивание находки P. phantapus и собранной последовательности. Для сравнения было взято выравнивание 2 последовательностей из рода Psolidium. Из сравнения выравниваний можно сделать вывод, что вероятнее всего секвенированная последовательность приналежит организму Psolus phantapus (полная систематика здесь). Для поиска функции не требуется дополнительного поиска, вся информация указана в записи находки P. phantapus. Запрос содержит кодирующую последовательность гистона H3, входящий в состав октомера.


Для аннотирования был выбран геном моржа (Odobenus rosmarus). Сборка имеет AC номер GCF_000321225.1. В сборке был выбран скэффолд KB229472.1. В нем был выбран контиг ANOP01043934.1. Чтобы найти в нуклеотидной последовательности участки, кодирующие белки, использовался blastx. Базой данных для поиска был взят SwissProt. Поиск таким образом осуществляется быстрее, и в находках оказываются только аннотированные белки. Остальные параметры поиска были оставлены по умолчанию, за исключением поля "Organism". BLAST проводился по всем белкам, кроме моржовых (taxid:9707). Ссылка на результат поиска.
Все находки сведетельствуют о том, что в контиге, есть участок с геном CaV2.1α1, который кодирует трансмембранный белок, α-субъединицу вольтаж-зависимого кальциевого канала. Его функции заключаются в селективном пропускании ионов Ca2+ в зависимости от мембранного потенциала клетки. Данный тип каналов располагается в мембране нейронов головного мозга, конкретнее в мембране персинаптической щели и соматодендритной мембарне, и принимает участие в регуляции секреции нейротрансмиттеров в синаптическую щель (ссылка). Возвращаясь к поиску, у находок очень маленький процент покрытия (1-2%), следовательно контиг содержит не весь ген. Для проверки был сделан поиск по соседнему из скэффолда контигу (ANOP01043933.1). Там тоже был найден небольшой участок гена CaV2.1α1 (ссылка на результат).
В задании сравнивались два генома, каждый представлен 1 кольцевой хромосомой, Bacillus shivajii (NZ_CP084703.1) и Bacillus sp. HNA3 (NZ_CP040881.1). При помощи blastn в парном выравнивании с настройками по умолчанию была посторена карта локальных свойств.
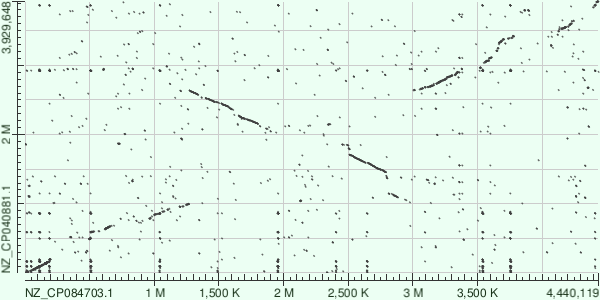
Из карты сходств следует, что геномы действительно гомологичны: много точек лежат на главных диагоналях. Точки, которые находятся на диагонали, которая выходит из начала координат, означают полное сходство участков. Точки, лежащие на второй диагонали, говорят о том, что в одном из геномов гомологичный участок инвертирован. В районе 2,8-2,9 М bp по оси x есть фрагмент, лежащий на второй диагонали и параллельный ему. По оси Y в промежутке 1,1-1,5 M bp над этим участком нет точек, следовательно либо в геном Bacillus sp. HNA3 произошла вставка длинной 400 тыс. bp, либо у Bacillus shivajii эта часть утратилась, например в процессе инверсии части генома.