
fiber -a gatc_a.pdb
А структура дуплекса в В-форме находится в файле gatc_b.pdb и получена с помощью команды
fiber -b gatc_b.pdb
| A-форма | B-форма | Файл dna25.pdb | |
| Тип спирали (правая или левая) | правая | правая | правая |
| Шаг спирали (Å) | 28.028 (T3A.C5* - A14A.C5*) | 33.752 (C32B.C5*-A22B.C5*) | 30.282 (C12A.P-G1A.P) |
| Число оснований на виток | 22 (11 пар) | 20 (10 пар) | 24 (12 пар) |
| Ширина большой бороздки | 9.627 ( A6A.P - C20B.P) | 20.584 ( A26B.P - A2A.P) | 18.543 ( G9A.P- G8A.P) |
| Ширина малой бороздки | 18.493 (A26B.P - T11A.P) | 13.199 ( A26B.P - C12A.P) | 16.485( U6A.P -C11A.P) |
Выводы: А-форма и В-форма ДНК являются правозакрученными двойными спиралями. А-форму можно визуально отличить от В-формы по следующим признакам: если посмотреть на молекулу с верху у А-формы будет хорошо заметное отверстие по середине, у В-формы оно очень маленькое. Ширина большой бороздки у А-формы меньше ширины малой бороздки. Для В-формы наблюдается обратное.
В файле dna25.pdb находится третичная структура РНК, так как в состав данной молекулы входит рибоза (а не дизоксирибоза, характерная для ДНК) и урацил (азотистое основание характерное для РНК). Также молекула состоит из только из одной цепи, а не из двух, как ДНК, хотя по внешнему виду можно решить, что это дуплекс. Данная РНК является правозакрученной двойной спиралью и по внешнему виду больше напоминает A - форму ДНК. Чило пар оснований на виток в 12, что так же подчеркивает сходство с А-формой ДНК ( у А-формы число пар оснований на виток 11-12). Однако у данной молекулы ширина большой и малой бороздок отличается всего 0.2 нм, т.е их ширина практически одинакова, а это уже не является сходством ни с А-формой и ни с В-формой.
|
Данные для gatc_a.pdb из файла gatc_a.out, найденные командой find_pair -t gatc_a.pdb stdout | analyze Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G --- 174.8 41.7 79.1 -147.7 -75.1 -157.2 |
Данная структура является очень стабильной. Это можно заметить по постоянству значений торсионных углов.Отклонения, конечно, имеются, но их мало и они не значительны. Отклонения значений торсионных углов (Strand I -delta, epsilon и zeta, Strand II - delta и epsilon) составляет 0.1 |
Данные для gatc_b.pdb из файла gatc_b.out, найденные командой find_pair -t gatc_b.pdb stdout | analyze Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 |
В-форма является менее стабильной структурой, в отличие от А-формы, так как данная структура поворочаивается относительно своей оси чаще Отклонение же торсионных углов (Strand I, Strand II - beta, gamma delta и chi) незначитально (0.1) |
Данные для dna25.pdb из файла gatc_b.out, найденные командой find_pair -t dna25.pdb stdout | analyze Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 178.8 77.3 68.2 -148.5 -76.9 -177.7 2 G -60.5 -174.6 50.4 77.1 -152.3 -77.9 -160.1 3 A -51.8 175.7 47.9 73.3 -154.6 -70.1 -167.7 4 C 162.1 -168.5 176.5 84.9 -135.9 -68.7 -172.9 5 U -68.0 178.1 57.5 72.5 -168.1 -96.6 -168.9 6 U -57.4 -175.0 45.4 79.2 -146.6 -81.9 -161.8 7 C -60.0 169.6 53.5 79.9 -149.6 -95.3 -163.2 8 G 148.2 -156.3 -169.7 97.2 -131.0 -80.1 173.9 9 G -55.3 179.2 38.1 82.6 -154.6 -77.9 -170.8 10 U -58.8 175.4 55.3 78.5 -146.0 -80.9 -164.7 11 C -50.7 165.0 51.3 75.5 -139.4 -77.2 -169.2 12 C -69.5 160.6 69.4 67.1 --- --- -164.9 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -69.5 160.6 69.4 67.1 --- --- -164.9 2 C -50.7 165.0 51.3 75.5 -139.4 -77.2 -169.2 3 U -58.8 175.4 55.3 78.5 -146.0 -80.9 -164.7 4 G -55.3 179.2 38.1 82.6 -154.6 -77.9 -170.8 5 G 148.2 -156.3 -169.7 97.2 -131.0 -80.1 173.9 6 C -60.0 169.6 53.5 79.9 -149.6 -95.3 -163.2 7 U -57.4 -175.0 45.4 79.2 -146.6 -81.9 -161.8 8 U -68.0 178.1 57.5 72.5 -168.1 -96.6 -168.9 9 C 162.1 -168.5 176.5 84.9 -135.9 -68.7 -172.9 10 A -51.8 175.7 47.9 73.3 -154.6 -70.1 -167.7 11 G -60.5 -174.6 50.4 77.1 -152.3 -77.9 -160.1 12 G --- 178.8 77.3 68.2 -148.5 -76.9 -177.7 |
Судя по значениям торсионных углов, данная структура является очень подвижной. Значения углов достаточно сильно варьируют, что так же приводит нас к выводу о том, что это молекула РНК. |
Кроме значений торсионных углов в выходном файле программы analyze содержится информация о конформационных углах, определяемых для остатков дезоксирибозы, о том, в экзо- или эндо-форме находятся эти остатки. Для каждой пары комплементарных оснований указываются длины водородных связей. Вводится система координат относительно которой определены координаты каждой пары оснований и их нормальные вектора. С помощью analyze можно не прибегая к помощи RasMol узнать форму ДНК (она определяется опять же для каждой пары нуклеотидов) и ширину бороздок, которая приведена в двух вариантах ( во втором варианте учитывается направление хода сахарофосфатного остова в пространстве.)
Стопочные структуры для А-формы ДНК из файла gatc_a.pdb созданы командами
pdb2img -c gatc_a.pdb gatc_a.ps rotate_mol -b gatc_a.pdb gatc_a2.pdb pdb2img -c gatc_a2.pdb gatc_a2.ps
| Вид сверху | Вид сбоку |
 |
 |
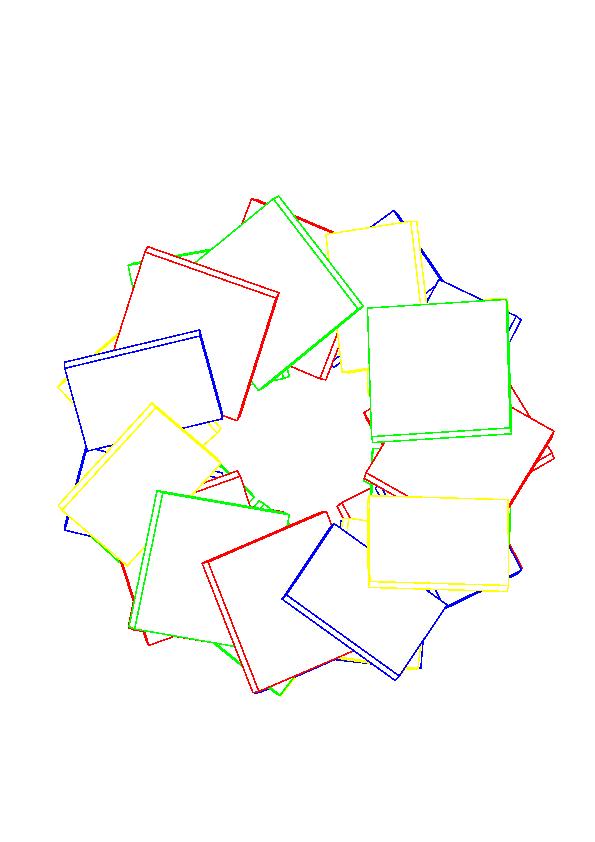
Видна характерная для А-формы ДНК полость внутри спирали. На виде сбоку заметно, что азотистые основания наклонены.
Стопочные структуры для B-формы ДНК из файла gatc_b.pdb созданы командами
pdb2img -c gatc_b.pdb gatc_b.ps rotate_mol -b gatc_a.pdb gatc_b2.pdb pdb2img -c gatc_b2.pdb gatc_b2.ps
| Вид сверху | Вид сбоку |
 |
 |
Внутри структуры отсутствует полость, что говорит о том, что это В-форма ДНК. На виде сбоку заметно, что азотистые основания находятся практически перпендикулярно к оси спирали.
Стопочные структуры для файла dna25.pdb созданы командами
pdb2img -c dna25.pdb dna25.ps rotate_mol -b dna25.pdb dna25t.pdb pdb2img -c dna25t.pdb dan25t.ps
| Вид сверху | Вид сбоку |
 |
 |
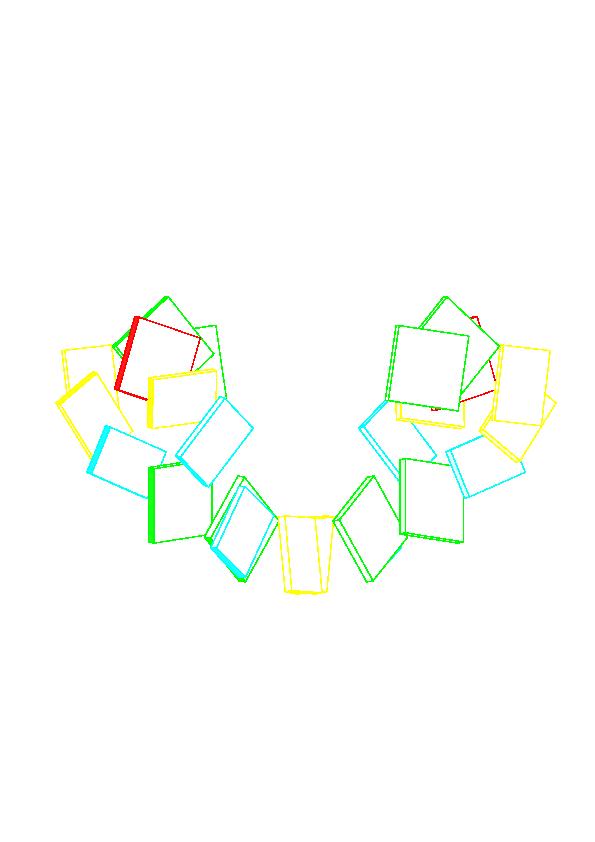
На виде сверху слабо все-таки видно, что структура имеет полость, которая придает сходство с А-формой.На виде сбоку заметно, что азотистые основания наклонены под меньшим углом к оси спирали, но при этом они не перпендикулярны ей.