Занятие 8. Структура тРНК
- В директории Term3 создана поддиректория Practice8
| PDB-код |
тРНК |
Организм |
Идентификатор цепи тРНК |
Макромолекулы в структуре |
| 1qrt |
tRNA(Gln) |
Escherichia coli |
B |
ATP,PROTEIN (GLUTAMINYL-TRNA SYNTHETASE (E.C.6.1.1.18)) |
- Последовательность тРНК из файла 1qrt.pdb:
(2) G G G G U A U C G C C A A G C G G G A A G G C A C C G G A U U C U G A U U
C C G G C A U U C C G A G G U U C C A A U C C U C G U A C C C C A G C C A (76)
Модифицированных оснований нет
* Интересно, что в данной тРНК нет мутаций, однако на сайте SRS дана последовательность тРНК не совпадающая с pdb-файлом: в 19 положении стоит U (вместо G), а в в 56 положении находится G (вместо С)
Структура 1qrt.pdb была проанализирована с помощью программы find_pair:
find_pair -t 1qrt.pdb stdout | analyze
Среди выданных программой файлов находился скрипт col_helices.scr для выделения различных alfa-спирлей структуры.
В итоге в структуре оказалось 4 спирали:
| Номер спирали |
Длина спирали |
Атомы, входящие в спираль |
| I |
6 нуклеотидных пар |
2:B, 71:B
3:B, 70:B
4:B, 69:B
5:B, 68:B
6:B, 67:B
7:B, 66:B
|
| II |
3 нуклеотидных пары |
10:B, 25:B
11:B, 24:B
12:B, 23:B
|
| III |
7 нуклеотидных пар |
37:B, 33:B
38:B, 32:B
39:B, 31:B
40:B, 30:B
41:B, 29:B
42:B, 28:B
43:B, 27:B
|
| I |
6 нуклеотидных пар |
49:B, 65:B
50:B, 64:B
51:B, 63:B
52:B, 62:B
53:B, 61:B
54:B, 58:B
|
Участки, входящие в разные спирали
2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38
G G G G U A U C G C C A A G C G G G A A G G C A C C G G A U U C U G A U U
39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75
C C G G C A U U C C G A G G U U C C A A U C C U C G U A C C C C A G C C A
Изображение цепи тРНК, созданное RasMol
На рисунке цепь тРНК в остовной модели, найденные спирали покрашены в разные цвета, при атомах фосфора 5'-концевого и 3'-концевого нуклеотидов подписаны номера остатков.
| 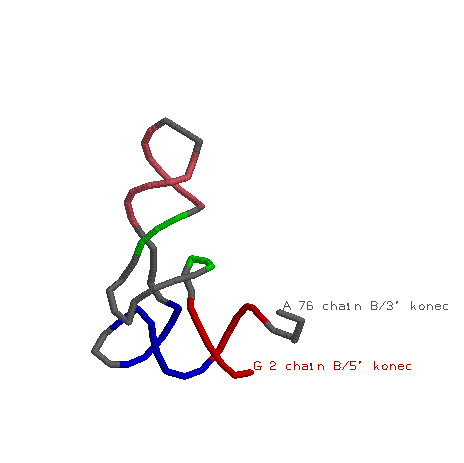
|
background white
restrict nucleic
backbone 200
wireframe off
color grey
define helix1 2-7:B,66-71:B
select helix1
color red
define helix2 10-12:B,23-25:B
select helix2
color green
define helix3 37-43:B,27-33:B
select helix3
color pink
define helix4 49-54:B,58-65:B
select helix4
color blue
select 2:B and *.P
label G 2 chain B/5' konec
select 76:B and *.P
label A 76 chain B/3' konec
|
Пример внеспирального стекинг-взаимодействия между основаниями:
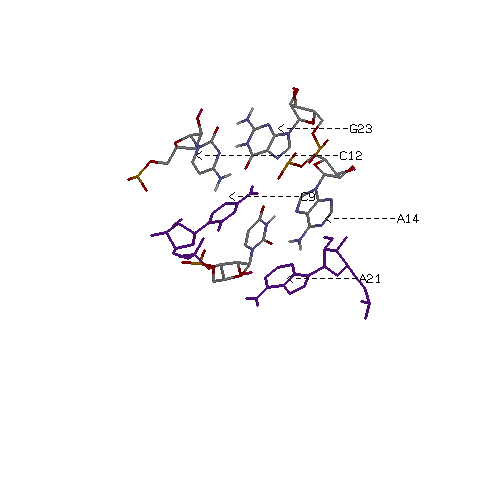
|
Внеспиральное взаимодействие между U8 и А21; между G23 и С9
А(14)-----U(8)-----A(21)
| |
| |
| |
| |
C(12)-----G(23)-----C(9)
|
Пример водородных связей между основаниями, не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований.
Данные о водородных связях между основаниями, не сводящимися к Уотсон-Криковскому спариванию комплементарных оснований из файла 1QRT.out:
Strand I Strand II Helix
13 (0.010) B:..55_:[..U]Ux**+xG[..G]:..18_:B (0.010) x
------------------------------------------------------------
15 (0.007) B:..38_:[..U]U-*---U[..U]:..32_:B (0.005) |
------------------------------------------------------------
21 (0.005) B:..44_:[..C]Cx*---A[..A]:..26_:B (0.005) |
------------------------------------------------------------
29 (0.003) B:..46_:[..U]U-**+-U[..U]:..47_:B (0.007) +
Note: This structure contains 4 non-Watson-Crick base-pairs.
В данной молекуле тРНК содержатся четыре спирали, при этом две из них состоят только из одной пары нуклеотидов. Следовательно, для выяснения вопроса: "На какую из форм ДНК похожи спирали РНК?" - буду рассматривать только первые две спирали:
| Helix I |
Helix II |
 |
 |
Из данных изображений видно, что спирали тРНК больше всего похожи на А-форму ДНК, так как на рисунках явно видно отверстие в спиралях.
| Helix |
Большая бороздка |
Малая бороздка |
| I |
11.507 (C49B.O4*-C68B.C4*) |
17.784 (U55B.C2*-U63B.O5*) |
| II |
12.349 (G30B.N7-G43B.C3*) |
17.799 (U32B.C5-G42B.O3*) |
Данные о длине большой и малой бороздки в спиралях также подтверждают, что спирали тРНК похожи на А-форму ДНК.
Программа einverted из пакета EMBOSS позволяет найти инвертированные последовательности в НК.Программа запускалась с параметром Minimum score threshold 10, так как при значениях параметра 50, 40 и 30, программа не давала никакого результата. По выданным прграммой выравниваниям можно предположить возможные двуспиральные участки в последовательности исследуемой РНК.
Результаты программы einverted:
Score 18: 6/6 (100%) matches, 0 gaps
1 ggggta 6
||||||
67 ccccat 62
Участки 1-6 и 62-67 целиком входят в спираль I, следовательно выравнивание соответствует данным полученным ранее. Однако программа einverted не нашла еще одну одну большую спираль в молекуле тРНК (двумя другими спиралями можно пренебречь, так как они состоят всего из одной нуклеотидной пары). Возможно, правильной работе данной програмы помешала сложная структура тРНК и внеспиральные стекинг-взаимодействия между основаниями. Следовательно программа einverted не может считаться универсальной, ее данные обязательно должны проверяться и уточняться.
Анализ последовательности тРНК программой mfold. Команда, использовавшаяся для работы с программой mfold:
mfold SEQ=1qrt.fasta P=15
Параметр P=15 был выбран потому, что в этом случае получается больше вариантов структур, а также потому что в выдаче присутствует структура наиболее похожая на реальную тРНК (форма клеверного листочка). При Р=10 и Р=20 получаются одни и теже структуры, которые не похожи на настоящую тРНК.
Предсказание вторичной структуры, максимально близкое к реальности:
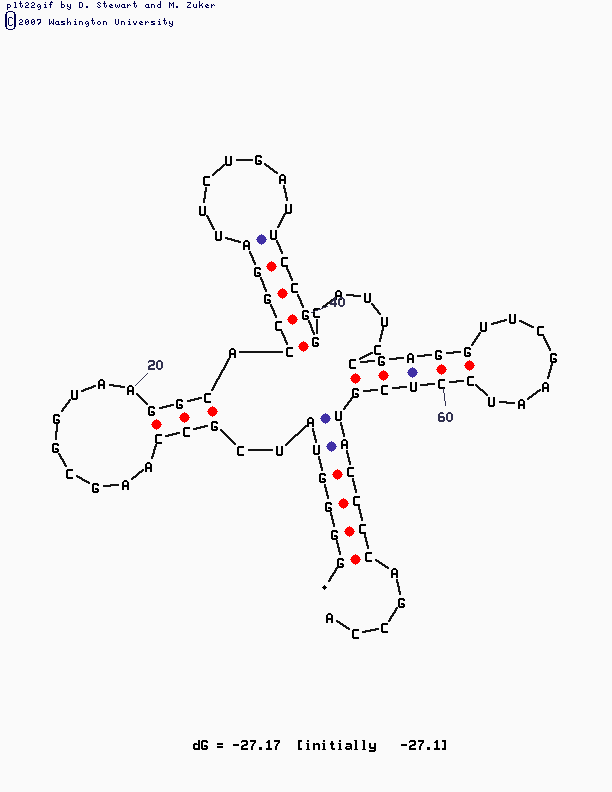
на страницу III семестра
на главную
