Страница курса биоинформатики
Факультет биоинженерии и биоинформатики
PDB-код структуры: 1YFG
тРНК: YEAST INITIATOR TRNA, цепь А
Организм: SACCHAROMYCES CEREVISIAE; BAKER'S YEAST
Последовательность тРНК:
A G C G C C G U 1MG 2MG C G C A G H2U G G A A G C G C M2G C A G G G C U C A U T6A A C C C U G A U 7MG H2U 5MC 5MC U C G G A U C G 1MA A A C C G RIA G C G G C G C U A C C A
Модифицированные основания:
1MG 1N-метилгуанозин-5`-монофосфат 2MG 2N-метилгуанозин-5`-монофосфат H2U 5,6-дигидроуридин -5`-монофосфат M2G N2-диметилгуанозин-5`-монофосфат 5MC 5-метилцитидин-5`-монофосфат 7MG 7N-метил-8-гидрогуанозин-5`-монофосфат RIA 2`-0-[(5`-фосфо)рибозил]аденозин-5`-монофосфатСтруктура тРНК была проанализирована с помощью программы find_pair
Результаты проанализированы. Найдено 4 спирали. Ниже представлена картинка, где тРНК в остовной модели раскрашена по спиралям:
Результаты программы: 1 | A:...1_:[..A]A-----U[..U]:..72_:A 0.42 2 | A:...2_:[..G]G-----C[..C]:..71_:A 0.33 3 | A:...3_:[..C]C-----G[..G]:..70_:A 0.94 4 | A:...4_:[..G]G-----C[..C]:..69_:A 0.48 5 | A:...5_:[..C]C-----G[..G]:..68_:A 0.58 6 | A:...6_:[..C]C-----G[..G]:..67_:A 0.74 7 | A:...7_:[..G]G-----C[..C]:..66_:A 0.88 8 | A:..49_:[5MC]c-----G[..G]:..65_:A 0.89 9 | A:..50_:[..U]U-----a[RIA]:..64_:A 0.47 10 | A:..51_:[..C]C-----G[..G]:..63_:A 0.24 11 | A:..52_:[..G]G-----C[..C]:..62_:A 0.23 12 | A:..53_:[..G]G-----C[..C]:..61_:A 0.56 13 | A:..54_:[..A]A-**--a[1MA]:..58_:A 4.12 14 | A:..55_:[..U]U-**+-G[..G]:..18_:A 5.20 15 | A:..57_:[..G]G-**+-A[..A]:..20_:A 4.34 16 x A:..56_:[..C]C-----G[..G]:..19_:A 1.04 17 | A:..35_:[..A]A-**--U[..U]:..33_:A 7.03 18 | A:..38_:[..A]A-*---C[..C]:..32_:A 3.79 19 | A:..39_:[..C]C-----G[..G]:..31_:A 0.93 20 | A:..40_:[..C]C-----G[..G]:..30_:A 0.50 21 | A:..41_:[..C]C-----G[..G]:..29_:A 0.90 22 | A:..42_:[..U]U-----A[..A]:..28_:A 0.55 23 | A:..43_:[..G]G-----C[..C]:..27_:A 0.58 24 | A:..44_:[..A]A-*---g[M2G]:..26_:A 1.78 25 | A:..10_:[2MG]g-----C[..C]:..25_:A 0.56 26 | A:..11_:[..C]C-----G[..G]:..24_:A 0.62 27 | A:..12_:[..G]G-----C[..C]:..23_:A 0.17 28 | A:..13_:[..C]C-----G[..G]:..22_:A 0.97 29 | A:..14_:[..A]A-**--U[..U]:...8_:A 4.79 30 | A:..15_:[..G]G-**+-c[5MC]:..48_:A 3.59
| |
При помощи Rasmol создана картинка, на которой изображена цепь тPНК в остовной модели.
background white define helix1 1-7, 66-72 define helix2 49-53, 61-65 define helix3 38-44, 26-32 define helix4 10-13, 22-25 select all wireframe off backbone 200 select helix1 color green select helix2 color cyan select helix3 color blue select helix4 color red select not (helix1, helix2,helix3,helix4) color orange select A76.p, A1.p label %r%n color black set fontsize 10 set fontstroke 2
| |
| |
Пример неспиральных стекинг взаимодействий можно наблюдать на примере взаимодействия 66-5-64 оснований цепи А.
| |
Водородные связи между основаниями, не сводящиеся в Уотсон-Криковскому взаимодействию можем наблюдать на примере взаимодействия 26 2N-метилгуанозин-5`-монофосфата (2MG) и 44 аденина; 20 аденина и 57 гуанина:
| |
Посмотрим на структуру РНК:
| |
| |
Визуально cтруктура похожа на А форму, когда мы смотрим на спираль с торца, то видим в центре отверстие.
C помощью программы einverted поищем возможные двуспиральные участки в последовательности исследуемой РНК.
Для этого создадим фаста-файл: trna.fasta, в котором все модифицированные основания мы заменили на N.
Программа выдала:
Score 35: 7/7 (100%) matches, 0 gaps
1 a g c g c c g 7
+ + + + + + +
71 t c g c g g c 65
Найден один очень маленький кусочек. В то время как при помощи find_pair найдено гораздо больше пар. Это происходит потому что einverted не учитывает неканонические взаимодействия. Получается, что тех взаимодействий, которые einverted учитывает (канонические комплементарные), очень мало, и поэтому программа выдает такой маленький кусочек. Однако, это выравнивание практически точно отражает предсказанную с помощью find_pair первую спираль
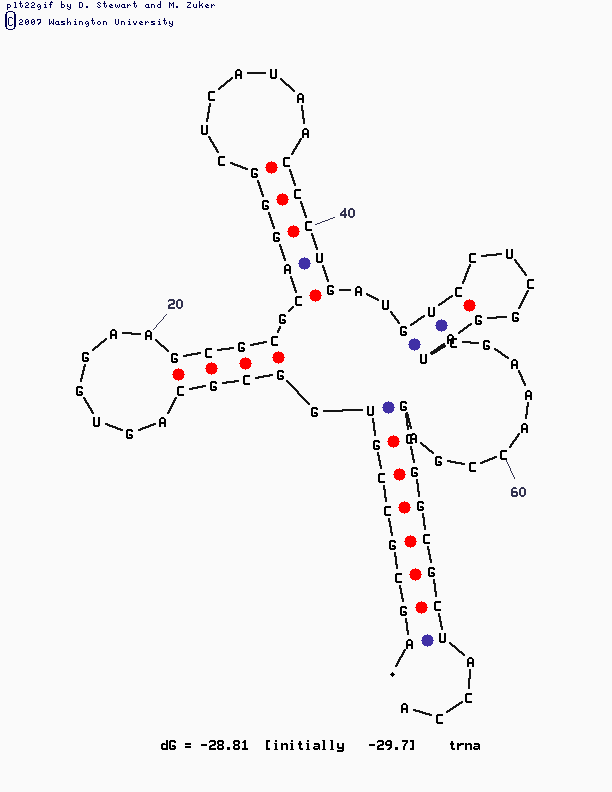
Программа m_fold была запущена несколько раз, наилучший результат тыл показан призначении параметра Р=10. Ниже приведена соответствующая картинка:
|