Мини-обзор генома бактерии Methylocystis heyeri
Автор: Лиференко Анастасия
Факультет Биоинженерии и Биоинформатики, Московский Государственный университет, Москва, Россия
Контактные данные: a.liferenko@fbb.msu.ru
Резюме: В данной работе рассматриваются стандартные данные о геноме бактерии Methylocystis heyeri, а также статистические данные о белках протеома и генах РНК данного штамма
Ключевые слова: Methylocystis heyeri, геном, протеом
Введение
Аэробные метанотрофные бактерии способны использовать метан в качестве единственного источника углерода и энергии. Они были разделены на два типа,, относящиеся к гамма- и альфапротеобактериям.
Мной выбранная бактерия- Methylocystis heyeri- типа II, обладает "фирменными" жирными кислотами метанотрофов типа I. Штаммы бактерии : H2(T), выделенный из кислого (pH 4,3) сфагнового торфяного озера (Teufelssee, Германия, 2001). Другой штамм Sakb1, выделенный из кислой (pH 4,2) почвы тропического леса (Таиланд,2005)
Описание клеток штаммов:аэробными, грамотрицательными, неподвижные палочки,покрытыми большими полисахаридными капсулами и содержащими внутрицитоплазматическую мембранную систему, типичную для метанотрофов II типа. Они обладают как частичной, так и растворимой монооксигеназой метана и используют сериновый путь усвоения углерода. Наиболее уникальной характеристикой этих штаммов является профиль жирных кислот фосфолипидов. Помимо характерной жирной кислоты метанотрофов.
Последовательности генов 16S рРНК имели 96-98% сходства с последовательностями других метанотрофов II типа в родах Methylosinus и Methylocystis. Филогения последовательности гена 16S рРНК и pmoA показала, что штаммы образуют новую линию внутри рода Methylocystis. Таким образом, предполагается, что эти два штамма представляют новый вид, Methylocystis heyeri sp. nov.
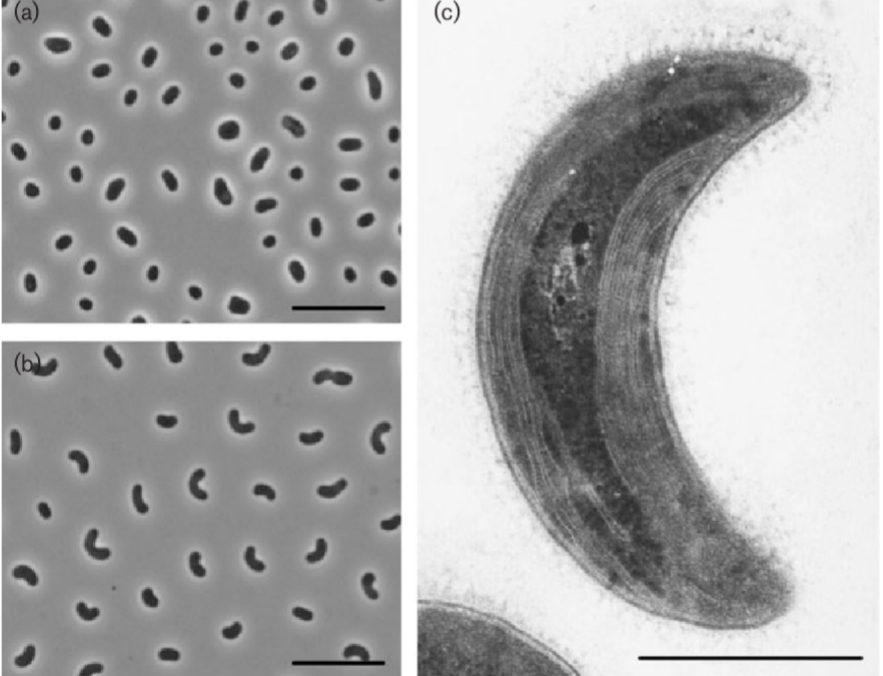
Рис.1. Фазово-контрастные микрофотографии клеток штаммов H2T (а) и Sakb1 (б), выращенных на твердой минеральной среде под метаном в течение 7 дней. (в) Электронная микрофотография ультратонкого среза клетки штамма H2T, выращенной на метане. второго типа, клетки также содержали большое количество жирной кислоты, которая ранее считалась характерной жирной кислотой метанотрофов первого типа
Материалы и методы
Для проведения вычислительных исследований из электронной библиотеки NCBI были взяты: нуклеотидная последовательность генома, аннотация протеома Methylocystis heyeri.
Вычислительные исследования на основе этих данных были проведены с помощью программы для работы с электронными таблицами Google Sheets и Python.
Количество белок-кодирующих генов, генов РНК, медианная длина белка и некоторые другие статистические данные были получены с помощью программы Google Sheets. Поиск встречаемости нуклеотидов и подсчет стоп-кодонов был реализован при помощи скриптов Python. Для поиска участка oriC и ter, использовала сервис Webskew.
Результаты
- 3.1 Стандартные данные о геноме Methylocystis heyeri
Геном Methylocystis heyeri состоит из одной кольцевой хромосомы и двух кольцевых плазмид. Название, размер, GC-состав этих молекул (Таблице 2.)
| Тип | Название | Размер(п.н.) | GC-состав(%) |
|---|---|---|---|
| Хромосома | NZ_CP046052 | 45,51947 | 63 |
| Плазмида 1 | unnamed 1 | 145,219 | 61 |
| Плазмида 2 | unnamed 2 | 30,622 | 60 |
Таблица 2. Состав и размер генома
- 3.2 Зависимость количества белковых продуктов от их длины
Чтобы получить картину распределения белковых продуктов по длинам, была составлена гистограмма (рис. 2).
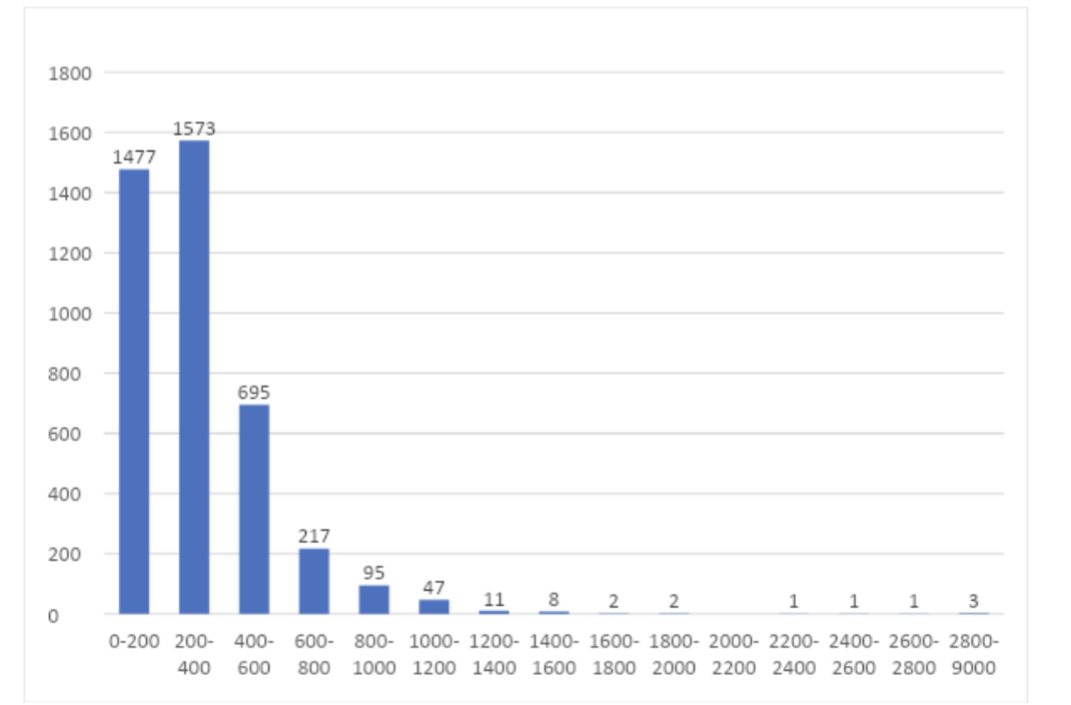
Рисунок 2 – Распределение белков по длинам аминокислотных цепей
Из этой гистограммы видно, что разброс протеома значителен, однако большая его часть приходится на белки длиной до 1400 аминокислотных остатков (а.о.). Наиболее многочисленными являются белки длиной от 200 до 400 а.о. – 1573 белка. Второе место занимают еще более мелкие белки длиной до 200 а.о. – 1477 представителей. Белков длиной более 1400 а.о. в сумме насчитывается 18 единиц, причем их длина не превышает 9000 а.о.
- 3.3. Распределение генов на прямой и обратной цепи генома
Всего у бактерии 4165 генов, кодирующих какой-либо белок. Из них на прямой(+) цепи находится 2046 генов, а на комплементарной (-) - 2119.
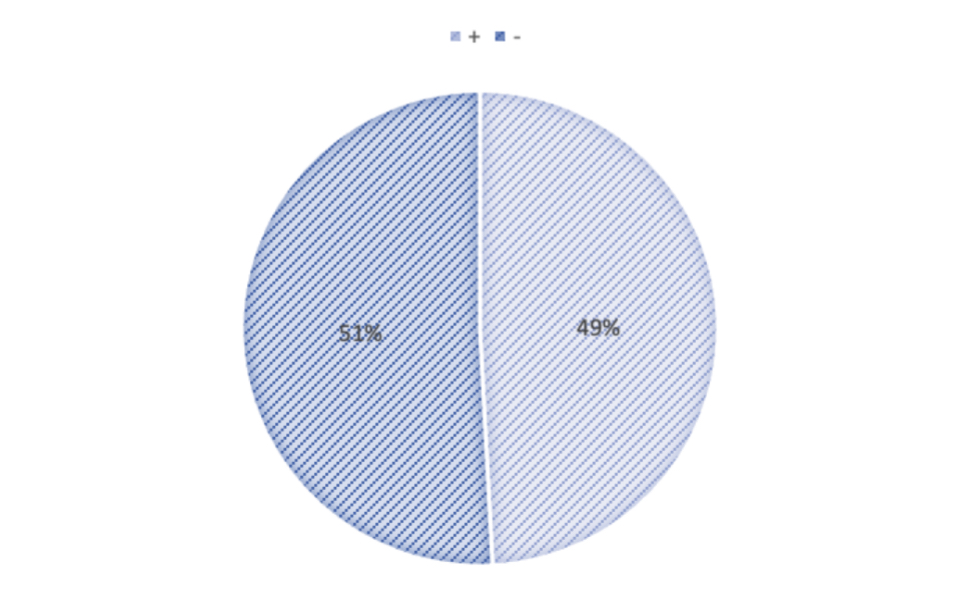
Рис 3. Распределение генов на прямой и обратной цепи генома бактерии
На рисунке выше (рис.3) представлено распределение относительно всего генома ( на цепи прямого направления находится 49.2% генов, а на обратной - оставшиеся 50.9%). Можно сказать, что гены распределены примерно поровну на обеих цепях. В следующей таблице(Таблица 3) показано более точное распределение в плазмидах и хромосоме.
| ДНК | Прямое направление цепи | Обратное направление цепи | %содержание от всех белков |
|---|---|---|---|
| Хромосома | 1965 | 2048 | 96,4% |
| Плазмида 1 | 66 | 61 | 3,05 |
| Плазмида 2 | 15 | 10 | 0,6% |
| Всего | 2046 | 2119 | 100: |
Таблица 3. Распределение генов, кодирующих белки на прямой и обратной цепи молекулы ДНК.
Для того, чтобы проверить случайность распределения на прямой и обратной цепи хромосомы и плазмиды, было рассчитано % содержание от всех белков, указанных в (Таблица 3)
- 3.4 Соотношение различных классов белков в геноме бактерии
С помощью таблицы геномных особенностей были получены данные о количестве разных классов белков в геноме Methylocystis heyeri
| класс белков | Кол-во | общая доля |
|---|---|---|
| рибосомальный | 41 | 1,9% |
| гипотетический | 822 | 19,6% |
| транспортный | 202 | 4,8% |
Таблица 4. Соотношение разных классов белков в геноме
- 3.5 Статистические данные о генах РНК
В ходе рассмотрения распределения генов различных функциональных РНК было выяснено:
| Кол-во | |
|---|---|
| Всего РНК-кодирующих генов | 62 |
| рРНК-кодирующих генов | 11 |
| тРНК-кодирующих генов | 51 |