Чтение последовательностей по Сэнгеру
Jalview проект с выравниваним прочтений прямой и обратной последовательности – тех частей, которые пригодны. Исправлены нуклеотиды,
которые программа определила неправильно – они есть в начале и в конце и написаны строчными буквами (к сожалению, может быть, плохо видно).
Полиморфизмов не найдено.
Выравнивание.jvp
Вот выравнивание с образанными концевыми участками, но неправильно определенные нуклеотиды не исправлены.
align_not_fixed.jvp
Итоговая последовательность в fasta формате. Исправленные проблемные нуклеотиды выделены строчными буквами.
final.fastaИсходные данные:
Файлы с хроматограммами прямой и обратной цепи в формате .ab1
orig_seq_f.ab1
orig_seq_r.ab1
Для просмотра и редактирования автоматического прочтения будем использовать программу Chromas (Lite).
Далее для удобства: прямая цепь == f-цепь; обратноя цепь == r-цепь.
Открываем файл с прямой последовательностью, проверяем нечитаемые участки по прямой последовательности. Открываем и разворачиваем (reverse + complement) обратную последовательность.
Пробуем выровнять цепи. Довольно легко находим соответствие: по участку "ТТТАТТТТТ" с 7 нуклеотида в f-цепи, с 56 нуклеотида в r-цепи.
Удаляем нечитаемые 5'- и 3'- концы, ориентируясь на f-цепь. И прямая и обратная последовательности будут начинаться с "ТТТАТ.." Из-за того, что мы удалили концы изменилась нумерация, зато теперь нет проблем с выравниванием.
Чтение последовательности | |
Изображение | Описание |
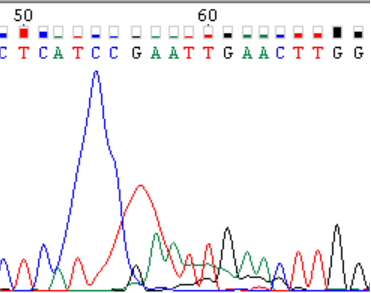 |
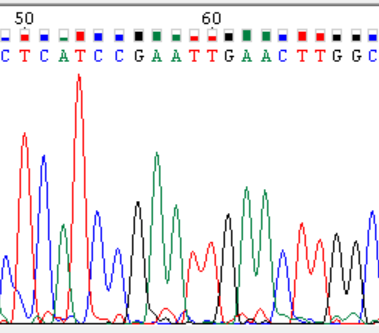
Проверяем участок 50 – 60 |
 |
|
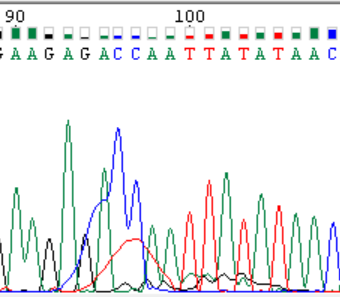 |
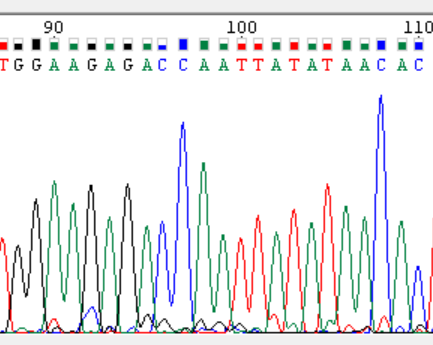
92 –100 f |
 |
Просмариваем прямую цепь. Со 105 нуклеотида уровень шума низкий, качество сигнала хорошее.
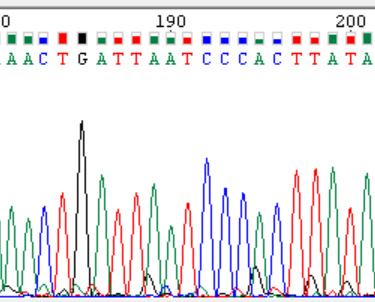 |
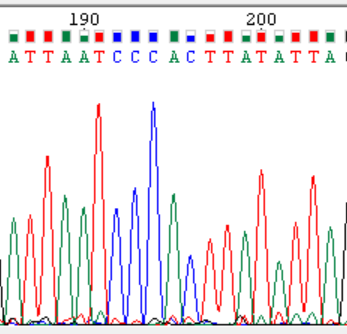
Нуклеотиды 189 и 195 |
 |
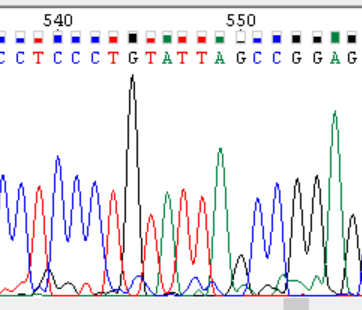 |
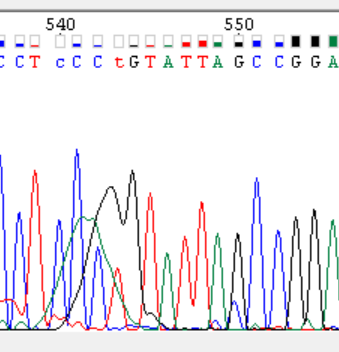
Участок 539 – 545 |
 |
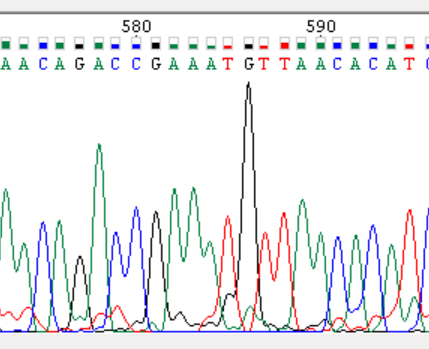 |
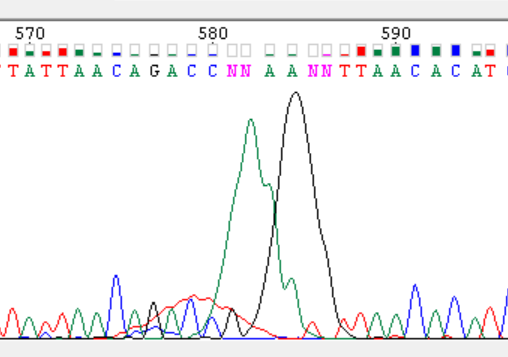
Нуклеотиды 574 – 588 проблемный участок на обратной цепи |
 |
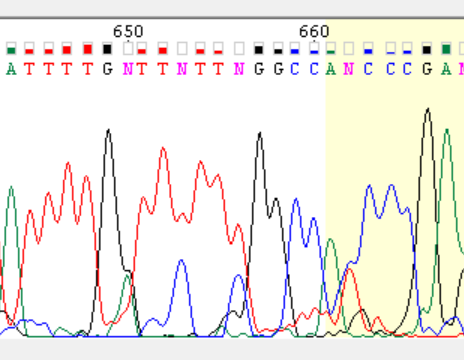 |
До 614 нуклеотида мы можем читать обе цепи. С №615 падает качество хроматограммы обратной цепи. Небольшой участок 615 – 650 можно попробовать восстановить по прямой цепи, но с №649 в прямой хроматограмме повышается уровень шума, возникают "спорные" пики (651, 654, 657, 663). И мы не можем сказать, полиморфизм это или высокий уровень шума, т.к. не можем проверить по обратной цепи. Скорее всего, это шум, потому что хроматограмма заканчивается и ее качество падает. |
Помним, что мы развернули обратную последовательность и перешли к комплементарной цепочке. \reverse+complement\
Будем считать, что мы можем восстановить цепочку до 630-го нуклеотида – обратную, до 650-ого – прямую. Далее удалим нечитаемые концы.
Последовательности с обрезанными нечитаемыми концами:
прямая
обратная
Прямая. Fasta-формат
Oбратная. Fasta-формат
Выравнивание
Пример нечитаемой хроматограммы
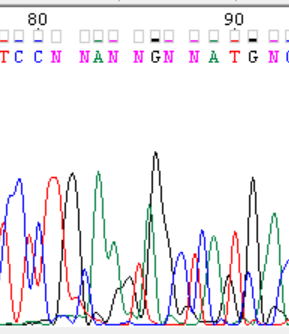
Рис.1. Высокий уровень шума. Невозможно разделить сигнал и шум, следовательно, невозможно прочитать последовательность.
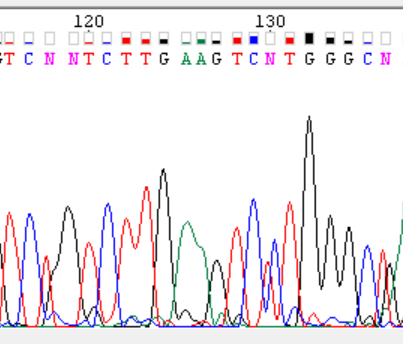
Рис.2. 118 и 119 – два пика сливаются в один
Между 129 и 130, а также между 135 и 136 пиками появляется дополнительный пик.
