Базы данных нуклеотидных послеовательностей
Качество сборки генома Серой крысы (Rattus norvegicus)
Серая крыса (англ. Norway rat) -- это не только вредитель и переносчик опасных заболеваний, но и важный модельный организм. Крысы служат экспериментальной моделью для изучения высшей нервной деятельности, психологии. Также на них изучают болезни человека, например, артрит, гипертонию, диабет, сердечно-сосудистые заболевания.
В базе данных Genome NCBI используем поиск по организму. Для Rattus norvegicus есть 7 сборок генома, 7 проектов по секвенированию организма и 7 образцов. [1]
Рассмотрим наиболее полную сборку -- первую в списке.
Информация о проекте:
Сборка The Brown Norway rat RGSC v3.1, охватывающая более 90%
генома серой крысы, предоставлена Консорциумом по секвенированию генома крысы -- Rat Genome Sequencing Consortium (RGSC).
Сборка была получена комбинированием (WGS) Whole Genome Shotgun подхода, использованного для генома мыши
и иерархического подхода (клонирования с помощью искусственных бактериальных хромосом),
использованного для генома человека.
[2]
Информация об образце:
Этот образец представляет собой комбинацию двух индивидуальных
биообразцов: SAMN02808218 и SAMN02808227.
Rnor 6.0. включает две женские особи линии BN/SsNHsdMCW
и одну мужскую особь линии SHR (также известной как SHR-Akr).
[3]
Другая информация о данной сборке собрана в таблице 1.
| Таблица 1. Информация о сборке GCA_000001895.4 [4] | |
|---|---|
| BIOSAMPLE ID [5] | SAMN02808228 |
| BIOPROJECT [6] | PRJNA10629 |
| Общая статистика [7] | |
| Общая длина последовательности | 2,870,184,193 |
| Общее количество хромосом и плазмид | 23 |
| Информация о скэффолдах | |
| Количество скэффолдов | 1,395 |
| N50 | 14,986,627 |
| L50 | 65 |
| Информация о контигах | |
| Количество контигов | 75,697 |
| N50 | 100,461 |
| L50 | 7,356 |
| Самый длинный контиг AABR07059913 | 2082903 bp |
| Самый короткий контиг AABR07072481 | 23 bp |
Информацию о самом коротком и самом длинном контиге мы получили, скачав таблицу контигов
отсюда.
Таблица контигов в формате .tsv
Таблица В виде файла Excel с сортировкой по убыванию.
В колонке "View" таблицы контигов есть ссылки на записи в GenBank, а также на последовательности контигов. Например, для контига AABR07000005:
Ключи, используемые в таблицах особенностей.
Подробное описание ключей из таблицы особенностей, которое легко найти, есть на сайте INSDC. Также там есть примеры аннотаций последовательностей. Разберем пример аннотации эукариотического гена.
- source
- Обязательный ключ, который идентифицирует биологический источник данной последовательности
- regulatory
- Любой участок последовательности, выполняющий функцию регуляции транскрипции, трансляции, репликации или структуры хроматина
- mRNA
- мРНК
- CDS
- Кодирующая последовательность -- последовательность нуклеотидов, которой соответствует последовательность аминокислот в белке.
- exon
- Участок последовательности, который кодирует часть сплайсируемой мРНК, тРНК, рРНК. Может содержать 5'UTR (нетранслируемую область), кодирующую последовательность и 3'UTR.
- intron
- Часть ДНК,которая транскрибируется, но удаляется в процессе сплайсинга.
- Другие ключи
- repeat_region
- Участок генома, содержащий повторяющиеся фрагменты
repeat_region 80..401 /rpt_type=DISPERSED /rpt_family="Alu-J"
- rep_origin
- Сайт начала репликации ДНК
source 1..2245 /organism="Escherichia coli" /plasmid="Plasmid XYZ" /strain="K12" /mol_type="genomic DNA" rep_origin 6 /direction=LEFT /note="ori"
- polyA_site
- РНК-транскрипт, к которому будут добавлены остатки аденина в ходе посттранскрипционного полиаденилирования.
source 1..985 /organism="Homo sapiens" /mol_type="mRNA" /db_xref="taxon:9606" /clone="mig-3" /cell_line="WI-38" /cell_type="fibroblast" polyA_site 983
- ncRNA
- Не кодирующий белок участок, с которого транскрибируется РНК, но не рибосомальная и не транспортная.
a non-protein-coding gene, other than ribosomal RNA and
transfer RNA, the functional molecule of which is the RNA
transcript;
gene 26533..26635 /gene="scr" /locus_tag="BSU6051_0018010" ncRNA 26533..26635 /ncRNA_class="SRP_RNA" /gene="scr" /locus_tag="BSU6051_0018010" /product="small cytoplasmic RNA, signal recognition particle-like (SRP) component Scr"
- operon
- region containing polycistronic transcript including a cluster of
genes that are under the control of the same regulatory sequences/promoter
and in the same biological pathway
Оперон -- область, содержащая полицистронный транскрипт, включающий в себя кластер
генов, которые находятся под контролем одной регуляторной последовательности / промотора
и участвуют в одном биологическом пути/процессе. Характерна для прокариот.
source 1..9430 /organism="Lactococcus sp." /strain="MG1234" /mol_type="genomic DNA" operon 160..6865 /operon="gal" regulatory 160..165 /operon="gal" /regulatory_class="minus_35_signal"
Геномные проекты.
Проект 100 000 геномов человека
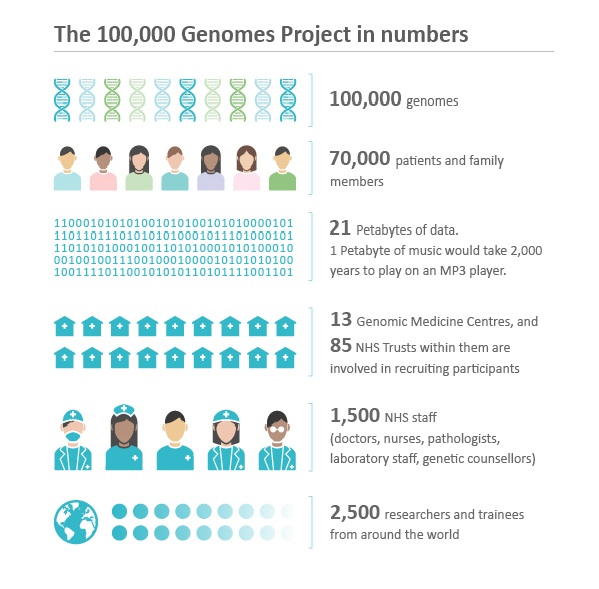
В рамках проекта 100 000 геномов, реализуемого в Великобритании, секвенируют 100 000 геномов 70 000 человек. Участники -- пациенты с редкими заболеваниями и их близкие родственники и онкологические больные.
Цель проекта -- создание нового сервиса для геномной медицины, который изменит подход к диагностике и лечению болезней. Проект также сделает возможным новые медицинские исследования. Используя данные секвенирования и данные из медицинских карт исследователи смогут изучить, как правильно использовать геномику в здравоохранении и как правильно интерпретировать данные для помощи пациентам. Будут исследованы причины, диагностика и лечение болезней. В настоящее время данный проект -- самый масштабный проект такого рода
Проект был запущен в конце 2012 года.
Дата окончания -- конец 2017 года.
На данный момент секвенировано 16171 геномов.
Цифру в 100 000 геномов выбрали, основываясь на опыте предыдущих исследований, например программы UK10K . Стоимость секвенирования снижается, а объем знаний о редких аллелях растет. Было решено, что 100 000 геномов обеспечат баланс между ценой и пользой для пациентов, чтобы создать клиническое и исследовательское наследие.
Будут секвенированы геномы 70 000 людей.У 25 000 онкобольных для сравнения будут секвенированы геномы здоровых и больных клеток, т.е. всего 50 000 геномов. На каждого из 15 000 пациентов с редким заболеванием будет приходиться по 3 генома -- собственно геном пациента и геномы двух его близких кровных родственников.
Участники проекта получают возможность узнать свой диагноз и получить необходимое специальное лечение.
Другую информацию о проекте можно узнать на сайте проекта.
Таблица митохондриальных генов
Нужно составить таблицу митохондриальных генов одного из организмов отдела
Rhodophyta [1] .
Я выбрала организм Rhodymenia pseudopalmata [2] .
Для того чтобы найти все полные митохондриальные геномы таксона, составила запрос:
(complete mitochondrion genome) AND Rhodophyta[Organism] [3]
Найдена 71 запись в GenBank и 47 RefSeq – всего 118 записей.
Переходим по ссылке "полный митохондрильный геном Rhodymenia pseudopalmata "[4].
Для получения списка митохондриальных генов я перешла по ссылке Gene в разделе Related information. Cортировала список по порядку генов в геноме (Sort by Chromosome).
Чтобы получить гены, кодирующие белки можно перейти по ссылке Protein [5].
Найдено 24 белка. На сайте можно скачать таблицу. Привели данную таблицу к требуемому виду.
Список белков
