Нуклеотидный BLAST
Определить таксономию и функцию прочтенной нуклеотидной последовательности
Запустили нуклеотидный BLAST с алгоритмом blastn (Somewhat similar sequences) по базе данных Nucleotide collection (nr/nt) с параметрами по-умолчанию. Результат работы blastn можно увидеть на рисунке ниже.
Данная последовательность является митохондриальным геном, кодирующим белок субъединицы 1 цитохром С оксидазы (CO1)
Цитохром с-оксидаза (цитохромоксидаза) или цитохром с-кислород-оксидоредуктаза, также известная как цитохром aa3 и комплекс IV — терминальная оксидаза аэробной дыхательной цепи переноса электронов, которая катализирует перенос электронов с цитохрома с на кислород с образованием воды. Цитохромоксидаза присутствует во внутренней мембране митохондрий всех эукариот, где её принято называть комплекс IV, а также в клеточной мембране многих аэробных бактерий.[1]
Организм, которому принадлежит этот ген, относится к роду Polycirrus
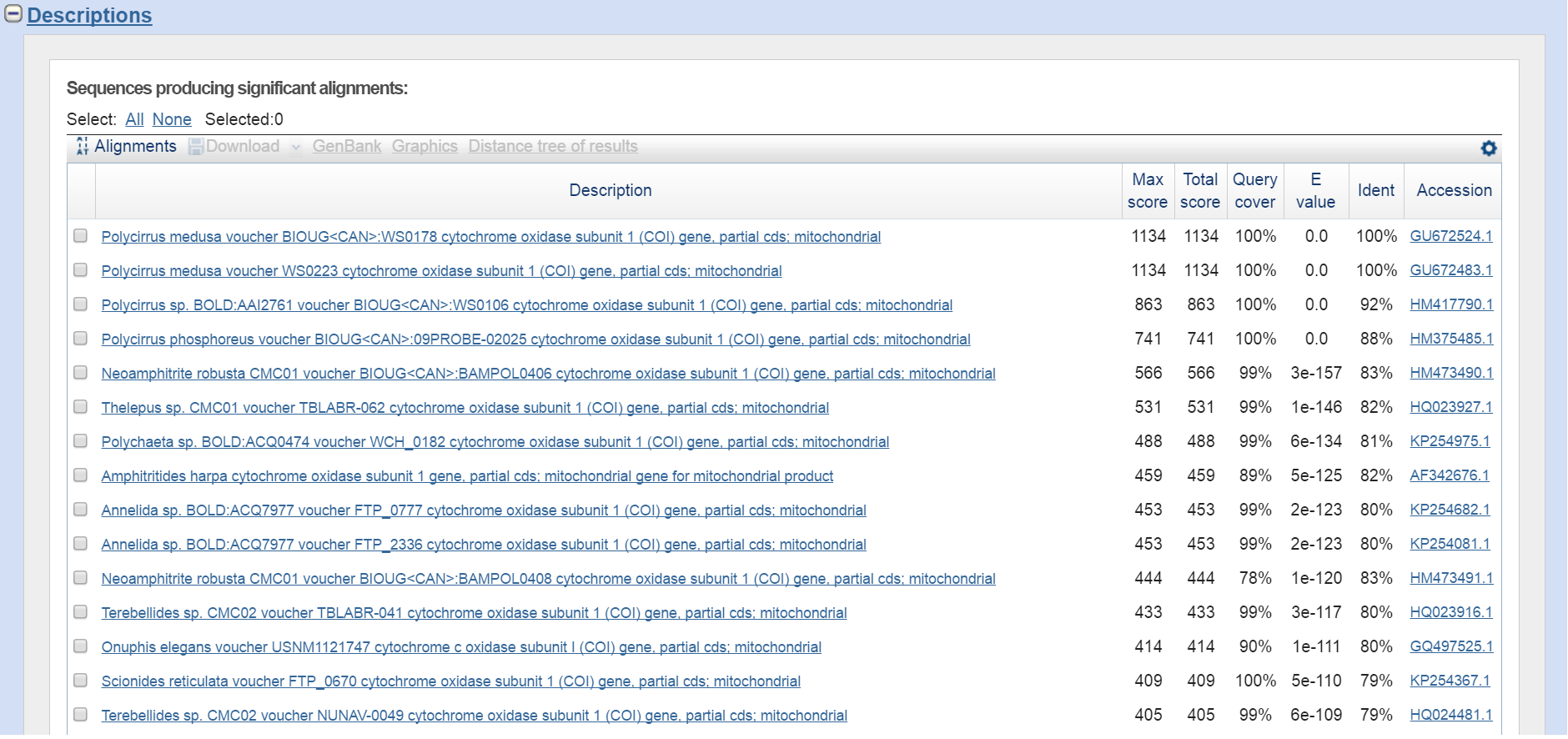
Первые 2 находки имеют следующие характеристики: E-value = 0, Identity = 100%, Query cover = 100%, Score = 1134; у них одинаковые характеристики, наверное, это две одинакове последовательности. Далее идут находки с меньшим покрытием, идентичностью и весом.
Сравнить списки находок нуклеотидной последовательности 3-я разными алгоритмами blast
Ограничили поиск blast внутри рода лучшей находки - Polycirrus. Ограничение на количество находок 5000 последовательностей.
blastn
Aлгоритм ищет все возможные похожие последовательности. Подходит для поиска информации о последовательности, о которой мы не знаем ничего. Всего было 34 находки. 27 из них были последовательности, кодирующие cytochrome oxidase subunit 1 (COI), а 7 - рибосомальную РНК (несмотря на то, что процент идентичности там 100%) программа нашла кусочек последовательности длиной 11 и выровняла его с исходной. Эти семь "последовательностей" далее ни в каких списках находок не присутствуют. Лучшая находка имеетс e-value 0.0. E-value худшей находки равен 2.5.

Pис.2. Выдача алгоритма blastn
discontiguous megablast
Всего было найдено 27 последоватлеьностей. Все они были в предыдущей выдаче. Теперь e-value наихудшей находки стал 2e-147. Наилучшая осталась прежней. Этот алгоритм отсекает неинформативные выравнивания и оставляет наиболее значимые.
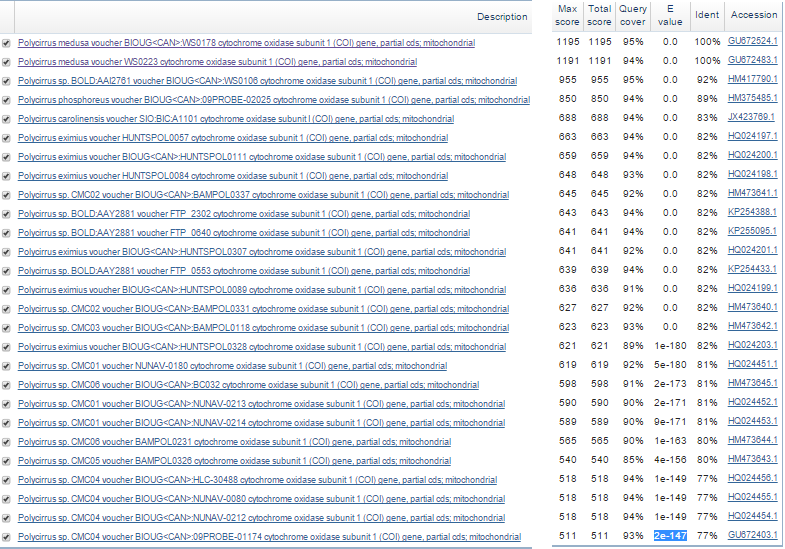
Pис.3. Выдача алгоритма discontiguous megablast
Megablast
Megablast oставил первые четыре находки. E-value "наихудшей" стал 0.0. Были отобраны самые близкие гомологи среди последовательностей. На этом этапе можно отследить отдельные нуклеотидные замены у представителей различных видов или одного вида.
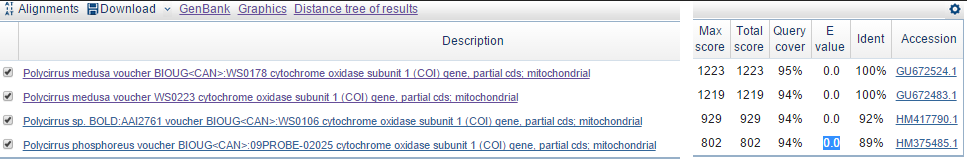
Pис.4. Выдача алгоритма megablast
Нужно выбирать алгоритм исходя из преследуемой цели. Megablast использууется для поиска очень близких гомологов. Discontiguous megablast позволяет наити просто бликие гомологи. Blastn служит для поиска похожих, не обязательно родственных последовательностей, т.е. нужно учитывать, что найденные последовательности могут быть негомологичны.
Megablast отсеивает большее количество находок и, следовательно, выдает только последовательности наиболее близкие к исходной. Он подходит для поиска близкородственных последовательностей.Работает достаточно быстро. Размер "затравки" (слова, инициирующего выравнивание) по умолчанию 28 bp и не может быть ниже 16bp.
Blastn работает медленнее, т.к. у него затравка 7 - 15bp.Проверить наличие гомологов трех белков в геноме организма Amoboaphelidium protococarum
Выполняли с помощью локального BLAST на kodomo.
- Подготовили локальную базу данных (makeblastdb -in X5.fasta -dbtype nucl)
- Для каждого из выбранных белков запустили по данной базе алгоритм tblastn, находящий гомологи белка в формальной трансляции нуклеотидного банка (tblastn -query xxx.fasta -db X5.fasta > xxx.out).
- HSP7C_HUMAN -- консервативный шаперон HSP70, белок теплового шока. Имеется у большинства организмов из всех царств. Играет роль репрессора активации транскрипции. Является компонентом PRP19-CDC5L комплекса, формирующего интегральную часть сплайсосомы, и необходим для активации сплайсинга пре-мРНК. Связывая бактериальные ЛПС (липополисахариды), является посредником в процессах ЛПС-индуцированного воспаления, включая секрецию моноцитами TNF (фактора некроза опухоли).
tblastn -query HSP7C_HUMAN.fasta -db X5.fasta > HSP7C_HUMAN.out
Результат записан в файл HSP7C_HUMAN.out. Лучшая находка имеет вес в битах 917 и E-value = 0.0.
> scaffold-199 Length=1112851 Score = 917 bits (2369), Expect = 0.0, Method: Compositional matrix adjust. Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%) Frame = -2Хороший E-value, достаточно высокие проценты Positives и Identities позволяют говорить, что у исследуемого организма есть гомолог данного белка.
-
CISY_HUMAN — (цитратсинтаза) — фермент, катализирующий реакцию конденсации ацетата (ацетил-CoA) и оксалоацетата, в результате чего образуется цитрат. Синтетаза цитрата обнаружена практически во всех клетках аэробных организмов, катализируемая реакция является лимитирующей на первом этапе цикла трикарбоновых кислот. Фермент располагается в митохондриальном матриксе эукариот, однако кодируется ядерным геномом. Синтез осуществляется на рибосомах цитоплазмы, а затем синтаза цитрата транспортируется в матрикс митохондрии5. Можно предположить, что такой белок должен быть у всех аэробных организмов.[1]
tblastn -query CISY_HUMAN.fasta -db X5.fasta > CISY_HUMAN.out
Результат записан в файл CISY_HUMAN.out
Четыре находки. Лучшая имеет следующие параметры:
> scaffold-693 Length=1268102 Score = 565 bits (1457), Expect = 2e-180, Method: Compositional matrix adjust. Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%) Frame = +1Хорошие параметры сходства: e-value = 2(5)e-180; identities:69% из них positives:84% -- позволяют предположить, что у исследуемого организма действительно имеется гомолог цитратсинтазы.
-
TERT_HUMAN TERT_HUMAN - теломераза, восстанавливающая длину хромосомы при репликации. Имеется у большинства, но не у всех эукариот. Активна в прогениторных и раковых клетках, в нормальных же соматических не активна или проявляет очень низкую активность. Играет важную роль в процессах старения и предотвращении апоптоза.
tblastn -query TERT_HUMAN.fasta -db X5.fasta > TERT_HUMAN.out
Результат записан в файл TERT_HUMAN.out
Лучшая имеет следующие параметры:
> scaffold-17 Length=2125590 Score = 105 bits (263), Expect = 8e-23, Method: Compositional matrix adjust. Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%) Frame = +1BLAST выдал три находки, однако даже у самой лучшей из них параметры сходства слишком низкие. Гомология доменов маловероятна, т.к. совпадения равномерно распределены по всей длине последовательности. Вывод: у Amoboaphelidium protococarum нет гомолога данного белка.
Найти один ген белка, закодированный в одном скэффолде "Amoboaphelidium"
-
Полоучили информацию о длинах скэффолдов, чтобы выбрать скэффолд подходящей длины.
infoseq X5.fasta -only -name -length > info.txt Выбрали один скэффолд подходящей длины
В файле info.txt представлена информация о длинах скэффолдлов. Выбираем один размером около 20 000 пар нуклеотидов -- unplaced-665.
seqret X5.fasta:unplaced-665 -out unplaced-665.fastaПоследовательность скэффолда сохранена в файле unplaced-665.fasta
- Запускаем blastn с параметрами по умолчанию. Результат на рисунке 2.
Рис.2. Выдача blastn
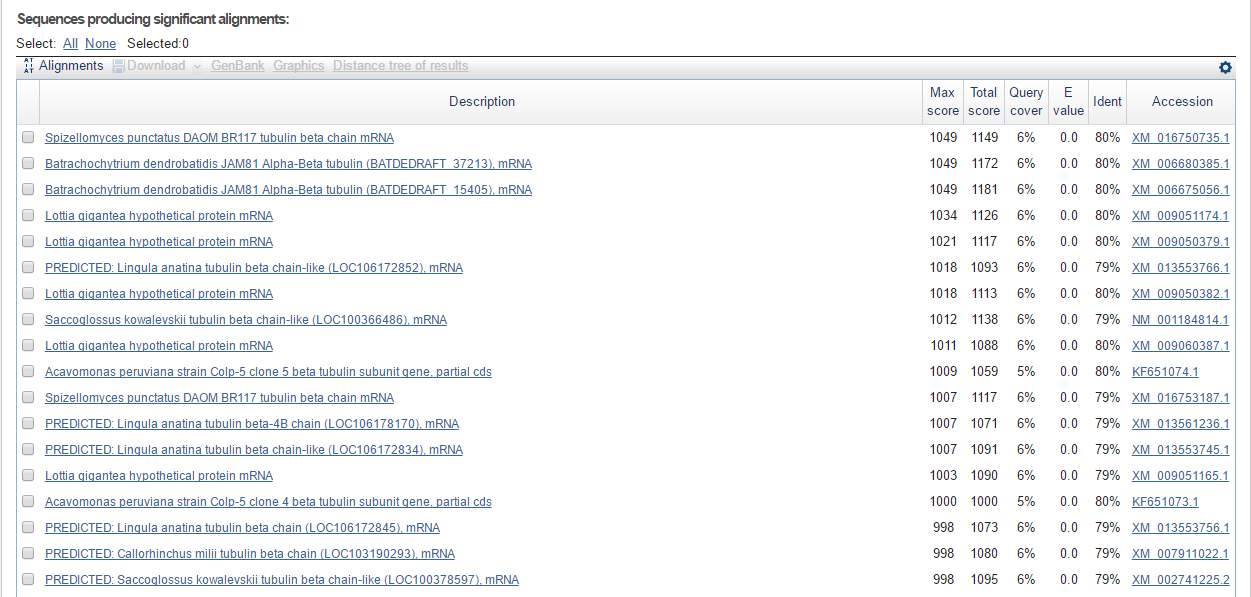
Как можно видеть, все лучшие находки, для которых показано, какой белок они кодируют, кодируют составные части белка тубулина. Покрытие для лучших находок всего 6 %, так что исследуемый контиг содержит ген субъединицы тубулина.
