Предсказание генов прокариот
Информация о плазмиде
- ID – CP005089
- Организм – Sphingobium sp. Таксономия
- Размер – 53908 пар нуклеотидов
1. Работа с EMBOSS
C помощью EMBOSS скачали последовательность
плазмиды cp005089 в форматах gff и fasta.
seqret embl:cp005089 plasmid.fasta
Файл plasmid.fasta содержит полную нуклеотидную последовательность плазмиды.
embl:cp005089 -feature plasmid.gff
В файле plasmid.gff сохранены особенности последовательности, и оттуда
с помощью скрипта CDS_only.py можно извлечь интересующую нас информацию о координатах кодирующих белки последовательностей.
CDS_plasmid.gff
2. Предсказание Prodigal
Программа Prodigal используется для предсказания генов прокариот.
Она запускается из командной строки.
<'PATH'>prodigal.exe -i plasmid.fasta -o plasmid_prod.gff -f gff
-i – файл, который подается программе на вход -o – выходной файл программы -f – формат выходного файла – мы выбрали формат gff.
3. Обработка результатов предсказания
Теперь мы хотим получить информацию о количестве правильно и неправильно предсказанных генов. Для этого используем скрипты на Python.
Подготавливаем файлы с помощью скрипта CDS_only.py
cds_plasmid.gff
cds_prod.gff
Аннотировано 76 генов, предсказано 80.
Check_prediction_equal.py – базовый скрипт.
Получает на вход файлы cds_plasmid и cds_prod и имя выходного файла
записывает файл
Получилось 55 генов. Список совпадающих генов: eq.out
Для подсчета генов, у которых правильно предсказан только N- или только C-конец, используем модификации базового скрипта.
Учитываем ориентацию гена. На комплементарной цепи меньшая координата – С-конец гена, большая – N-конец.
Check_prediction_equal_start.py – ищет гены у которых неправильно предсказан С-конец и правильно предсказан N-конец.
программа выдает 0 – таких генов нет
Check_prediction_equal_stop.py – ищет гены у которых неправильно предсказан N-конец и правильно предсказан C-конец.
11 генов 5 на прямой и 6 на комплементарной. Список генов: eqstop.out
Количество генов, которые предсказаны неправильно, вычисляем с помощью простой арифметики:
80 - 55 - 11 = 14 генов предсказано неверно.
| Качество предсказания генов | ||
|---|---|---|
| Всего | аннотиовано | 76 |
| предсказано Prodigal | 80 | |
| Совпадает оба конца | число | 55 |
| % | 68.7% | |
| Совпадает только N-конец | число | 0 |
| % | 0 | |
| Совпадает только С-конец | число | 11 |
| % | 13.7% | |
| Полное несовпадение | число | 14 |
| % | 17.5% | |
4. Причины несовпадения
Посмотрим в геномном браузере несколько генов для которых не совпала аннотация одного из концов. Заметим, что нет генов, для которых Prodigal правильно предсказал N-конец, но ошибся в предсказании С-конца. Для 11 генов Prodigal верно предсказал С-конец, но ошибся в выборе стартового кодона. Посмотрим список этих генов.
Рассмотрим ген с координатами 10614..10823 в геномном браузере.
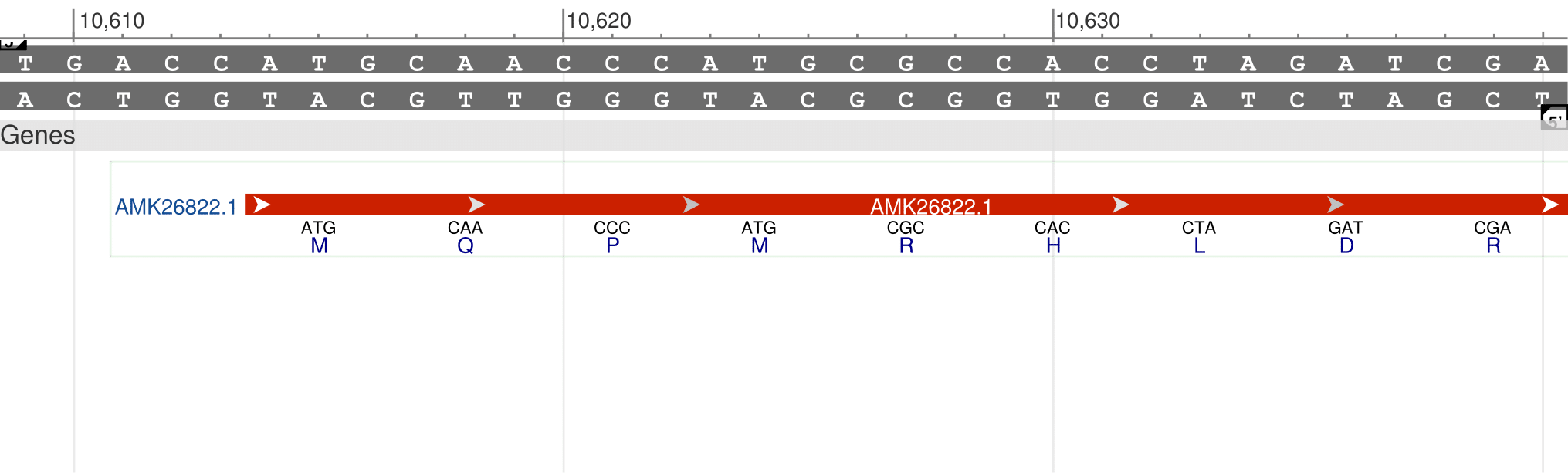
Prodigal неверно предсказал стартвый кодон этого гена и предсказанный ген немного короче аннотированного. Как мы видим на картинке рядом находятся два возможных стартовых кодона ATG (10614..10616 и 10623..10625). Это привело к ошибке программы.
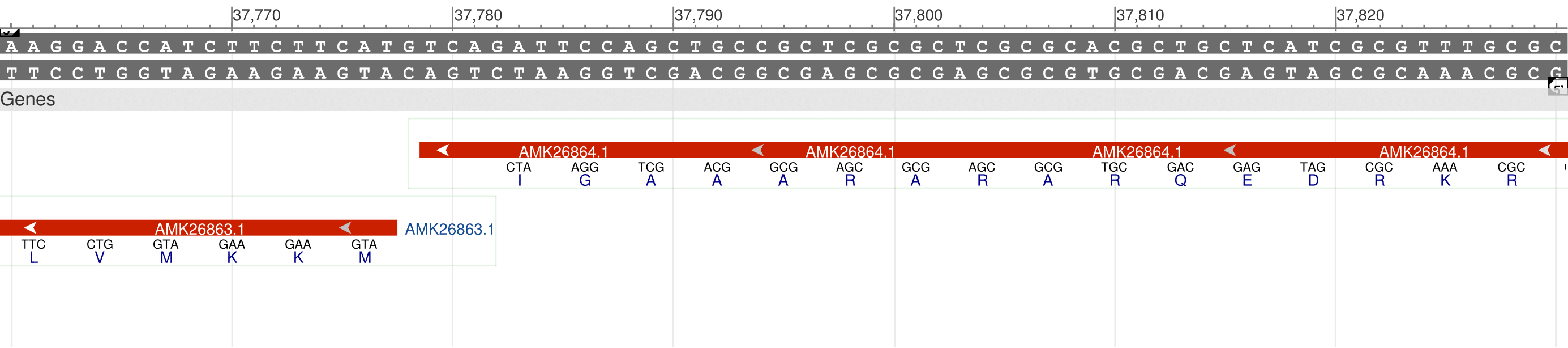
Еще один пример: 37777..37472. Также ошибка в выборе стартового кодона. В позиции 37819..37817 стоит последовательность ATG – потенциальный стартовый кодон. Но на самом деле координата N-конца гена – 37777
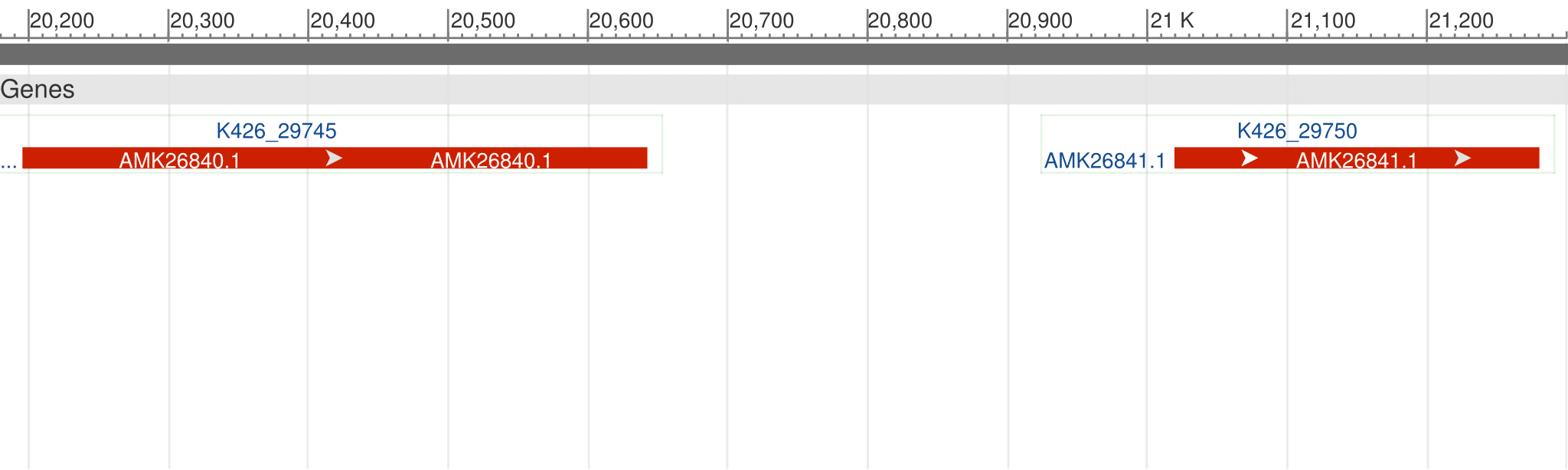
Вообще неправильно предсказанный ген (20724..20993) на комплементарной цепи. Аннотированы гены (20196..20642) и (21020..21280), причем эти гены правильно предсказаны Prodigal.
Посмотрим последовательность:
Это последовательность прямой цепи, а ген предсказан на комплементарной. Это означает, что стартовый кодон GTG, а стоп-кодон ТАG. Стоп-кодон точно не противоречит таблице генетического кода.
Насчет стартового кодона информация с сайта NCBI:
"Tаблица 11 используется для Бактерий, Архей, прокариотических вирусов и хлоропластных белков. Наиболее эффективна инициация по стартовому кодону AUG. Для Архей и Бактерий были задокументированы стартовые кодоны GUG и UUG. В E. coli, UUG служит инициатором для приблизительно 3% белков, а CUG известен как инициатор одного закодированного в плазмиде белка (RepA). В дополнение к инициации через NUG, в редких случаях у Бактерий может наблюдаться инициация трансляции кодоном AUU, как, например, в случае poly(A) полимеразы PcnB и гена InfC, кодирующего фактор инициации трансляции IF3."
[1]
Т.е. формально начало и конец гена могут быть такими, но на самом деле, ген с данной последовательностью, с данными координатами не существует.
Гены могут быть неправильно предсказаны из-за того, что есть несколько вариантов старт-кодов и стоп-кодонов. Последоватьльность всего из трех букв может много раз встретиться в длинной последовательности плазмиды (хромосомы). Гены пересекаются, т.е. даже если последовательность, потенциально стартовый кодон, попадает в какой-то ген, все равно с нее может начинаться другой ген. Т.е. правильно предсказать гены прокариот не так просто. 70% предказаний, совпадающих с аннотацией – это довольно неплохо.
