Предсказание генов эукариот
* Не нашла гены, для которых предсказан альтернативный сплайсинг в выданном контиге – беру резервный.
Информация о контиге NW_003724208
Контиг NW_003724208 принадлежит организму Vitis vinifera . Такое красивое латинское название имеет хорошо знакомый всем культурный виноград. wiki

Таксономия: Цветковое растение класса двудольные, семейства виноградовые
| Информация о контиге | |
|---|---|
| Размер | 324868 пар оснований |
| Число генов | 39 |
| Число белок-кодирующих последовательностей | 40 |
С двух генов транскрибируется некодирующая РНК. Для двух генов предсказан альтернативный сплайсинг.
Для гена с координатами 93,647..97,868 предсказан альтернативный сплайсинг.
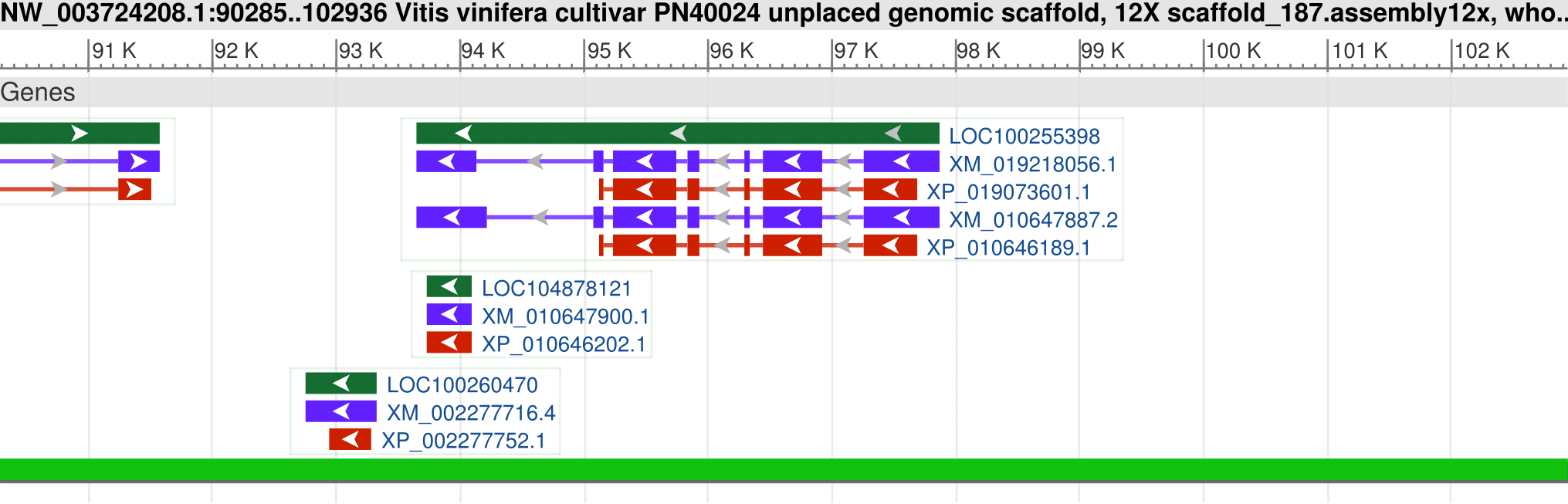
На рисунке представлено геномное окружение этого гена, различные варианты транскрибируемых с него мРНК и соответсвующие им изоформы белков. Для данного гена возможно 2 альтернативных варианта мРНК (синие полосы). Данным мРНК соответсвуют 2 изоформы белка (красные полосы).
Предсказание генов
Используем Web-сервис AUGUSTUS .
Виноград это растение. класс двудольные AUGUSTUS предлагает 4 растения: Arabidopsis thaliana Solaneum lycopersicum, Triticum/wheat, Zea mays Пшеница и кукуруза это однодольные растения, т.е. Резуховидка Таля и томат ближе по систематике. Я выбрала резуховидку.
Предсказание. Выбрали организм. Загрузили fasta файл с последовательностью контига. Выбрали параметры: предсказать гены на обеих цепях, не предсказывать 3'UTR (нетранслируемую область), Предсказать любое число, возможно, неполных генов (альтернативы: предказывать только полные гены, предсказывать только полные гены и предсказать по крайней мере один, предсказать ровно один ген. Не предсказывать альтернативные транскрипты. Не игнорировать конфликты с другой цепью.
Программа возвращает архив с 6 файлами.
| Расширение файла | Содержание фала |
|---|---|
| .aa | белковые последовательности предсказанных генов в fasta формате |
| .cdsexons | нуклеотидные последовательности экзонов в fasta формате |
| .codingseq | последовательности кодирующих участков ДНК в fasta формате |
| .gbrouse | трек-файл предсказания для геномного браузера |
| .gff | информация о предсказанных генах в формате gff (Gene Feature Format) |
| .gft | информация о предсказаных генах в формате gft |
Посмотрим файл с расширением gff, т.к. у нас есть скрипты для оценки пресказания с преддыдущего практикума.
Обработаем файл augustus.gff скриптами
CDS_only.py
Gene_only.py
Check_prediction_equal.py
Check_prediction_equal_start.py
Check_prediction_equal_stop.py
Аugustus предсказывает 69 генов. Посмотрим их границы.
1 полностью правильно предсказанный ген
4 гена с правильно предсказанным С-концом
Что-то не впечатляет.
Попробуем посмотреть CDS:
168 CDS в аннотации
243 в предсказании
Получилось 138 правильно предсказанных CDS
Попробуем еще раз, только теперь для образца выберем томат.
Повторим все действия для файла augustus_tomato.gff
Аugustus предсказывает 70 генов.
0 полностью правильно предсказанных генов
2 c правильно предсказанным N-концом
4 гена с правильно предсказанным С-концом
Попробуем посмотреть CDS:
168 CDS в аннотации
222 в предсказании
Получилось 124 правильно предсказанных CDS
AUGUSTUS довольно плохо предсказывает гены. Он предсказывает больше белок ккодирующих последовательностей, чем есть на самом деле, но предсказывае их довольно качественно. Как интерпретировать этот результат, не очень понятно.
При выборе параметров предсказания я указывала "не предсказывать альтернативные транскрипты", т.к. в этом контиге всего два таких гена. Запустим AUGUSTUS еще раз.
Я взяла немного другой fasta файл – с участком 92000..100000. Там 3 гена, для одного из которых предсказан альтернативный сплайсинг.
Вот файл gff, который выдал AUGUSTUS. Предсказано 3 гена. Для третьего предсказано много CDS – это экзоны, а альтернативный сплайсинг не предсказан. Не понятно, правильно ли предсказаны границы генов, т.к. сбилась нумерация. Но нас ведь интересовало, предскажет ли программа альтернативный сплайсинг.
Нелегкое это дело, предсказывать гены эукариот(((
