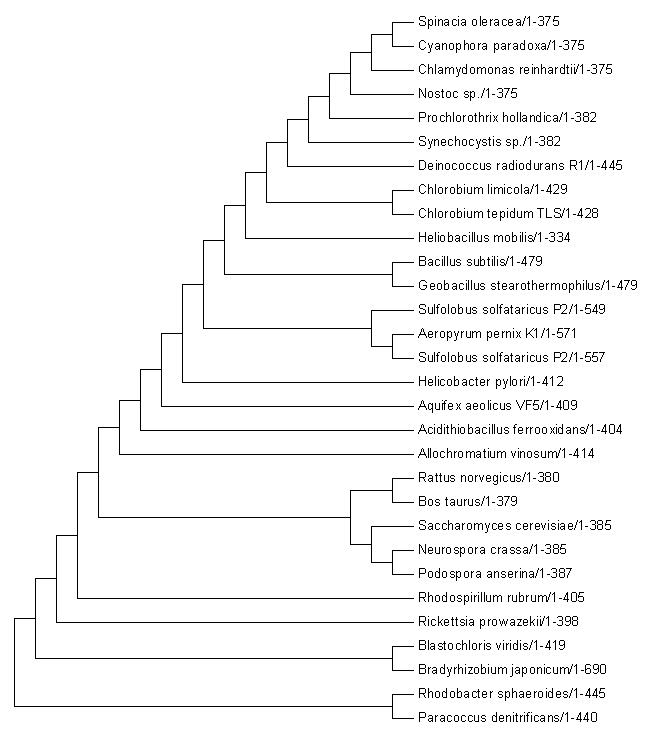
Филогенетические деревья
Проверка дерева цитохромов b
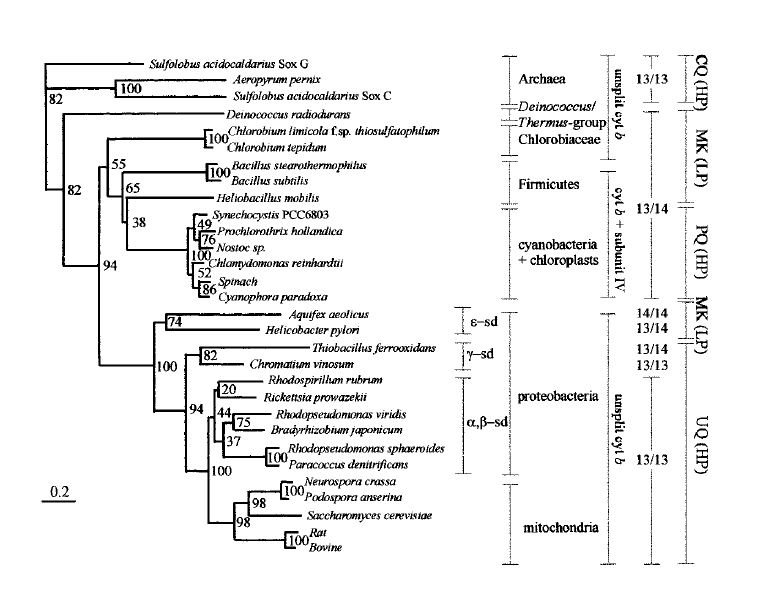
Мы повторили работу, проделанную в 2000 году Michael Schütz и коллегами. [1]
Для построения филогенетического дерева авторы использовали метод Neighbour-joining и провели Бутстрэп анализ с 5000 репликами. Дерево укореняли, используя внешнюю группу Archaebacteria
Рис.1 Дерево из статьи
Мы получили последовательности белков, которые использовали авторы из файла. Построили выравнивание в Jalview программой Mussle. С помощью программы MEGA реконструировали дерево тремя методами.
Рис.2 Дерево, построенное методом Neighbour-joining. Проведен Bootstrap-анализ с 5000 репликами. На рисунке представлено бутстрэп-консенсусное дерево.
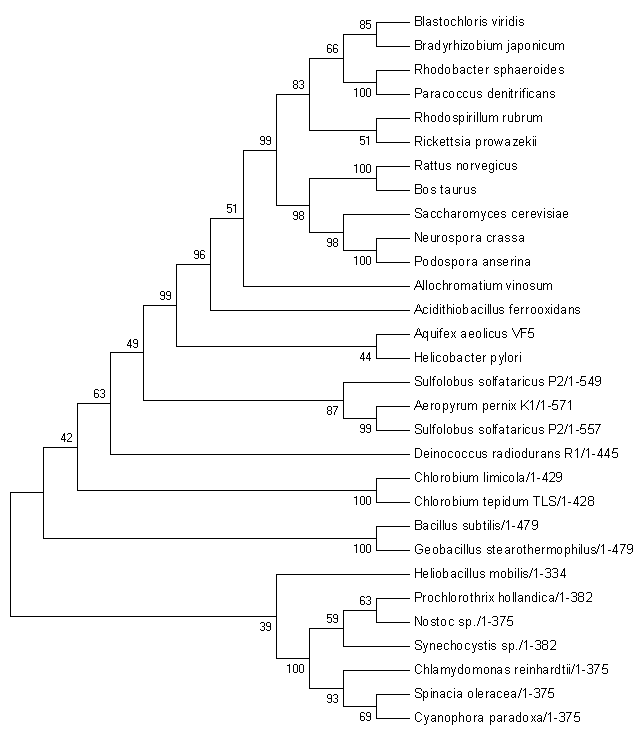
Рис.3 Дерево, построенное методом UPGMA. Консенсусное дерево бутстрэп-анализа с 5000 репликами.
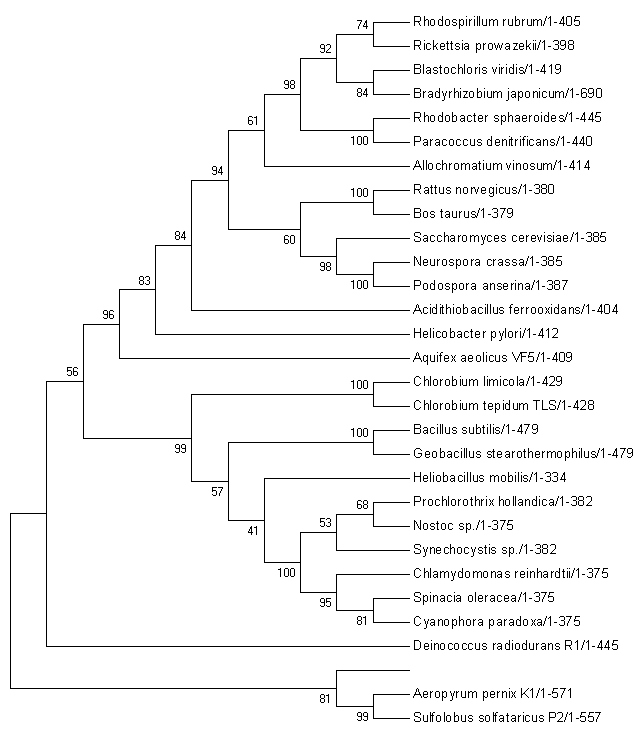
Рис.4 Дерево, построенное методом максимальной экономии (Maximum Parsimony) без бутстрэп-анализа.