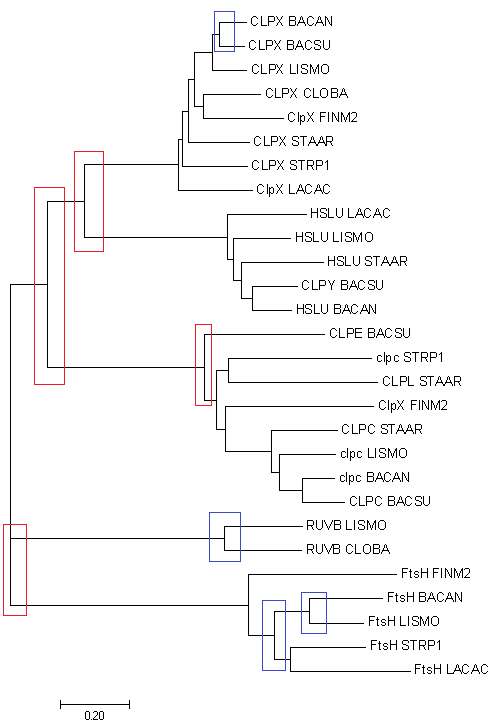
Филогенетические деревья
Построение дерева по нуклеотидным последовательностям
Получение последовательностей 16S rRNA
Мы получили последовательности 16S рибосомальной РНК каждой из бактерий из базы полных геномов NCBI (ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/). Мы находили каталог, соответствующий данному виду бактерий. Обычно находилось несколько каталогов, они соответсвуют разным штаммам. На штаммы мы не обращали внимания. В каталоге находится несколько файлов. Нас интересует файл с расширением .frn --- именно в нем находятся нуклеотидные последовательности РНК в fasta-формате. Находим одну из последовательностей, в описании которой есть 16S ribosomal RNA. 8 последовательностей 16S рРНК записали в файл bact_16s_rrna.fasta.
Выравнивание последовательностей и построение дерева
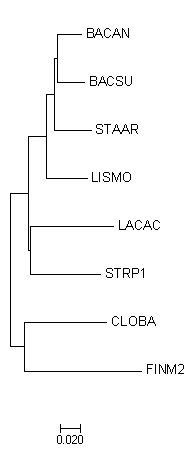
В Jalview выровняли последовательности программой Mussle. Загрузили выравнивание в MEGA, построили дерево методом минимальной эволюции. См. рисунок 1. Дерево укоренено в ветвь {CLOBA, FINM2} против {LACAC, STRP1, BACAN, BACSU, LISMO, STAAR}.
Рис.1. Филогенетическое дерево бактерий, построенное на основе последовательностей 16S rRNA методом минимальной эволюции.
Сравним построенное дерево с правильным из практикума 1.
Рис.2. Эталонное дерево

Как видно, деревья похожи по топологии. На обоих есть следующие нетривиальные ветви:
- {CLOBA, FINM2} против {LACAC, STRP1, BACAN, BACSU, LISMO, STAAR}
- {LACAC, STRP1} против {CLOBA, FINM2, BACAN, BACSU, LISMO, STAAR}
- {BACAN, BACSU} против {LISMO, STAAR, CLOBA, FINM2, LACAC, STRP1}
- {BACAN, BACSU, LISMO, STAAR} против {CLOBA, FINM2, LACAC, STRP1}
Отличие между деревьями заключается в том, что на правильном дереве есть ветвь {BACAN, BACSU, LISMO} против {STAAR, CLOBA, FINM2, LACAC, STRP1}, в то время как на реконструированном по последовательностям 16S рРНК дереве эта ветвь заменена на {BACAN, BACSU, STAAR} против {LISMO, CLOBA, FINM2, LACAC, STRP1}.
Сравним данное дерево с деревом, реконструированным по последовательностям белков. См. рис.3
Рис. 3. Дерево, реконструированное по белковым последовательностям Пептидил-тРНК гидролаз методом Neighbor Joining Using % Identity в Jalview.
Это дерево сильнее отличается по топологии от правильного. На нем есть ветви {BACAN, BACSU, LISMO} против {STAAR, CLOBA, FINM2, LACAC, STRP1} и {BACAN, BACSU, LISMO, STAAR} против {CLOBA, FINM2, LACAC, STRP1}, но отсутствует ветвь {LACAC, STRP1} против {CLOBA, FINM2, BACAN, BACSU, LISMO, STAAR}. Бактерия LACAC объединена с CLOBA и FINM2; STRP1 и {BACAN, BACSU, LISMO, STAAR} образуют одну ветвь.
Можно сделать вывод, что последовательности рРНК позволяют реконструировать деревья точнее, чем последовательности белков.
Построение и анализ дерева, содержащего паралоги
- Нашли в своих бактериях достоверные гомологи белка CLPX_BACSU.
* CLPX_BACSU -- это АТФ связывающая субъединица ClpX АТФ-зависимой CLP протеазы (ATP-dependent Clp protease ATP-binding subunit ClpX)Для поиска гомологов мы использовали установленный не kodomo blastp.
- Скопировали протеомы выбранных бактерий (см. практикум 1) в один файл bacs_prot.fasta
- Создали локальную базу данных из файла с протеомами бактерий.
makeblastdb -in bacs_prot.fasta -out db_bacs -dbtype prot - Запустили поиск
blastp -query query_bact.fasta -db db_bacs -out bacs_data.fasta -evalue 0.001 - Находки записали в файл bacs_data.fasta
Таким образом, было найденно 38 последовательностей. Информация о них собрана в таблице 1.
Таблица 1. Список находок blastp
Мнемоника Продукт E Value CLPX_BACSU ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 CLPX_BACAN ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 CLPX_LISMO ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 CLPX_STAAR ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 CLPX_CLOBA ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 B0S2N5_FINM2 ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 Q5FKR6_LACAC ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 J7M389_STRP1 ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 CLPX_STRP1 ATP-dependent Clp protease ATP-binding subunit ClpX 0.0 HSLU_LACAC ATP-dependent protease ATPase subunit HslU 1e-24 CLPY_BACSU ATP-dependent protease ATPase subunit ClpY 4e-23 HSLU_BACAN ATP-dependent protease ATPase subunit HslU 2e-22 HSLU_LISMO ATP-dependent protease ATPase subunit HslU 2e-22 HSLU_STAAR ATP-dependent protease ATPase subunit HslU 3e-22 Q81VV9_BACAN ATP-dependent Clp protease ATP-binding subunit ClpC 5e-10 CLPC_BACSU Negative regulator of genetic competence ClpC/MecB 9e-09 CLPL_STAAR ATP-dependent Clp protease ATP-binding subunit ClpL 2e-08 CLPE_BACSU ATP-dependent Clp protease ATP-binding subunit ClpE 3e-08 CLPC_STAAR ATP-dependent Clp protease ATP-binding subunit ClpC 6e-08 Q8Y8B1_LISMO ATP-dependent protease 2e-07 Q8YAB6_LISMO Endopeptidase Clp ATP-binding chain C 2e-07 B0S3X9_FINM2 ATP-dependent protease Clp ATP-binding subunit ClpC 4e-07 B0S0E3_FINM2 ATP-dependent zinc metalloprotease FtsH 6e-07 Q5FM98_LACAC ATPase 9e-06 J7MBF9_STRP1 Negative regulator of genetic competence 2e-05 Q99XR9_STRP1 Putative endopeptidase Clp ATP-binding chain C 2e-05 Q5FHW6_LACAC ATP-dependent protease 2e-05 J7M6I1_STRP1 ATP-dependent zinc metalloprotease FtsH 3e-05 Q9A200_STRP1 ATP-dependent zinc metalloprotease FtsH 3e-05 B0S3J0_FINM2 Chaperone protein ClpB 3e-05 RUVB_CLOBA Holliday junction ATP-dependent DNA helicase RuvB 1e-04 Y1421_STAAR Uncharacterized protein SAR1421 2e-04 Q81VX5_BACAN ATP-dependent zinc metalloprotease FtsH 4e-04 Q8YAC6_LISMO ATP-dependent zinc metalloprotease FtsH 4e-04 RUVB_LISMO Holliday junction ATP-dependent DNA helicase RuvB 5e-04 Q5FMA3_LACAC ATP-dependent zinc metalloprotease FtsH 7e-04 Q5FLA7_LACAC ATP-dependent Clp protease, ATP-binding subunit 7e-04 B0S222_FINM2 ATP-dependent zinc metalloprotease FtsH 7e-04
Мы удалили идентичные находки -- одинаковые последовательности, различающиеся идентификаторами. Также чтобы немного облегчить себе задачу, мы удалили белки с неточно указанной функцией и для некоторых белков придумали свои мнемоники, по которым нам будет понятна их функция. Таким образом мы взяли 28 последовательностей гомологов. Данные о них приведены в таблице 2.
Таблица 2. Отобранные гомологи
Мнемоника Продукт Мнемоника для дерева CLPX_BACSU ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_BACSU CLPX_BACAN ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_BACAN CLPX_LISMO ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_LISMO CLPX_STAAR ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_STAAR CLPX_CLOBA ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_CLOBA B0S2N5_FINM2 ATP-dependent Clp protease ATP-binding subunit ClpX clpx_FINM2 Q5FKR6_LACAC ATP-dependent Clp protease ATP-binding subunit ClpX clpx_LACAC CLPX_STRP1 ATP-dependent Clp protease ATP-binding subunit ClpX CLPX_STRP1 HSLU_LACAC ATP-dependent protease ATPase subunit HslU HSLU_LACAC CLPY_BACSU ATP-dependent protease ATPase subunit ClpY CLPY_BACSU HSLU_BACAN ATP-dependent protease ATPase subunit HslU HSLU_BACAN HSLU_LISMO ATP-dependent protease ATPase subunit HslU HSLU_LISMO HSLU_STAAR ATP-dependent protease ATPase subunit HslU HSLU_STAAR Q81VV9_BACAN ATP-dependent Clp protease ATP-binding subunit ClpC clpc_BACAN CLPC_BACSU Negative regulator of genetic competence ClpC/MecB CLPC_BACSU CLPL_STAAR ATP-dependent Clp protease ATP-binding subunit ClpL CLPL_STAAR CLPE_BACSU ATP-dependent Clp protease ATP-binding subunit ClpE CLPE_BACSU CLPC_STAAR ATP-dependent Clp protease ATP-binding subunit ClpC CLPC_STAAR Q8YAB6_LISMO Endopeptidase Clp ATP-binding chain C clpc_LISMO B0S3X9_FINM2 ATP-dependent protease Clp ATP-binding subunit ClpC clpc_FINM2 B0S0E3_FINM2 ATP-dependent zinc metalloprotease FtsH FtsH_FINM2 Q99XR9_STRP1 Putative endopeptidase Clp ATP-binding chain C clpc_STRP1 J7M6I1_STRP1 ATP-dependent zinc metalloprotease FtsH FtsH_STRP1 RUVB_CLOBA Holliday junction ATP-dependent DNA helicase RuvB RUVB_CLOBA Q81VX5_BACAN ATP-dependent zinc metalloprotease FtsH FtsH_BACAN Q8YAC6_LISMO ATP-dependent zinc metalloprotease FtsH FtsH_LISMO RUVB_LISMO Holliday junction ATP-dependent DNA helicase RuvB RUVB_LISMO Q5FMA3_LACAC ATP-dependent zinc metalloprotease FtsH FtsH_LACAC
- В Jalview получили последовательности (fetch sequences) из Uniprot, затем сделали множественное выравнивание программой Mussle.
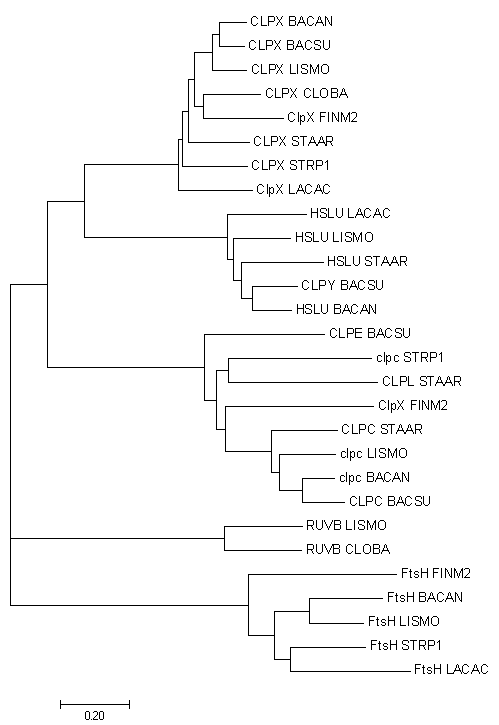
- Реконструировали филогенетическое дерево в программе MEGA методом Neighbor Joining. См. Рис.1
Рисунок 1. Реконструированное филогенетическое дерево
Два гомологичных белка называются ортологами, если они из разных организмов и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
- Примеры групп гомологов по данному дереву:
- белки с идентификатором CLPX -- АТФ-связывающая субъединица Х АТФ-зависимой протеазы Clp.
- Белки с идентификатором FtsH -- АТФ-зависимые цинк металлопротеиназы
- Белки RUVB_CLOBA и RUVB_LISMO -- ДНК-хеликазы.
- Примеры паралогов:
- Субъединицы АТФ зависимой Clp протеазы (субъединицы ClpX, HslU, ClpY, ClpC, ClpE, ClpL)
- ДНК-хеликазы RUVB и АТФ-зависимые цинк металлопротеиназы Ftsh
Рис.2 примеры эволюционных событий. Красным выделена дупликация генов, синим разделение путей эволюции белков в результате видообразования.