Ферменты и метаболические пути. База данных KEGG.
Выбор пары ортологических рядов для дальнейшей работы
Для работы я выбрала метаболический путь гистидина (Histidine metabolism)
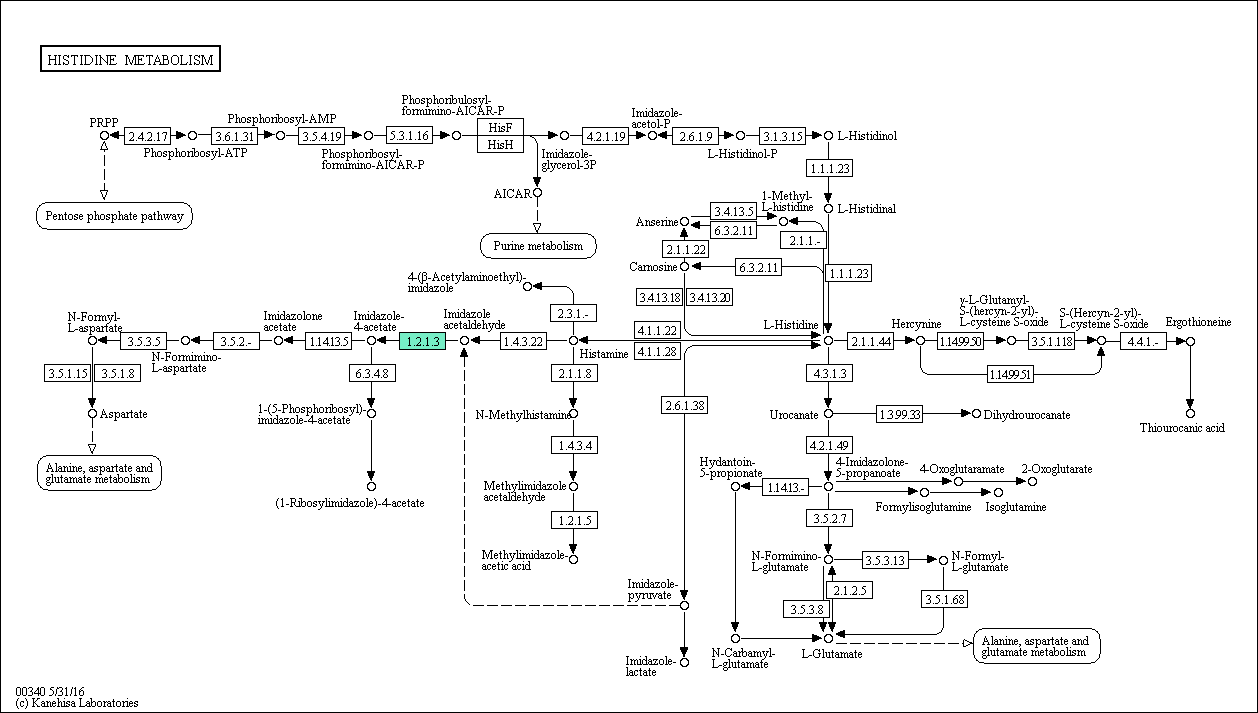
Рис. 1. Метаболический путь гистидина.
Я выбрала реакцию с ЕС-кодом 1.2.1.3. (На рис. 1 покрашена бирюзовым цветом). <.p>
Ее катализирует 3 ортологических ряда белков:
- K00128 (aldehyde dehydrogenase (NAD+)) (4778 белковых последовательностей в Uniprot) ;
- K00149 (aldehyde dehydrogenase family 9 member A1) (56 белковых последовательностей в Uniprot);
- K14085 (aldehyde dehydrogenase family 7 member A1) (125 белковых последовательностей в Uniprot).
Получение совместного множественного выравнивания
Сначала мы получили последовательности белков из Uniprot c помощью сервиса "Retrieve/ID mapping" этой базы данных.
С помощью скрипта модифицировали идентификаторы.
Итоговые файлы с последовательностями:
K14085.fasta
K00149.fasta
С помощью Muscle построил множественное выравнивание, которое открыли в Jalview и покрасили по Clustalx.
align.fasta
align.jvp
Проверка гомологичности белков в выравнивании
Выравнивание получилось плохого качества. Удалили короткие белки, которые содержали много гэпов в тех участках, где в других последовательностях выравнивания консервативные колонки. Например K7JC23_NASVI|K14085; G4YWR2_PHYSP|K14085; B6TPU3_MAIZE|K14085 и другие. Также удалили такие последовательности как Q4SSM8_TETNG|K00149; A0A158PZE7_BRUMA|K00149, которые длиннее прочих и имеют мало совпадений с другими последовательностями.
Итоговое выравнивание: align_modified.jvp
На рисунке 3 представлен фрагмент итогового выравнивания. Можно отчетливо выделить 2 блока. Гомологичными являются белки одного ряда, но белки двух рядов не гомологичны между собой, т.к. не обнаружено сколько-нибудь больших консервативных блоков между последовательностями из разных ортологических рядов. Таким образом, нельзя говорить о существовании множественного выравнивания.

Рис. 3. Фрагмент выравнивания, демонстрирующий негомологичность белков двух рядов.
Построение филогенетического дерева
Т.к. последовательности из двух ортологических рядов негомологичны, то нельзя строить дерево, потому что оно не будет нести биологического смысла.
