Определение сайтов связывания транскрипционного фактора в участке хромосомы человека
Работала с файлом chipseq_chunk12.fastq с ридами Illumina, полученными в результате ChIP-seq эксперимента.
Сделала контроль качества прочтений с помощью программы FastQC.
fastqc chipseq_chunk12.fastqОтчет report.html
Всего в файле 8325 ридов, качество чтений хорошее, нет необходимости в чистке с помощью Trimmomatic.
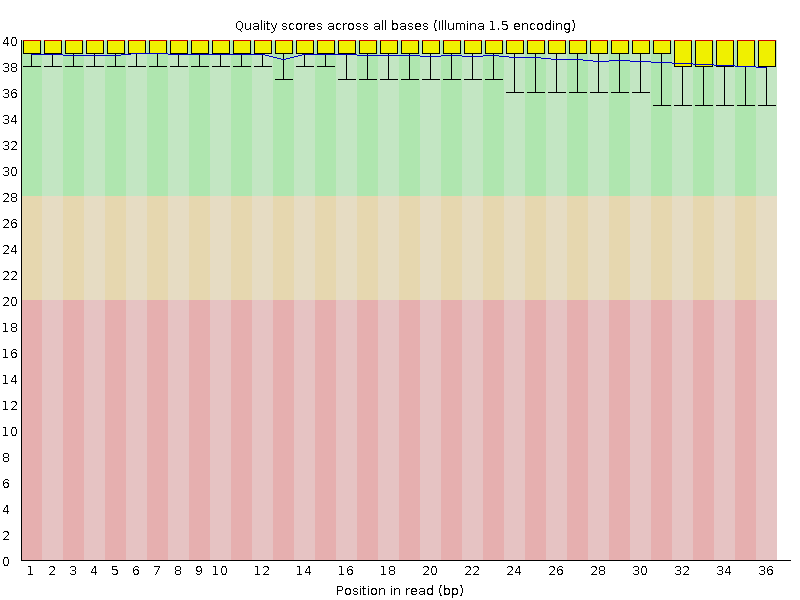
Рис. 1. Per Base Sequence Quality
Картировала прочтения на геном человека hg19 с помощью программы BWA. Провела анализ полученного файла
- Перевод в бинарный формат:
samtools view -bSo chipseq_chunk12.bam chipseq_chunk12.sam - Сортировка по координате в референсе начала чтения: samtools sort chipseq_chunk12.bam -T chip_temp -o chipseq_chunk12.sorted.bam
- Индексация: samtools index chipseq_chunk12.sorted.bam
- Чтобы ответить на вопрос, с какой хромосомы анализируемые прочтения используем:
samtools idxstats chipseq_chunk12.sorted.bam > chipseq_chunk12.idxstats
Больше всего ридов (7909) было откартировано на хромосому 3, следовательно, можно предположить, что для анализа были предложены прочтения с 3-ей хромосомы.
- Получение информации o количестве откартированных ридов: samtools view -c chipseq_chunk12.sorted.bam 8325 ридов было откартировано
Для поиска пиков (peak calling) воспользовалась программой MACS.
При использовании команды:macs2 callpeak -t chipseq_chunk12.sorted.bam
выдавала ошибка --- слишком мало пиков. Запустила с другими параметрами:
macs2 callpeak -n chunk12 -t chipseq_chunk12.sorted.bam --nomodel
Получила три выходных файла:
- chunk12_peaks.narrowPeak
- chunk12_peaks.xls
- chunk12_summits.bed
Все они содержат информацию о найденных пиках. Наиболее полная информация представлена в файле chunk12_peaks.xls:
| chr | start | end | length | abs_summit | pileup | -log10(pvalue) | fold_enrichment | -log10(qvalue) | name |
|---|---|---|---|---|---|---|---|---|---|
| chr3 | 46441143 | 46441361 | 219 | 46441233 | 32.00 | 21.69714 | 7.89474 | 14.86425 | chunk12_peak_1 |
| chr3 | 46441776 | 46441975 | 200 | 46441896 | 26.00 | 15.73521 | 6.42857 | 9.24055 | chunk12_peak_2 |
| chr3 | 46448485 | 46448761 | 277 | 46448620 | 31.00 | 20.06465 | 7.37327 | 13.34609 | chunk12_peak_3 |
| chr3 | 46464159 | 46464461 | 303 | 46464290 | 47.00 | 42.43318 | 13.40782 | 35.03260 | chunk12_peak_4 |
| chr3 | 46550736 | 46550972 | 237 | 46550875 | 26.00 | 15.60623 | 6.36792 | 9.11946 | chunk12_peak_5 |
| chr3 | 46976977 | 46977286 | 310 | 46977125 | 59.00 | 56.10939 | 15.70681 | 46.64811 | chunk12_peak_6 |
| chr3 | 46988826 | 46989067 | 242 | 46988945 | 36.00 | 21.38397 | 6.92884 | 14.56614 | chunk12_peak_7 |
| chr3 | 46989533 | 46990017 | 485 | 46989660 | 36.00 | 20.80683 | 6.70290 | 14.00642 | chunk12_peak_8 |
| chr3 | 47018487 | 47018731 | 245 | 47018621 | 26.00 | 16.20140 | 6.65025 | 9.67664 | chunk12_peak_9 |
| chr3 | 47058618 | 47058842 | 225 | 47058739 | 20.00 | 14.05000 | 6.90789 | 7.72829 | chunk12_peak_10 |
Найдено 10 пиков. Они расположены в одном регионе хромосомы 3. Чем выше показатели -log10pvalue и -log10qvalue, тем ниже соответствующие показатели p-value и q-value, а следовательно, тем достовернее пик. Наиболее достоверны пики 4 и 6, наименее достоверный - пик 10.
Визуализировала пики с помощью UCSC Genome Browser.
Использовала файл chunk12_peaks.narrowPeak, предварительно
добавив в него дополнительную информацию:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks"
description="Peaks from chunk 12"
browser position chr3:46441000-47059000
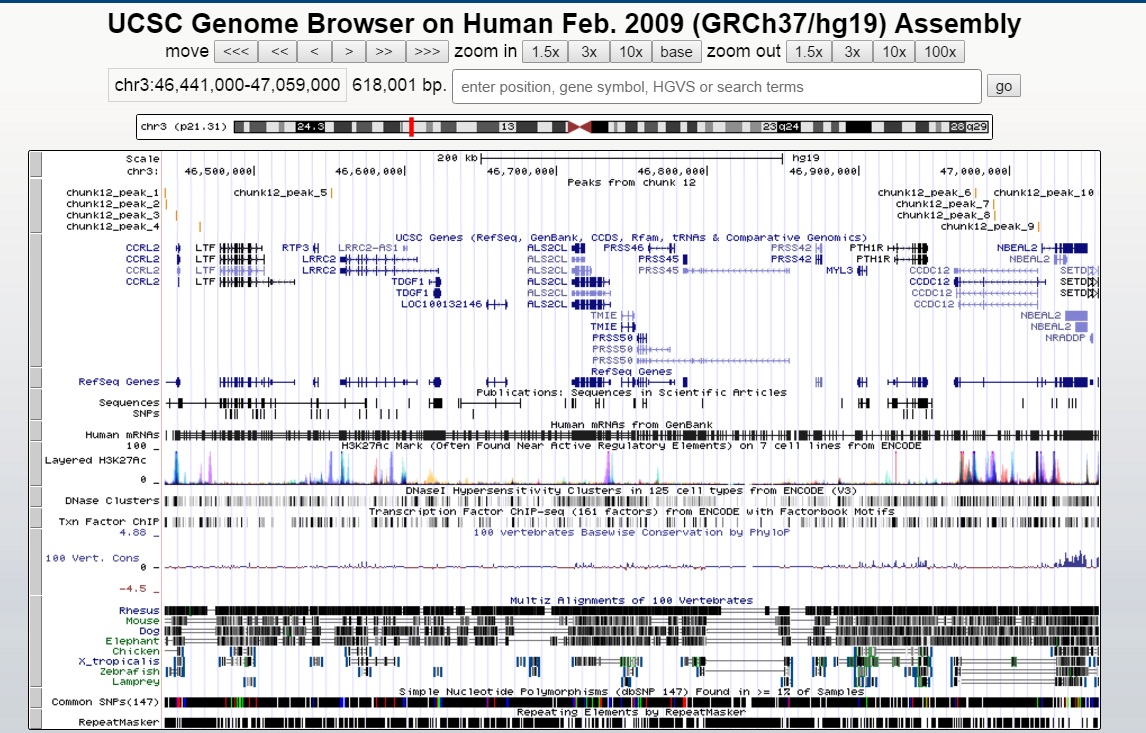
Рис. 2. Визуализация пиков в UCSC Genome Browser ссылка
Рассмотрим подробнее один из наиболее достоверных пиков --- пик 6.
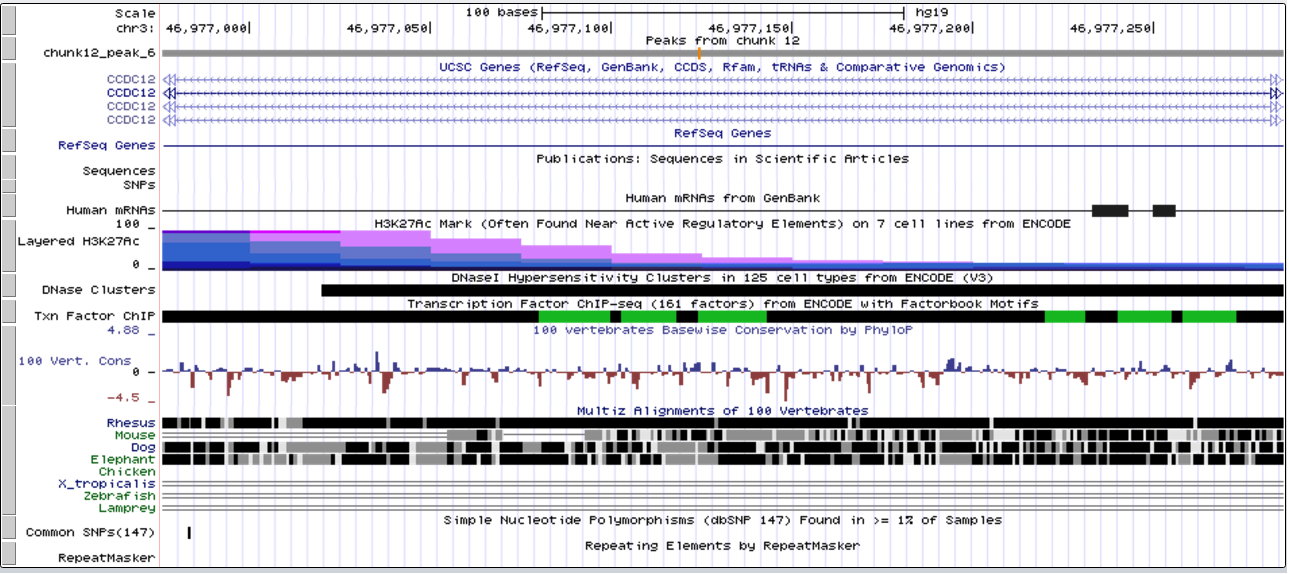
Этот пик пересекает ген CCDC12 с координатами chr3:46,963,220-47,023,500. Данный ген кодирут белок, входящий в состав сплайсосомы.
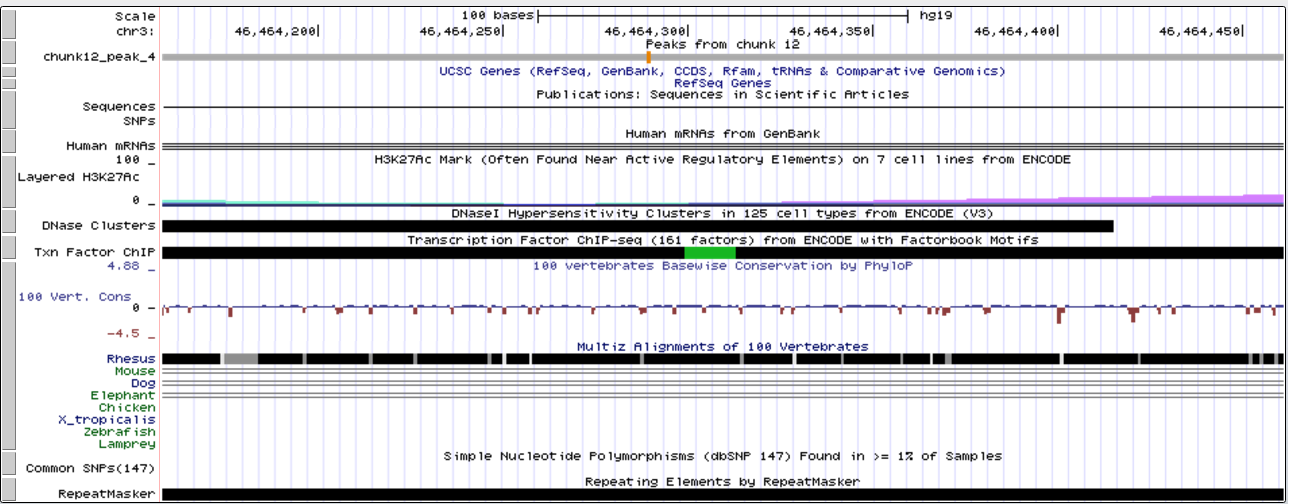
Другой достоверный пик --- пик 4. Не пересекается ни с каким геном.
