Восстановление "экспериментальной" электронной плотности
Выбор структуры
Для работы я выбрала белок Глюкокиназу с PDB ID 3vev. Глюкокиназа катализирует реакцию превращение D-глюкозы в D-глюкозо-6-фосфат, сопровождающуюся гидролизом АТФ.
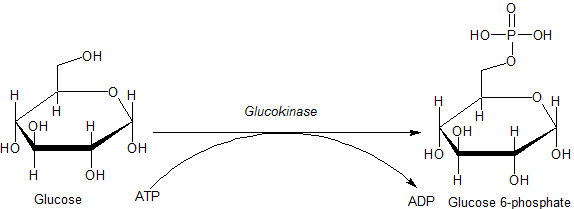
Рисунок 1. Реакция превращения глюкозы в глюкозо-6-фосфат, катализируемая глюкокиназой. Источник [1].
Кристаллическая структура 3vev с разрешением 1.8 Å представлет из себя глюкокиназу в комплексе с глюкозой и активатором.
Выбранная структура соответствует требованиям:
- Со страницы структуры на сайте PDBe доступен для скачивания файл с электронной плотностью и файл структурных факторов.
- Имеются подходящие структуры белков, сходных c выбранным белком. Таблица находок PDBeFOLD. Есть 5 или более разных белков с RMSD (колонка с оценкой сходства) от 0,8 до 3 ангстрем и N_align от 50% (235 AA) до 90% (423 AA) от числа аминокислотных остатков входного белка (470 АA).
- По результатам расшифровки структуры опубликована статья [2].
Построение изображения электронной плотности вокруг остова полипептидной цепи
При визуализации используются не физические единицы измерения — "заряд/кубический ангстрем", а статистические — превышение Z сигнала над шумом. Для вычисления Z рассчитывается среднее значение ЭП по ячейке кристалла M и среднее квадратичное отклонение σ . Для каждой точки вычисляется Z = (R – M)/σ. Здесь R — значение ЭП в данной точке пространства (x,y,z) в физических единицах, Z — значение ЭП "в сигмах" в той же точке.
Загружаем PDB-файл и файл с электронной плотностью, оставляем только атомы остова.
load pdb3vev.ent, protein #загружаем pdb файл со структурой load 3vev.ccp4, map #загружаем карту электронной плотности remove hetatm #убираем воду и лиганды remove sc. #убираем атомы боковых цепей #таким образом остается только остов (bb.)
Чтобы изобразить электронную плотность с уровнем подрезки 0.5, нужна следующая команда:
isomesh new_surface, map, 0.5, bb., carve = 2.0
Изображение электронной плотности с разными уровнями подрезки представлено на риисунке 1. Видно, что при увеличении уровня подрезки электронная плотность концентрируется, сужается к центрам атомов. Также можно заметить, что с увеличением уровня подрезки "оголяются" N- и С-концы, а также экспонированные петли. Возможно, это происходит из-за того, что данные части глобулы более подвижны.
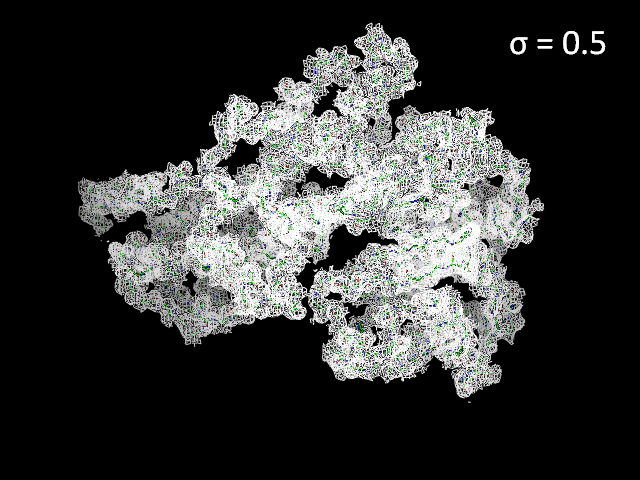
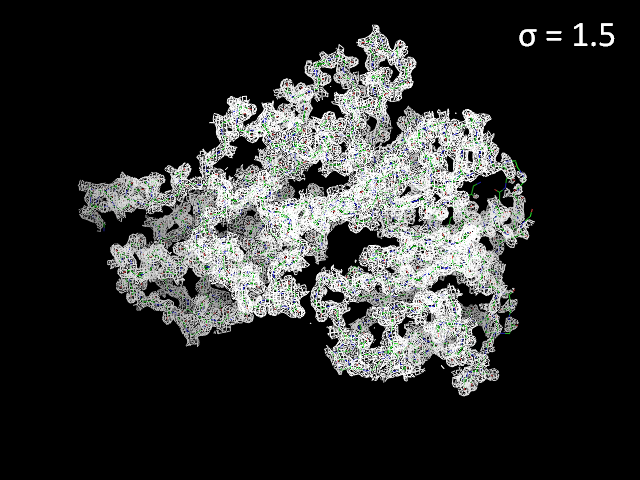
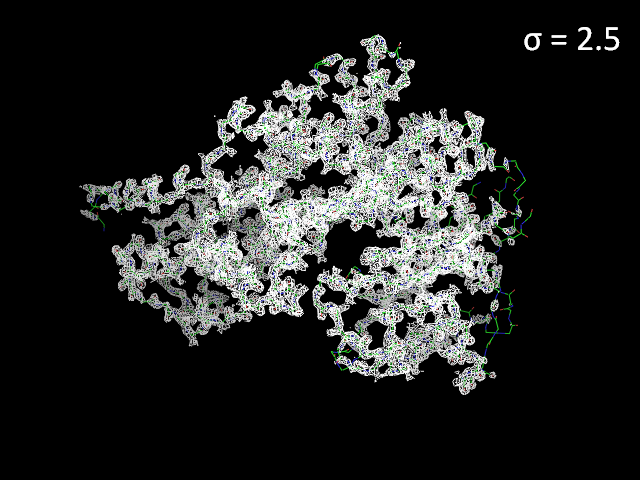

Рисунок 1. Электронная плотность вокруг остова полипептидной цепи с разным уровнем подрезки.
Чтобы увеличить картинку, нажмите на нее.
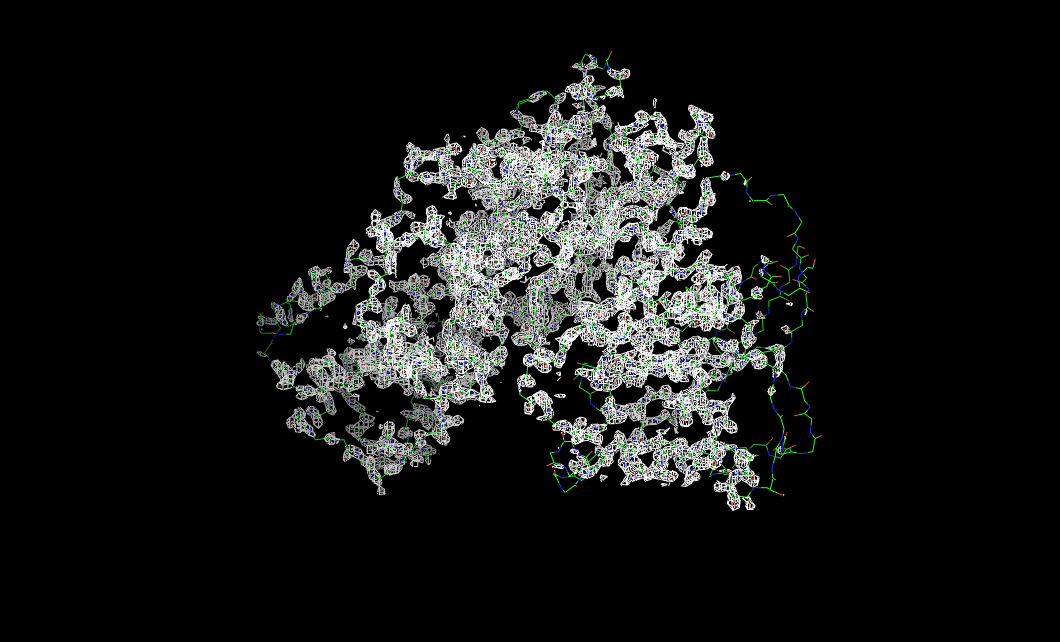
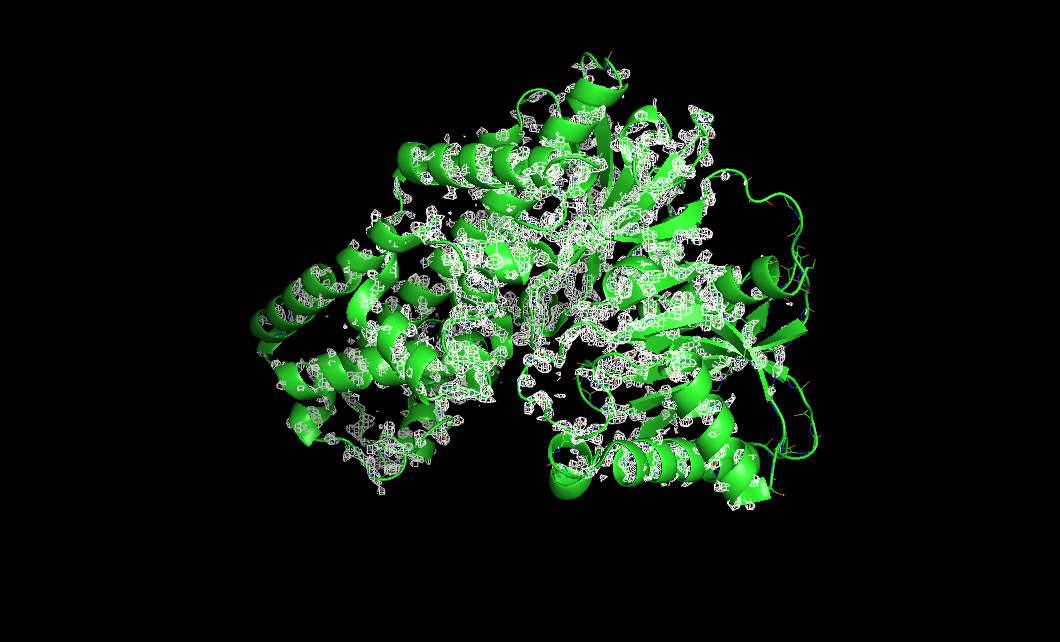
Рисунок 2. Электронная плотность вокруг остова полипептидной цепи с σ = 3. При таком уровне подрезки подвижные участки белковой глобулы (N- и С-конец, петли) "оголяются".
Чтобы увеличить картинку, нажмите на нее.
Построение изображения электронной плотности вокруг вокруг трех различных аминокислотных остатков
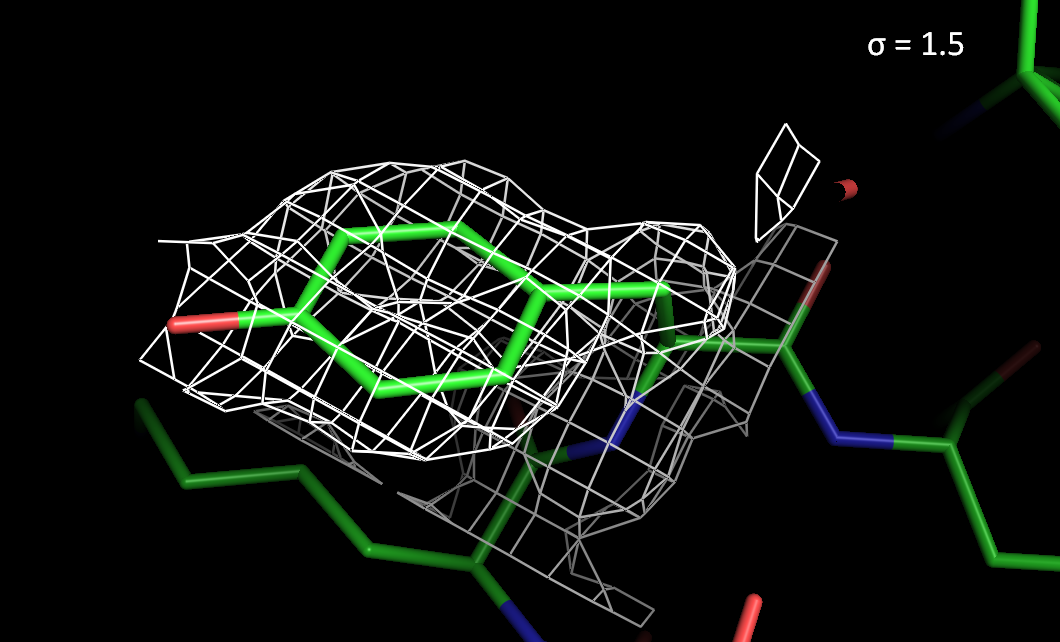
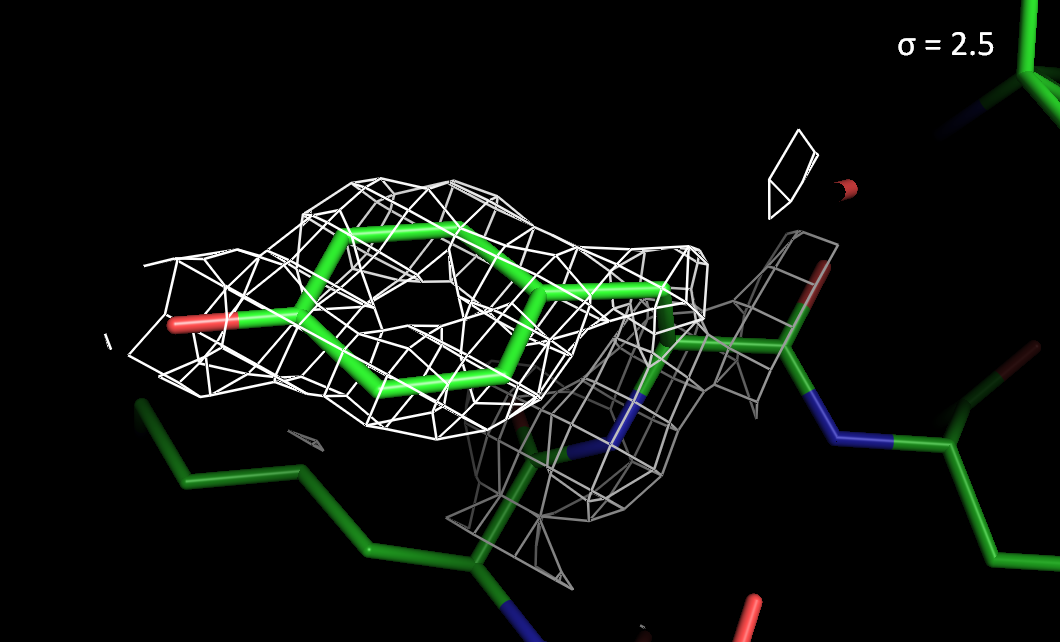
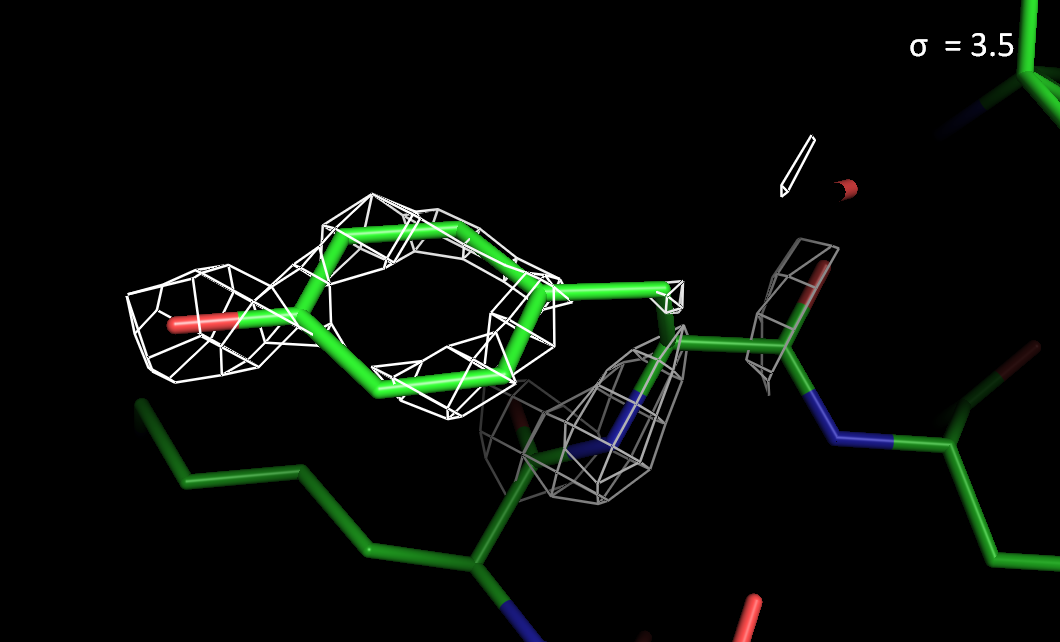
Рисунок 2. Электронная плотность вокруг остатка Tyr297 с разным уровнем подрезки.
Чтобы увеличить картинку, нажмите на нее.
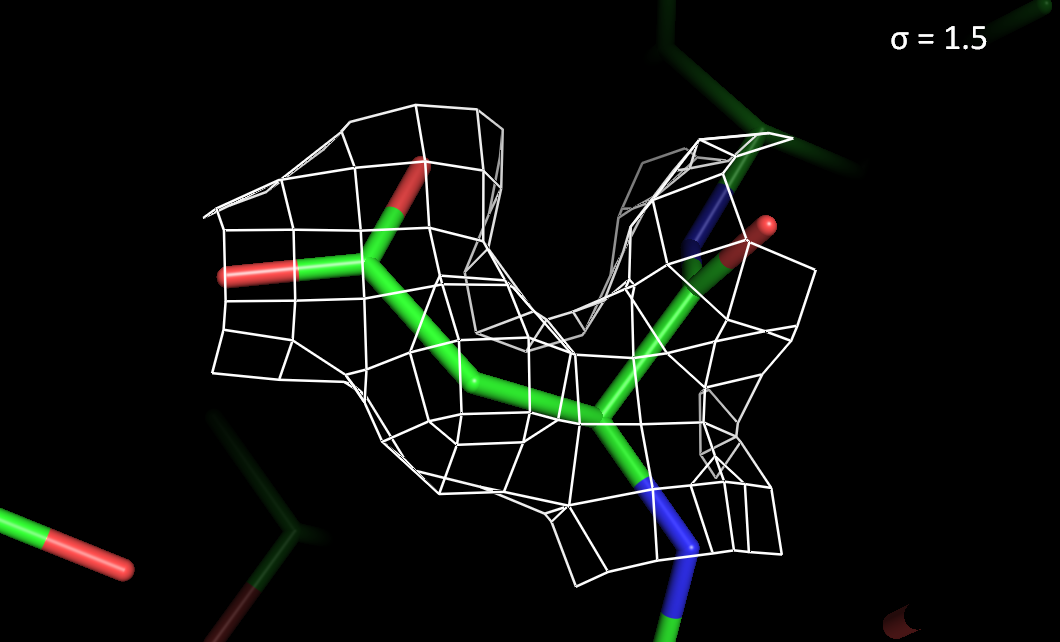
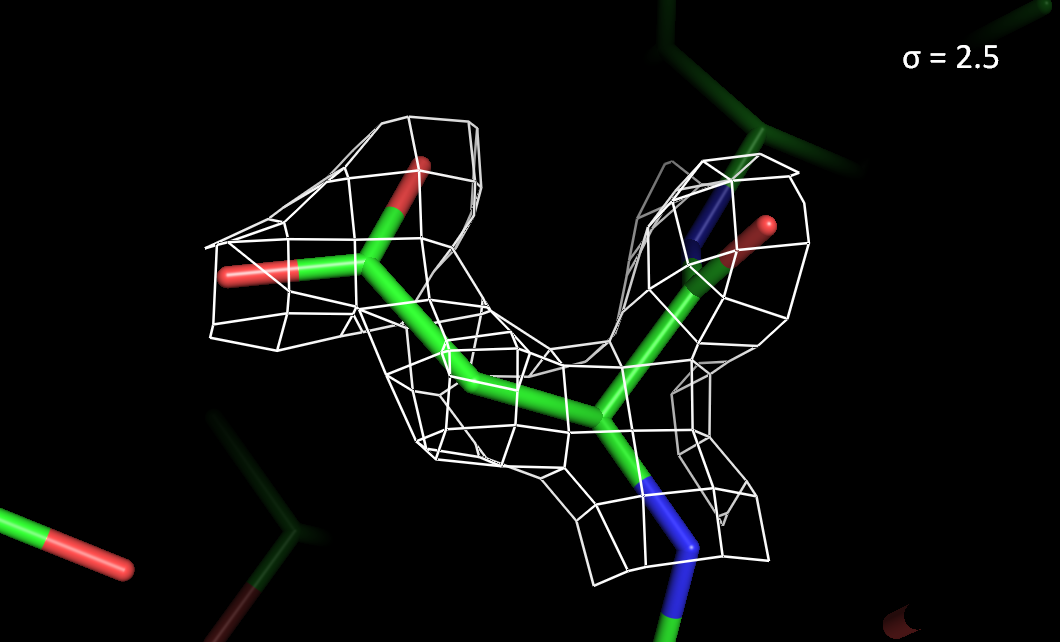
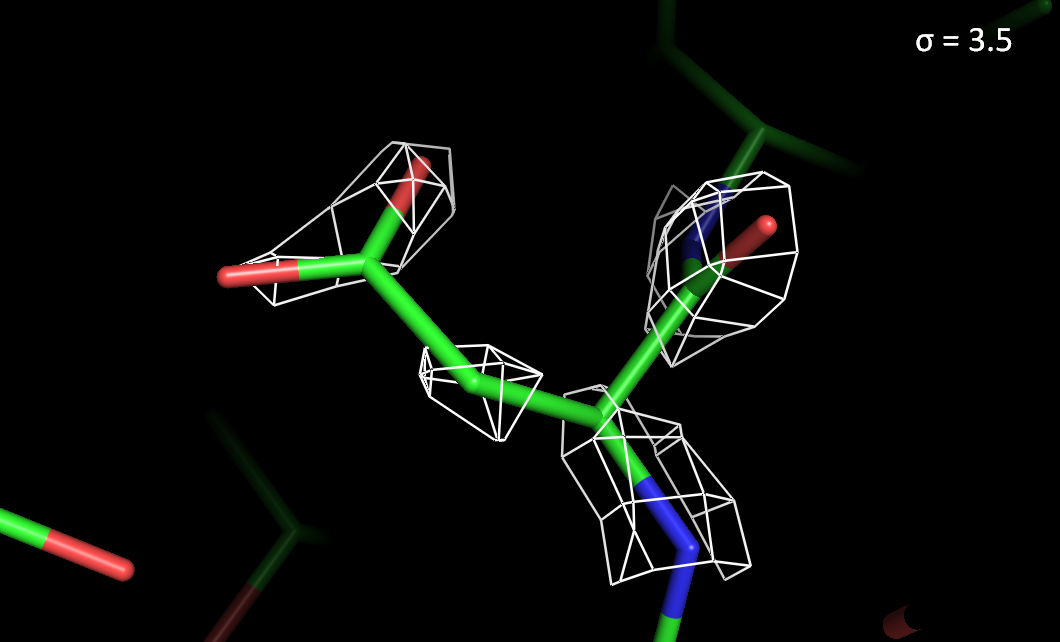
Рисунок 3. Электронная плотность вокруг остатка Asp78 с разным уровнем подрезки.
Чтобы увеличить картинку, нажмите на нее.
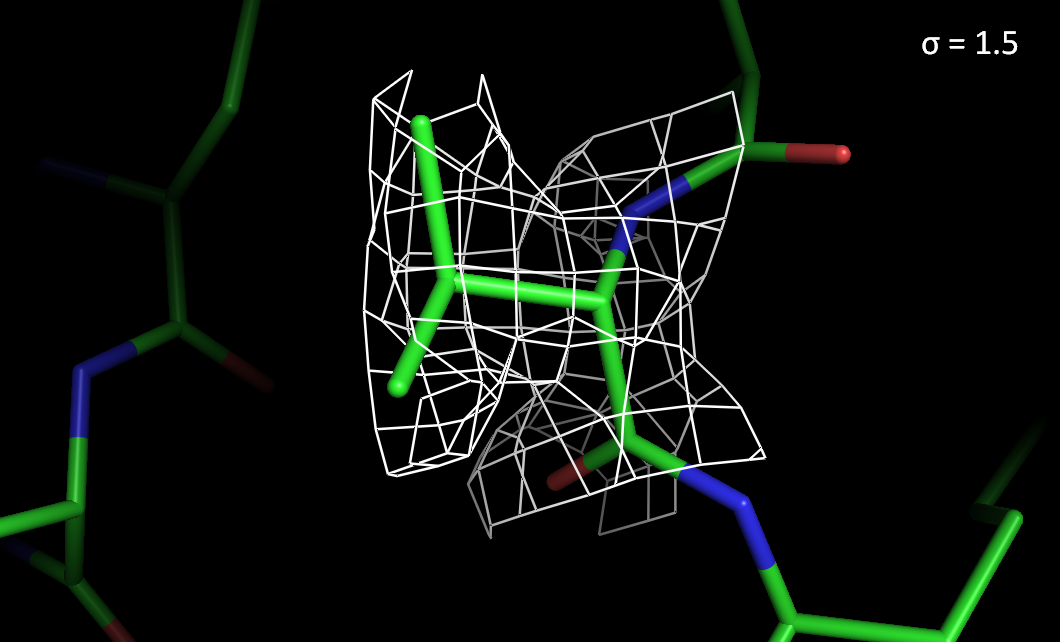
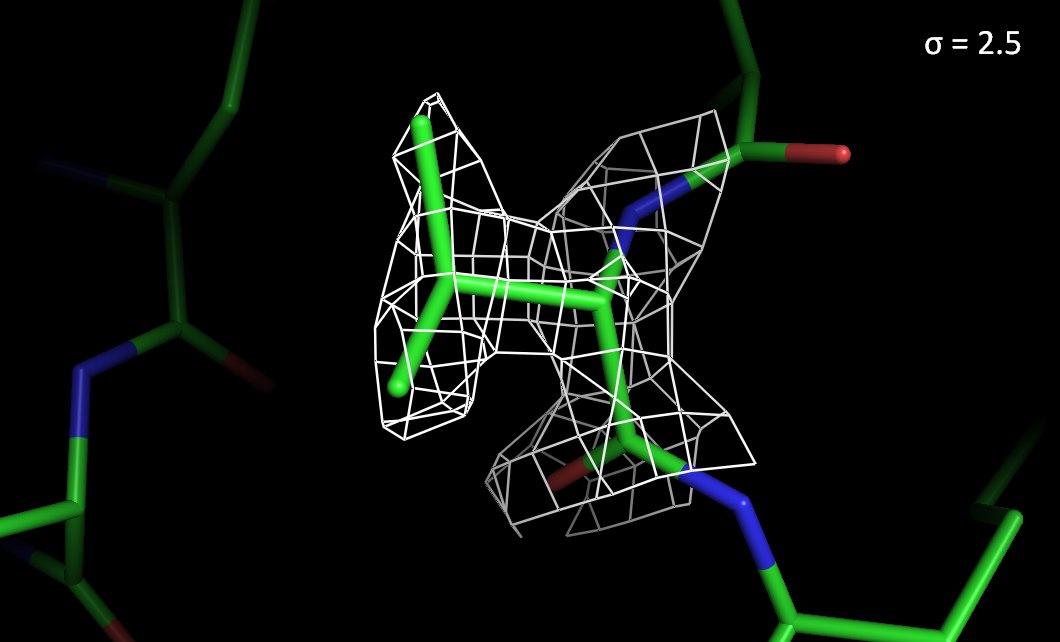
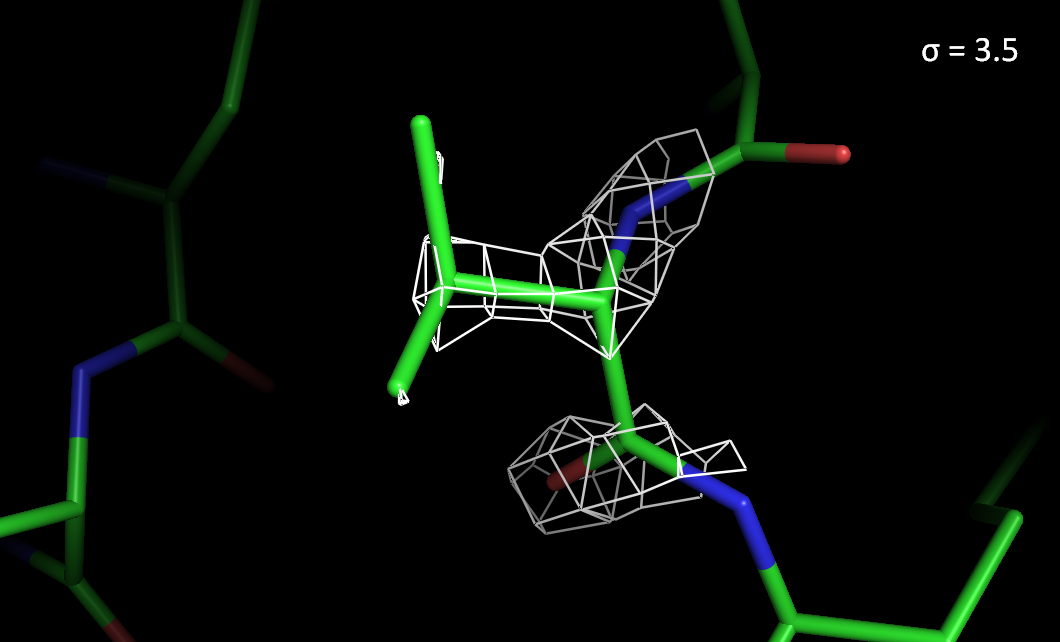
Рисунок 4. Электронная плотность вокруг остатка Val55 с разным уровнем подрезки.
Чтобы увеличить картинку, нажмите на нее.
> При высоких уровнях подрезки электронная плотность лучше сохраняется на атомах азота и кислорода, чем на атомах углерода.
> На ароматическом остатке Tyr297 электронная плотность сохраняется лучше, чем на Val55.
Ссылки:
[1] Wikipedia.org/Glucokinase[2] Liu S. et. al. Insights into mechanism of glucokinase activation: observation of multiple distinct protein conformations. The Journal of Biological Chemistry. Feb 2012, 287(17):13598-13610. DOI: 10.1074/jbc.M111.274126 pdf
