Восстановление кристалла из PDB файла
Для работы я выбрала белок Глюкокиназу с PDB ID 3vev. Глюкокиназа катализирует реакцию превращение D-глюкозы в D-глюкозо-6-фосфат, сопровождающуюся гидролизом АТФ.
Кристаллографические характеристики 3vev из записи PDB (содержатся в поле CRYST1):
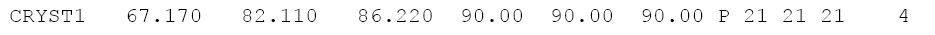
- длины направляющих векторов кристалла – a: 67.17Å, b: 82.11Å, c: 86.22Å
- углы между направляющими векторами – α: 90°, β: 90°, γ: 90°
- кристаллографическая группа – P212121 (Это примитивная группа с тремя осями винтовой симметрии 21 (поворот на 1/2 периода, 180 градусов)
- число молекул в ячейке – 4
На рисунке 1 приведено изображение части кристалла. Чтобы получить такую картинку мы в Pymol вводили следующие комманды:
load pdb3vev.ent, 3vev symexp crystal, 3vev, all, 30
Таким образом отобразили для объекта 3vev все образы, находящиеся не далее чем на 5Å, чтобы рассмотреть соседние молекулы на близком расстоянии, и образы на расстоянии 30Å, чтобы показать общий вид кристалла. Также изобразили границу элементарной ячейки (командой show cell). Полученные изображения представлены на рисунках 2 и 3.
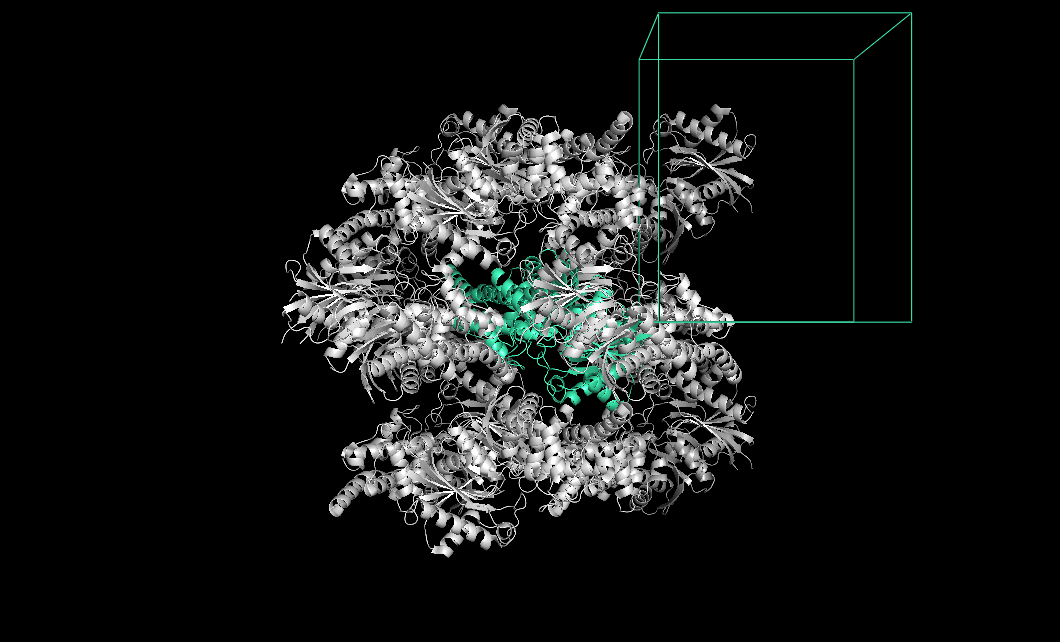
Рисунок 2. Изображение соседних асимметрических единиц в пределах 5Å.
Рисунок 3. Структура кристалла с изображением границ элементарной ячейки. Для объекта 3vev изображены все образы, находящиеся не далее чем на 30Å.

Рисунок 4. Структура кристалла с изображением границ элементарной ячейки. Другая проекция. Для объекта 3vev изображены все образы, находящиеся не далее чем на 20Å.
Затем нужно было исследовать зоны контакта между белками из соседних ячеек. При восстановлении кристалла получилось, что исходная цепь, из которой был восстановлен кристалл, не попала в ячейку и еще молекулы, которые в ячейку попадают, расположены сразу в двух ячейках, поэтому исследовали контакты не между белками из соседних ячеек, а просто между соседними молекулами белка.
Чтобы показать водородные связи между соседними молекулами в кристалле, мы определили два множества полярных атомов в соседних молекулах, после чего отобразили остатки, вовлечённые в водородные связи. В качестве критерия водородной связи брали расстояние не более 3.5 Å между полярными атомами белков. Молекулы воды не учитывались. В PyMOL это было сделано c помощью следующих команд:
symexp crystal, 3vev, all, 5 #восстановили ближайших соседей в кристалле remove hetatm # убираем молекулы воды и лиганды select set_a, 3vev and name n*+o* # полярные атомы в 3vev select set_b, all & (not) 3vev and name n*+o* #полярные атомы в окружающих молекулах #показали водородные связи (критерий водородной связи расстояние менее 3.5 Å distance hbonds, set_a, set_b, 3.5 # показали остатки в месте контакта select set1, byres (set_a and (set_b around 3.5)) select set2, byres (set_b and (set_a around 3.5)) show lines, set1 show lines, set2
Таким образом показали водородные связи, остатки, между которыми есть водородные связи показали линиями и еще места контакта покрасили красным. Результат на рисунке 5.
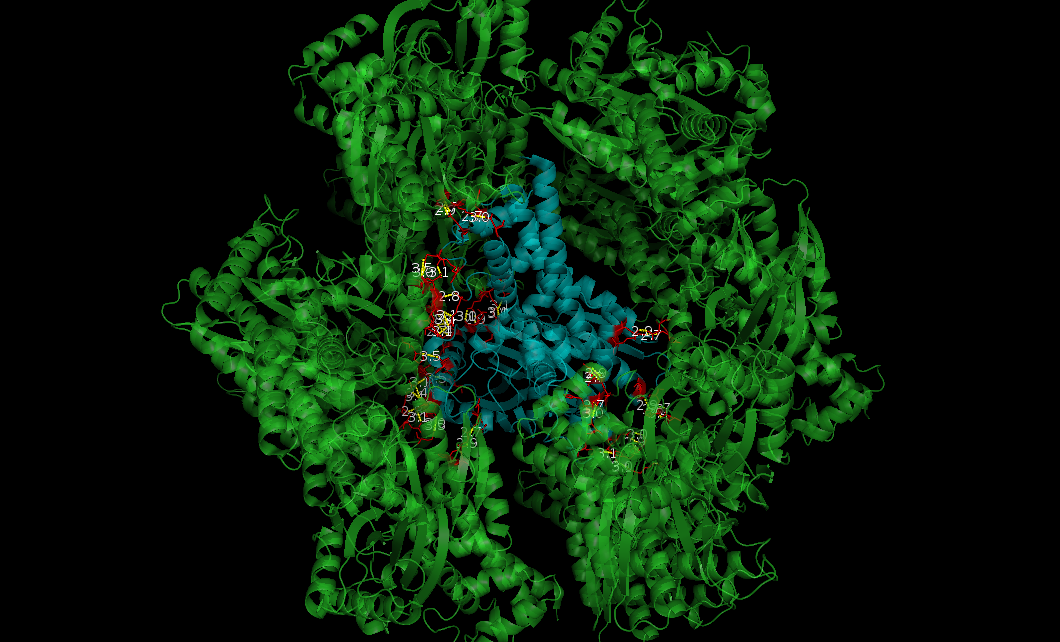
Рисунок 5. Зоны контакта между соседними молекулами в кристалле.
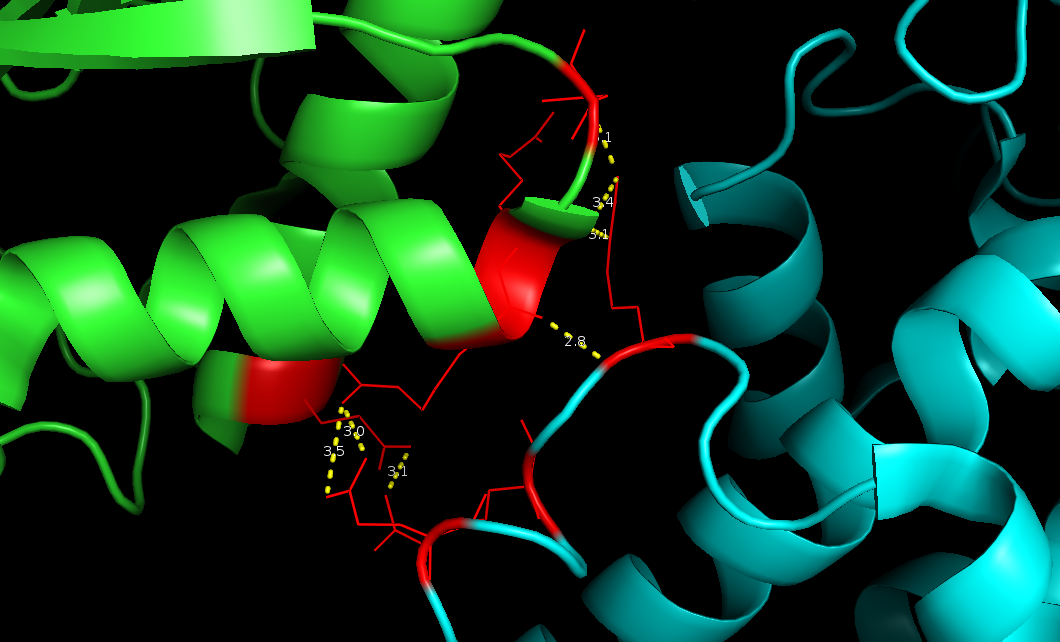
Рисунок 6. Одна из зон контакта в близком рассмотрении.
В зонах контакта число водородных связей и контактирующих остатков небольшое. При ближашем рассмотрении оказывается, что в контакт происходит через остатки, принадлежащие петлям (например как на рисунке 6). Так как глюкокиназа – это мономер, контакты между белками можно считать артефактами кристаллизации, никак не отражающими природные взаимодействия.
PyMOL сессия, если захочется посмотреть еще что-нибудь.
Cтранное расположение белковых цепей в структуре ДНК-белкового комплекса.
Для работы я выбрала структуру комплекса гомеодомена engrailed Drosophila melanogaster и ДНК с разрешением 2.20 Å 3hdd
Если просто загрузить структуру командой fetch 3hdd, то расположение одной из полипептидных цепей (Цепь В) кажется странным – эта цепь как бы свисает с края цепи ДНК. (рисунок 7, полипептидная цепь В со странным расположением окрашена синим). Но если восстановть соседнюю ячейчку, то видно что на самом деле белковая цепь В взаимодействует с двухцепочечной ДНК не только из своей, но и из соседней ячейки (Рисунок 8).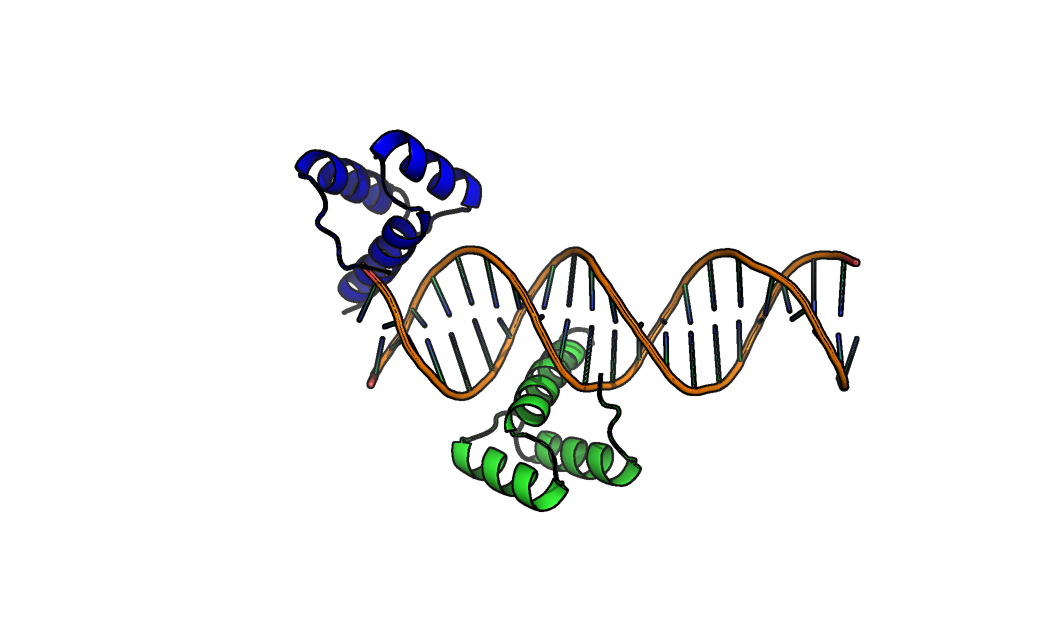
Рисунок 7. Структура 3hdd. Одна из полипептидных цепей (окрашена синим) расположена странно – слишком близко к краю.
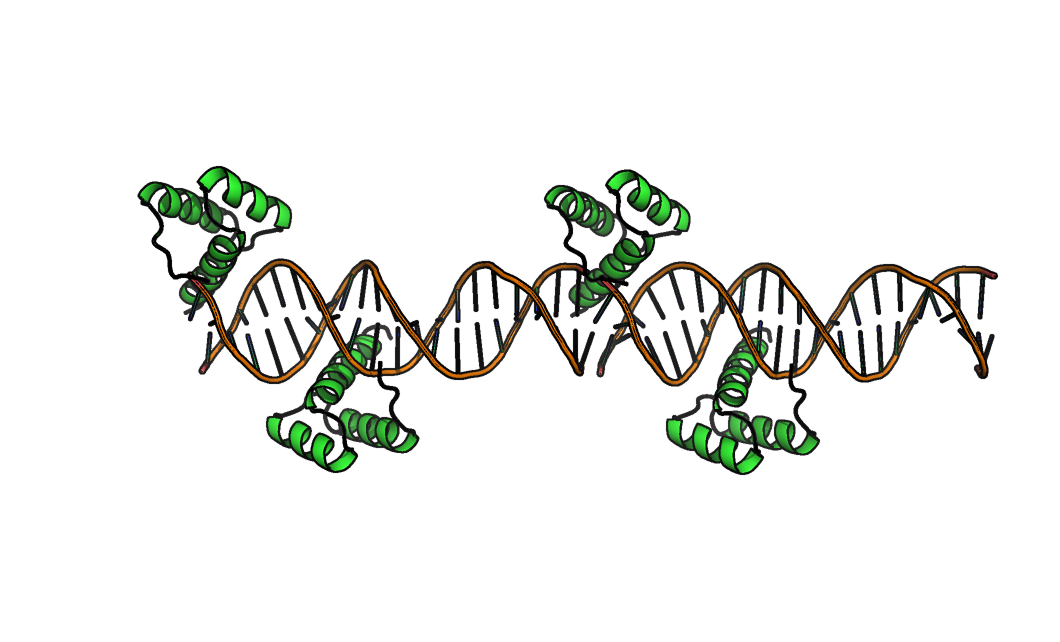
Рисунок 8. Структура 3hdd. Восстановена соседняя ячейка. Полипептидная цепь, положение которой казалось странным, взаимодействуетс ДНК в соседней ячейке.
Примеры PDB файлов, асимметрическая единицы которых не совпадает с биологической единицей
Для поиска таких структур воспользовались Аdvanced search в PDB по полям number of chains (asymm. unit) и number of chains (Biol. assembly), задав для них разные значения, например 1 и 2 или 2 и 1 или 2 и 4 и т.д. Чтобы визуализировать такие структуры, а использовала комманду set assembly. PyMOL загрузит ассиметрическую единицу, если assembly="0" и биологическую единицу, если assembly="1".
Пример белка у, которого в ассиметрической единице 2 цепи, а в биологической 4 цепи – алкоголь дегидрогеназа из Lactobacillus brevis 6h07
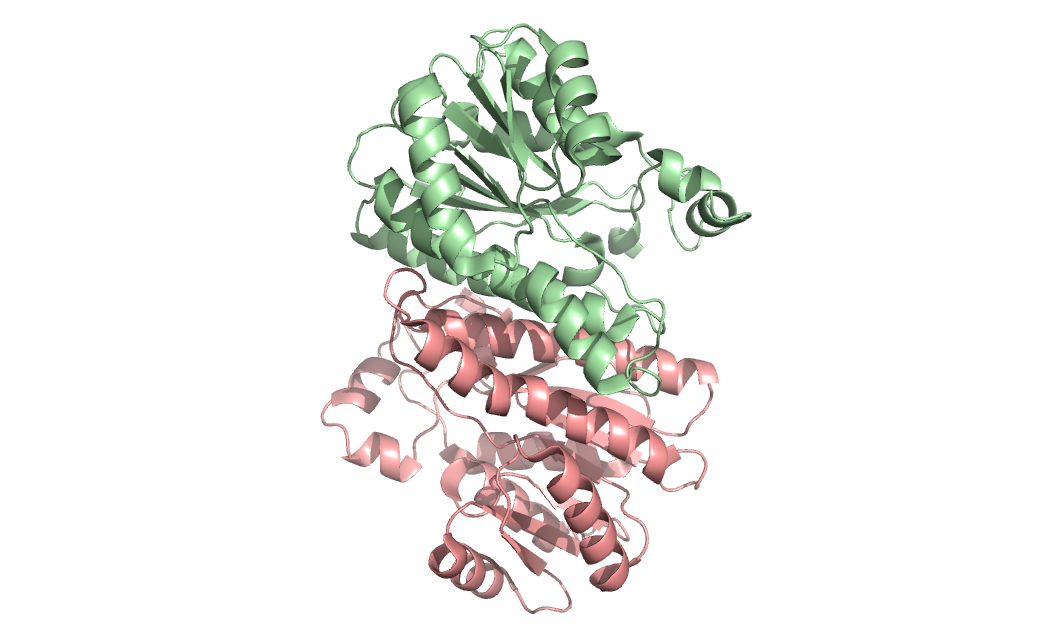
Рисунок9. Ассиметрическая единица 6h07 из 2х цепей
set assembly, 0 fetch 6h07 show cartoon
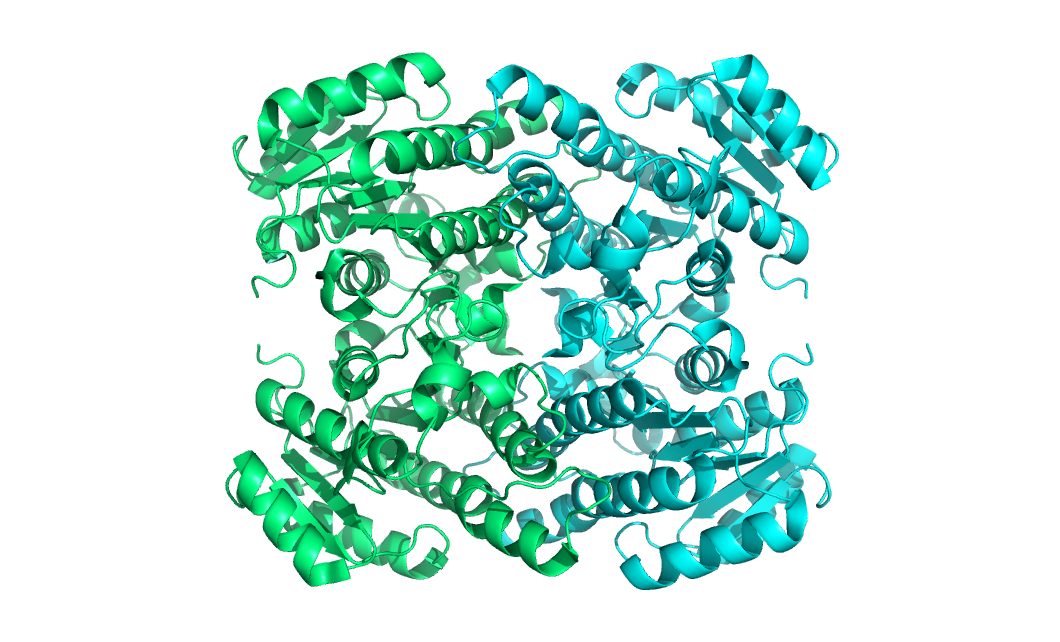
Рисунок 10. Биологическая единица 6h07 из 4х цепей (из 2х ассиметрических единиц)
set assembly, 1 fetch 6h07 show cartoon
Другой пример белка с разным количеством цепей в ассиметрической и биологической единицах – PPARgamma лиганд связывающий домен человека 6mcz, у него в ассиметрической единице 2 цепи, а в биологической – одна
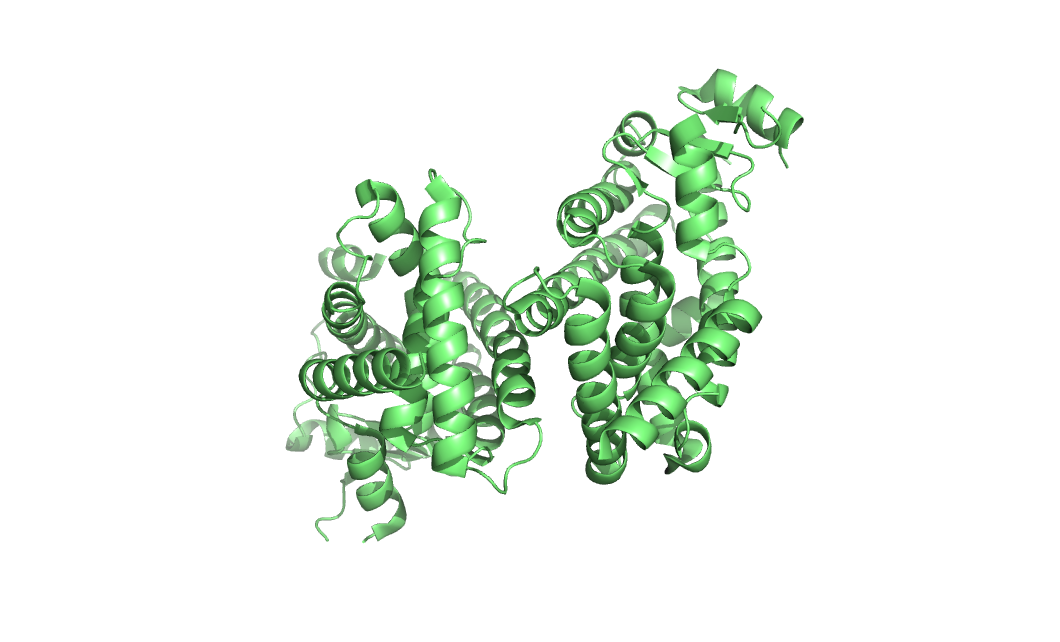
Рисунок 11. Ассиметрическая единица 6mcz из 2х цепей, которые не абсолютно идентичны.
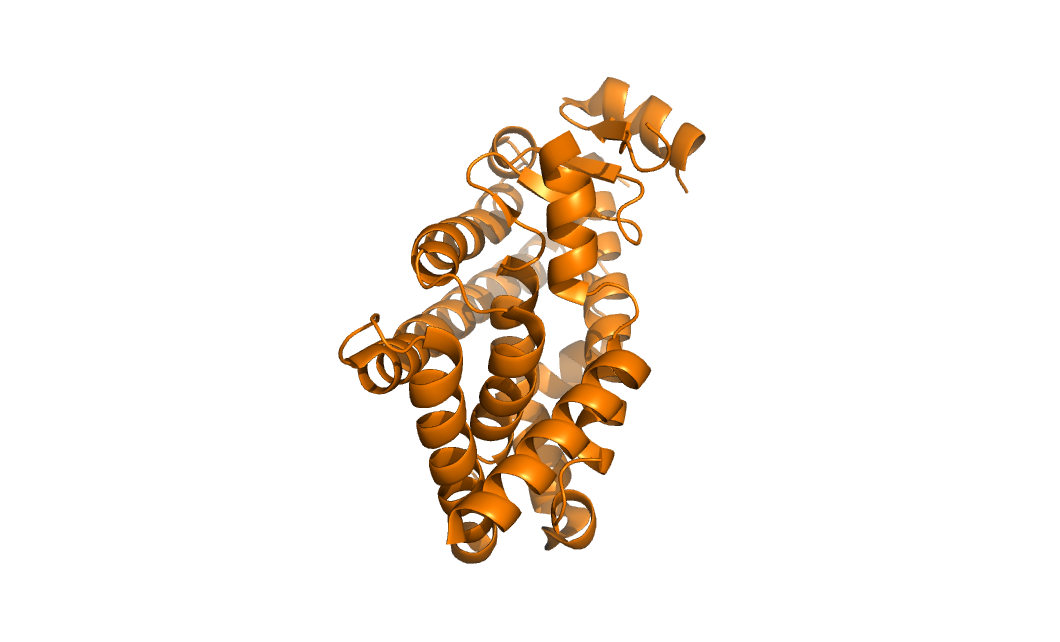
Рисунок 12. Биологическая единица 6mcz из 1ой цепи. В природе этот белок является мономером.
