Поиск по PDB и содержание PDB файла
Occupancy не равно 1
Для того чтобы найти такие структуры, я искала в PDBe с помощью Advanced search структуры, решенные с помощью X-ray с разрешением 1 - 1.5 Å. Вот 2 примера: структура 6gz8 (на рисункке 1 фрагмент PDB файла) и структура 5lkv фрагмент PDB файла на рисунке 2.
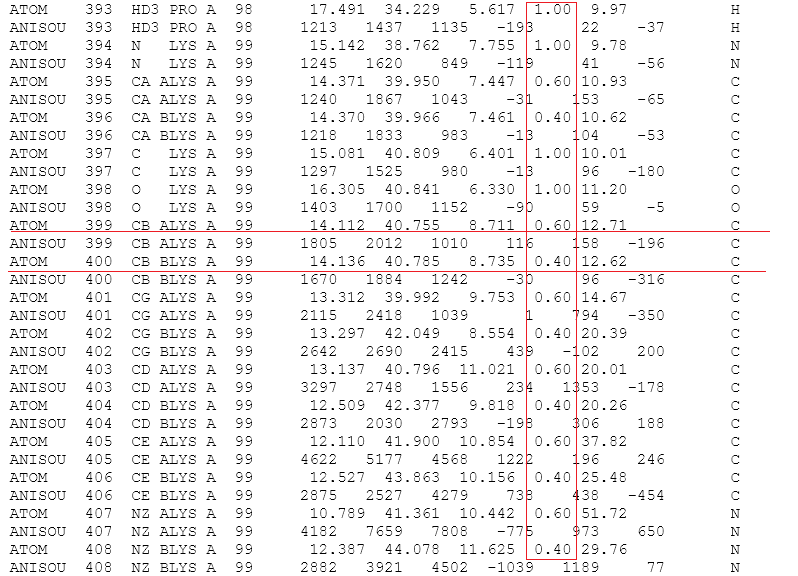
Рисунок 1. Фрагмента PDB файла 6gz8. Красной рамочкой выделена колонка Occupancy. Для нескольких атомов остатка Lys99 оccupancy равно 0.6 и 0.4.
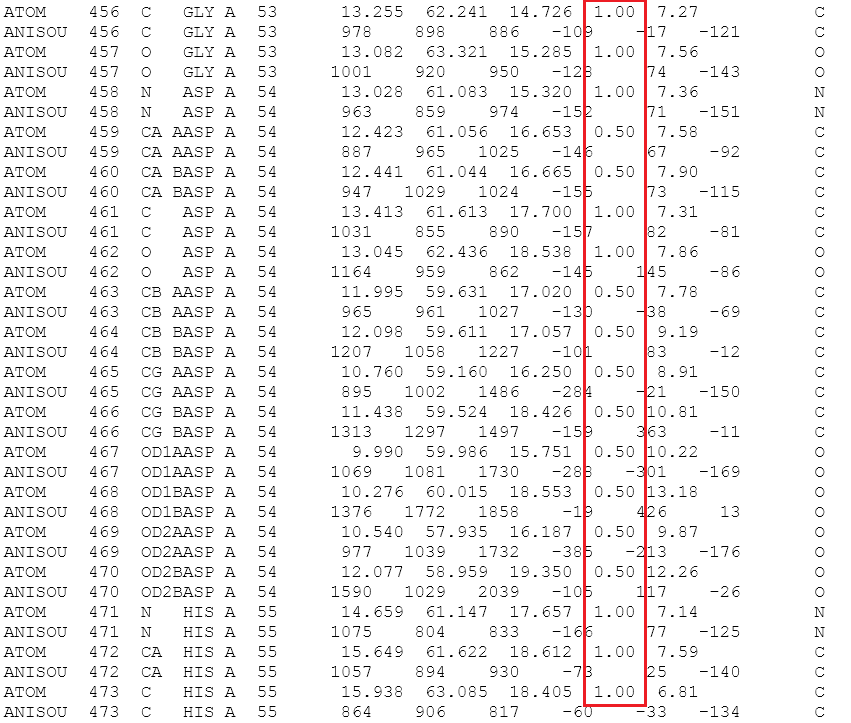
Рисунок 2. Фрагмента PDB файла 5lkv. Красной рамочкой выделена колонка Occupancy. Для нескольких атомов остатка Asp54 оccupancy равно 0.5.
Не расшифрованные аминокислотные остатки (Missing residues)
Не расшифрованные аминокислотные остатки я нашла в PDB файле структуры 3vev, с которой работала ранее. Фрагмент PDB файла представлен на рисунке 3. Не расшифрованы аминокислотные остатки (Missing residues) на N- и С-конце белка, а также не расшифрованы остатки 94-98 – возможно, они не расшифрованы из-за высокой подвижности данной петли. На рисунке 4 можно увидеть, как это выглядит при визуализации в Pymol.
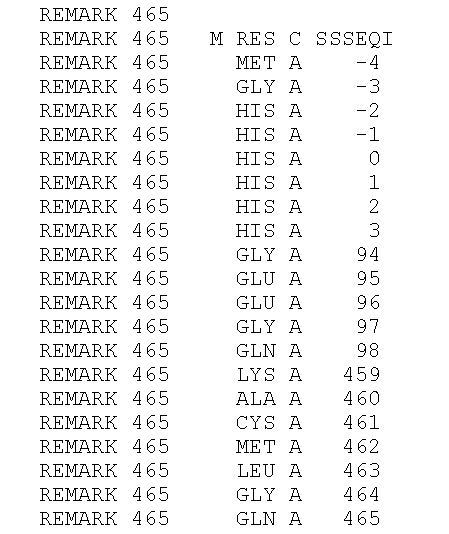
Рисунок 3. Фрагмента PDB файла 3vev. Не расшифрованные аминокислотные остатки (Missing residues).
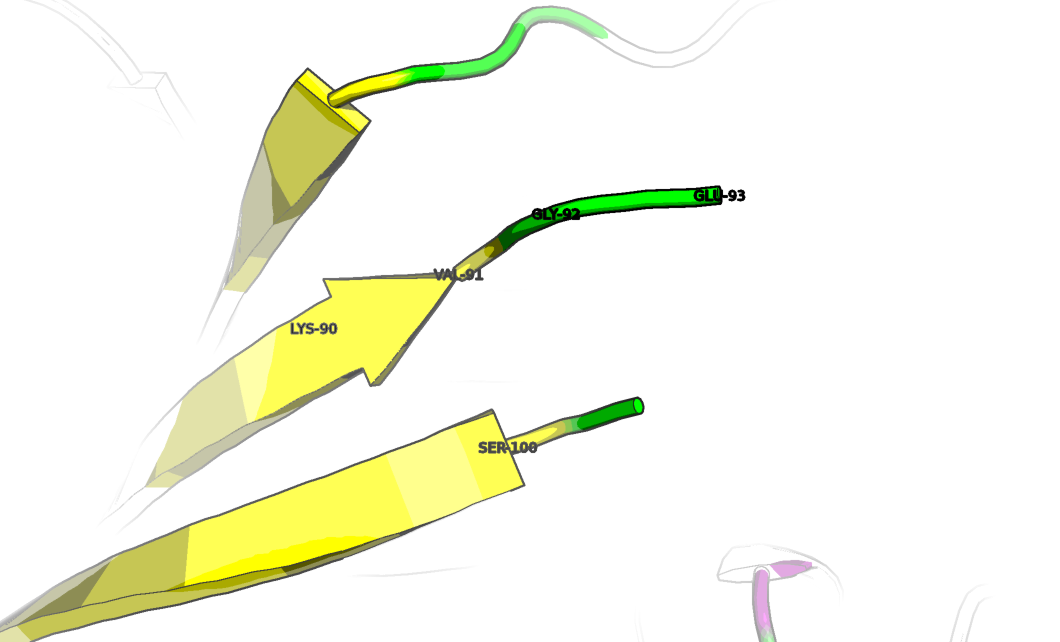
Рисунок 4. Аминокислотные остатки 94-99 не расшифрованы и не отображаются.
Различия последовательностей природного и кристаллизованного белка
C PDB структурой белка связаны три последовательности: (1) последовательность природного белка из Uniprot; (2) последовательность белка, который кристаллизовали – он может отличаться наличием тэгов, и быть частью природного белка, например, доменом; (3) та часть последовательности (2), которую удалось кристаллизовать. Для структуры 3vev последовательности (1) и (2) не совпадают, так как белок был экспрессирован с тегом (различия – во фрагмента PDB файла на рисунке 5). Также удалось кристаллизовать не весь белок – missing residues обсуждались в предыдущем разделе (см. рисунок 3).
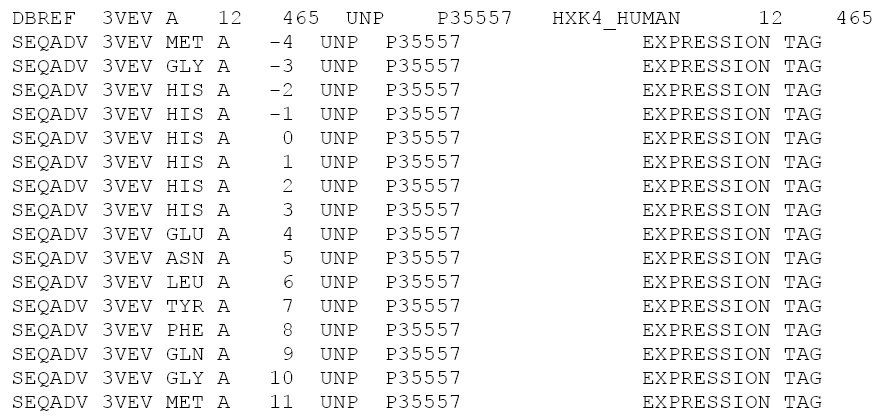
Рисунок 5. Различия последовательностей природного и кристаллизованного белка 3vev.
Чтобы найти и другие примеры. в RSCB PDB использовала Advanced search с параметрами Experimental Method: X-Ray и Wild Type Protein - Include Expression Tags (yes) + Percent coverage of Uniprot Sequence (Any). Из находок я выбрала одну из первых – структуру бета-маннозидазы мыши 6DDT (PDB файл). Отличия между природной и кристаллизованной последовательностью и пропущенные остатки показаны на рисунке 6.

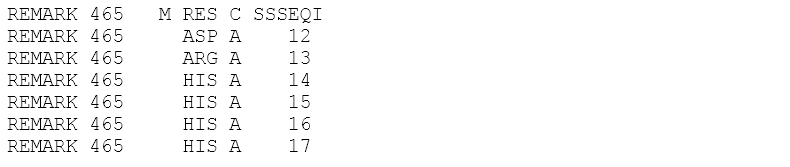
Рисунок 6. Различия последовательностей природного и кристаллизованного белка 6DDT.
Худший и лучший B-фактор в структуре 3vev
Чтобы определить лучший и худший В-фактор в структуре 3vev, я с помощью grep отсортировала строчки ATOM и HETATM, сохранила выдачу в текстовый файл и потом сделала Excel-таблицу, в которой отсортировала колонки по возрастанию В-фактора. Самый маленький В-фактор получился у атома N ARG155 = 6.95 (у этого атома occupancy = 0.5; для альтернативного положения N ARG155 B-фактор = 21.801). Самый большой В-фактор у атома С LYS458 (В-фактор = 117,24). Фрагменты файла PDB на рисунке 7.


Рисунок 7. Худший и лучший B-фактор в структуре 3vev. Нужная строчка подчеркнута, значение В-фактора взято в рамочку.
