Вторичная структра
Задача: определить вторичную структуру белка с программ DSSP или Stride.
Я взяла для работы ранее использованную структуру глюкокиназы 3vev. Файл PDB 3vev.
Вторичная структура белка была предсказана программой DSSP установленной на kodomo. Также вторичная структура была предсказана web-сервисом Stride. У web-сервиса Stride есть графическая выдача (рисунок 1).
Выдача DSSPВыдача Stride
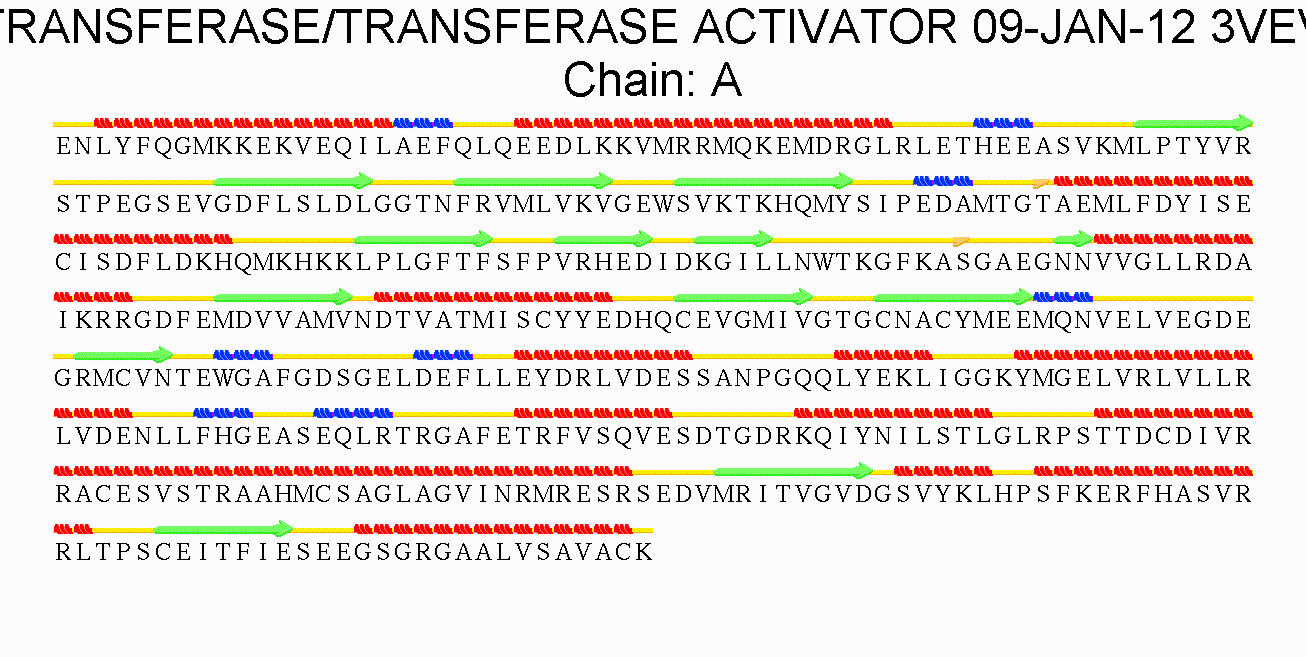

Рисунок 1. Выдача Stride в графическом виде.
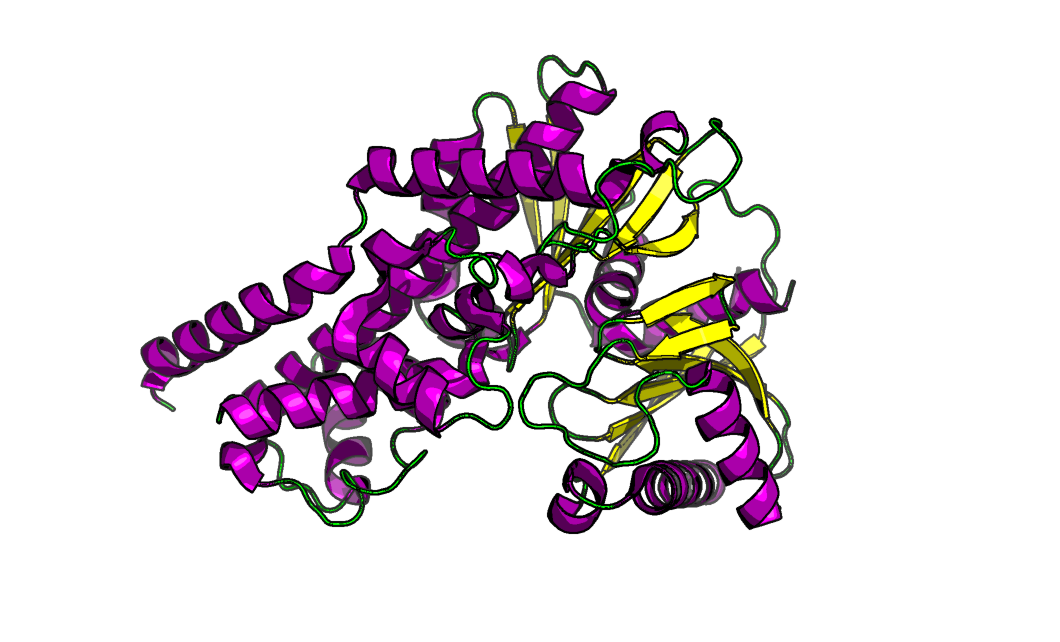
Рисунок 2. 3vev в виде cartoons с раскраской по элементам вторичной стуктуре. Фиолетовым – α-спирали, желтым – β-тяжи, зеленым – петли и повороты
Для анализа качества предсказания вторичной структуры программами DSSP и Stride я выбрала несколько элементов вторичной структуры, координаты я взяла из файла PDB из строк HELIX и SHEET. На рисунке 3 выбранные элементы визуализированы. В таблице 1 представлено сравнение границ выбранных элементов вторичной структуры, полученных программами, с границами, приведенными в заголовке PDB файла.
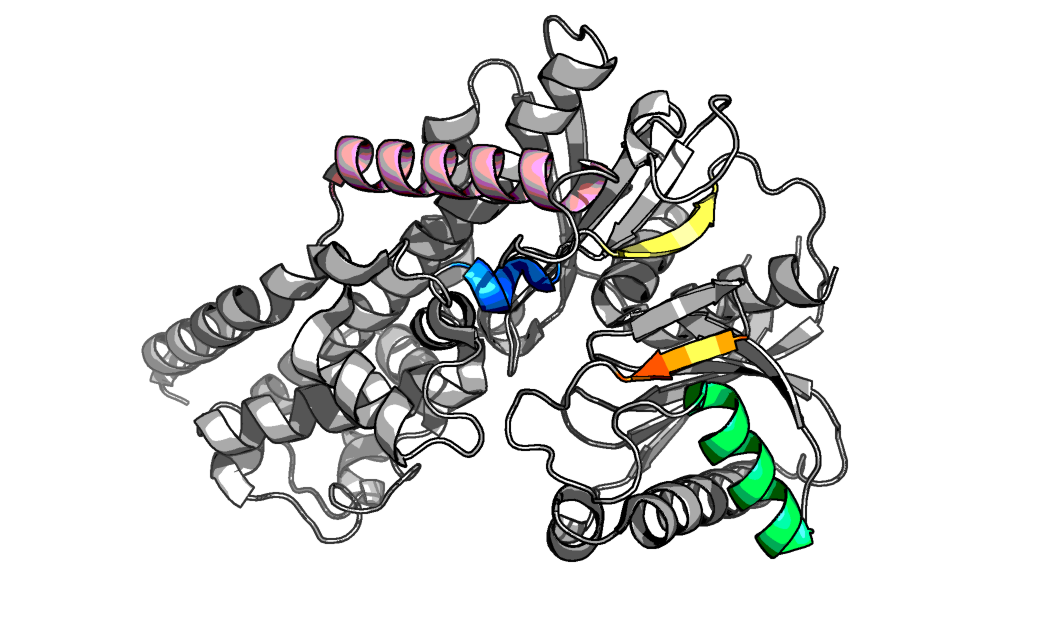
Рисунок 3. Элементы вторичной структуры, выбранные для анализа качества предсказания. α-спирали: h1 – розовым, h2 – зеленым, h3 – синим; β-тяжи: β1 – желтым , β2 – оранжевым.
Таблица 1. Сравнение границ элементов вторичной структуры, полученных программами Stride и DSSP с границами, приведенными в заголовке PDB файла.
| Элемент вторичной структуры (SSE) | PDB | Stride | DSSP |
|---|---|---|---|
| α-спираль 1 (h1) | GLN 26 - ARG 46 | GLU 27 - LEU 45 | GLU 27 - LEU 45 |
| α-спираль 2 (h2) | ASN 180 - GLY 193 | VAL 181 - ARG 192 | VAL 181 - ARG 192 |
| α-спираль 3 (h3) (*) | GLU 256 - PHE 260 | TRP 257 - ALA 259 | TRP 257 - ALA 259 |
| β-тяж 1 (&beta1) | LEU 58 - ARG 63 | LEU 58 A - ARG 63 | LEU 58 A - ARG 63 |
| β-тяж 2 (&beta2) | LYS 161 - LEU 164 | LYS 161 - LEU 164 | LYS 161 - LEU 164 |
как видно из таблицы 1, границы бета-тяжей совпадают, границы альфа-спиралей отличаются на 1 остаток. Возможно, различие в границах альфа спиралей связано с какими-то договоренностями, включать или не включать крайние остатки, то есть отличия в предсказании альфа-спиралей не существенно. Предсказания Stride и DSSP совпадают.
В таблице звездочкой отмечена h3 – это не альфа-спираль, а 3(10)-спираль. При визуализации в Pymol я не могу отличить друг от друга эти спирали, и Pymol красит все спирали одинаково. В PDB файле в строчках с 3(10)-спиралями стоит цифра 5 (рис. 4, подчеркнуто зеленым).
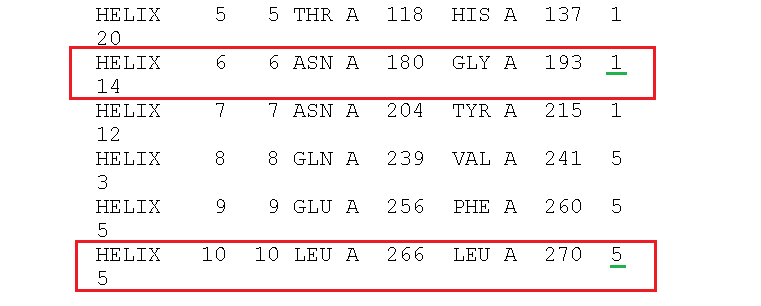
Рисунок 4. Разные спирали в файле PDB.
На самом деле, в 3vev довольно много 3(10)-спиралей. Это можно увидеть, например, на графической выдаче Stride (см. Рисунок 1). На рисунке 5 приведен фрагмент текстового файла выдачи Stride. В файле выдачи DSSP это менее удобно смотреть, но если все-таки посмотреть, видно, что предсказания совпадают (у 3(10)-спиралей однобуквенное обозначение G).
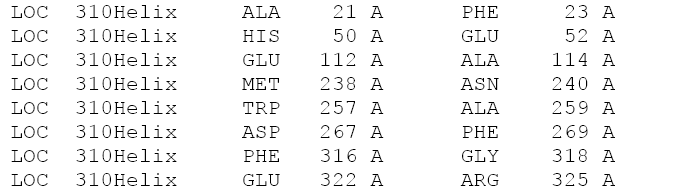
Рисунок 5. 3(10)-спирали в файле выдачи Stride.
Возможно, все 8 3(10)-спиралей не стоят внимания, но можно показать еще одну (помимо 3(10)-спирали, покрашенной синим цветом на рисунке 3).
При визуализации структуры 3vev в Pymol можно обратить внимание на странно изогнутый спиральный участок (на рисунке ниже, окрашено синим). При окрашивании по вторичной структуре Pymol окращивает остатки 266-280 как спираль (рис. 6А). На самом деле, это не одна кривая спираль, а 2, в чем можно убедиться посмотрев файл PDB (рис. 6В). 266-270 – это 3(10)-спираль и 271-280 – это α-спираль, на рисунке 6С они покрашены в фиолетовый и синий соответсвенно. Stride предсказывает их границы чуть-чуть по-другому: ASP 267 - PHE 269 + GLU 272 - SER 280
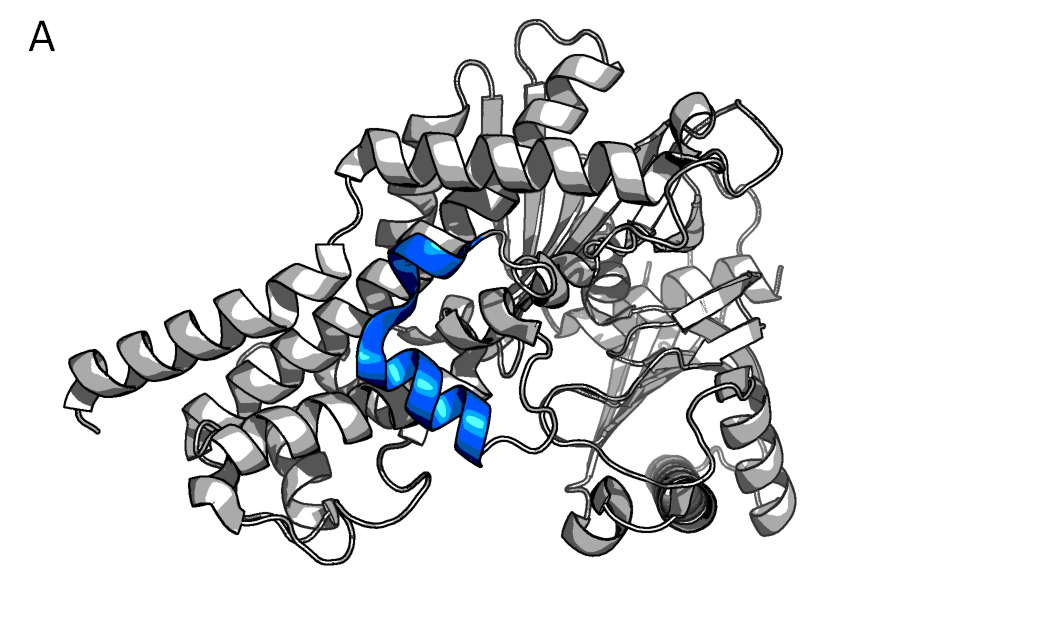

Рисунок 6.Участок 266-280. 3(10)-спираль + α-спираль.
Также из необычных элементов в структуре есть изолированные β-мостики. По предсказанию Stride – это остатки 118 и 174. DSSP помимо 118 и 174 предсказывает остаток 180, но Stride на этом месте предсказывает бета-тяж (хотя по виду он короткий и странный (см. рис.1). Остатки THR118 и SER174 расположены в структуре рядом, возможно, в растворе у них другое положение и они взаимодействуют друг с другом (рисунок 7).
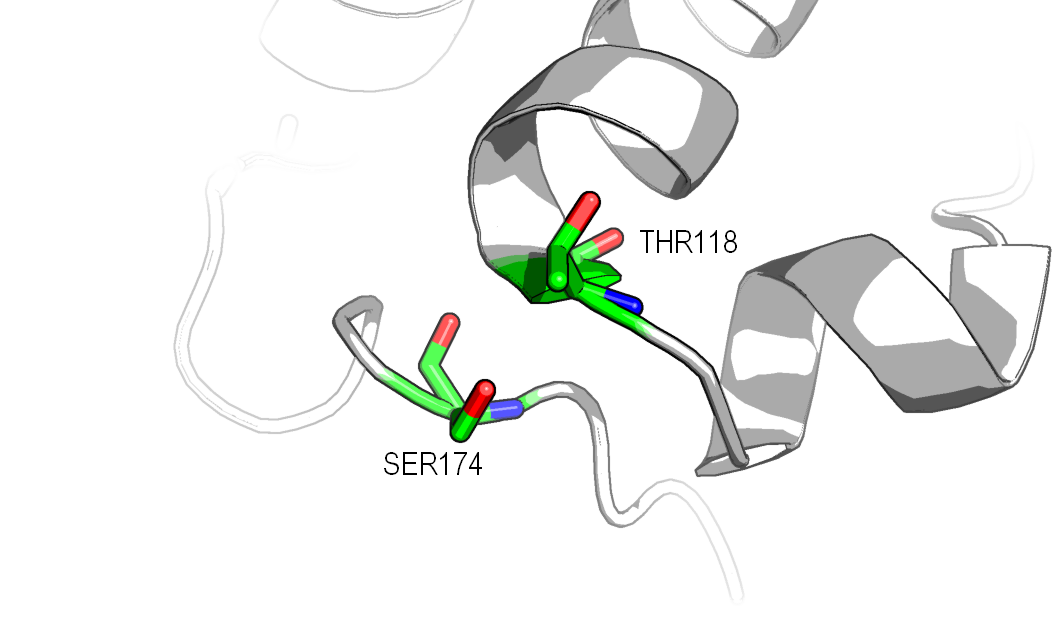
Рисунок 7. Остатки THR118 и SER174.
