Совмещение структур
Работали с белком 3vev из предыдущих практикумов.
С помощью PDBeFold нашли структурные гомологи белка. Выдача PDBeFold
Выбрали структуры разных белков с RMSD между 0,8 и 2,5 и длиной выравнивания более 50% от длины белка 3vev.- 4qs9; RMSD = 1.439; Nalign = 392;
Arabidopsis Hexokinase 1 (AtHXK1) mutant S177A structure in glucose-bound form - 4imx; RMSD = 1.084; Nalign = 436;
Crystal Structure of human glucokinase in complex with a synthetic activator - 3hm8; RMSD = 1.224; Nalign = 428;
Crystal structure of the C-terminal Hexokinase domain of human HK3 (в структуре 4 цепи) - 3o8m; RMSD = 1.615; Nalign = 404;
Crystal structure of monomeric KlHxk1 in crystal form XI with glucose bound (closed state)
Затем с помощью PDBeFold построили множественное структурное выравнивание (совмещение) выбранных последовательностей и скачали следующие файлы:
- Выравнивание последовательностей, построенное по структурам struct_align.fastq
- Файл с совмещёнными структурами send.rasmol
- Тот же файл в pdb, чтобы можно было открыть в PyMOL superposed.pdb
Полученное структурное выравнивание показано на рисунке 1.
Также в программе Jalview построили множественное выравнивание последовательностей сервисом Muscle с параметрами по умолчанию. Результат на рисунке 2. Файл с выравниванием.
В файле со структурным выравниванием, хотя я, вроде, выбирала только цепь А, оказалась более длинная последовательность, поэтому выравнивание я обрезала слева и справа, так чтобы два выравнивания было удобнее сравнивать.

Рисунок 1. Структурное выравнивание последовательностей построенное PDBeFold. Раскрашивание Clustal.
height='210px'
Рисунок 2.Множественное выравнивание последовательностей, построенное сервисом Muscle с параметрами по умолчанию. Раскрашивание Clustal.
Выравнивнивания сделанные программами Muscle и PDBeFold почти одинаковые, но на некоторых участках выравнивания все-же различаются (например на участках 90-110, 120-150, 450-470 – нумерация по структурному выравниванию). Особенно много различающихся колонок на участке 320-390 (нумерация по структурному выравниванию). Как это выглядит в Jalview показано на рисунке 3. На рисунке 3А - структурное выравнивание, 3В - множественное выравнивание Muscle.
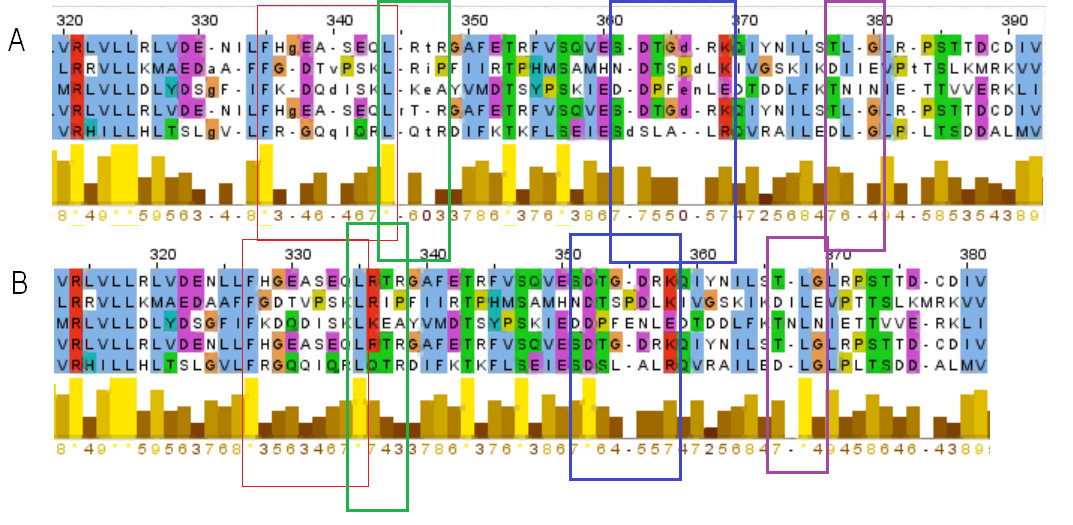
Рисунок 3. Различающиеся колонки на одном из участков выравнивания. На рисунке 3А - структурное выравнивание, 3В - множественное выравнивание Muscle.
Рассмотрим для некоторых участков совмещение структур в PyMOL, чтобы понять, какое выравнивание правильнее.
Вот например, на участке 120-150 различается несколько колонок (рисунок 3). Я визуализироала остаток Asp на позизиции 137 структуры 3vev и остатки, которые с ним совмещаются. Эти остатки входят в альфа-спираль (хотя расположены близко к краю альфа-спирали, но все-таки в нее, наверное, входят) и соседние с ними тоже (рисункок 4А, В). Остаток Аla структуры 4qs9 совмещается с соответствующими остатками других последовательностей. На этом участке структурное выравнивание правильнее.
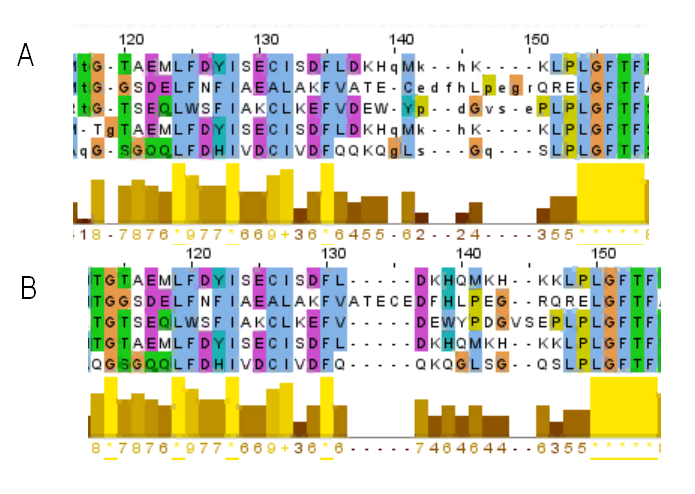
Рисунок 3. Различающиеся колонки на участке 120-150 выравнивания. На рисунке 3А - структурное выравнивание, 3В - множественное выравнивание Muscle. Обратить внимание на остаток Asp (позиция 137)
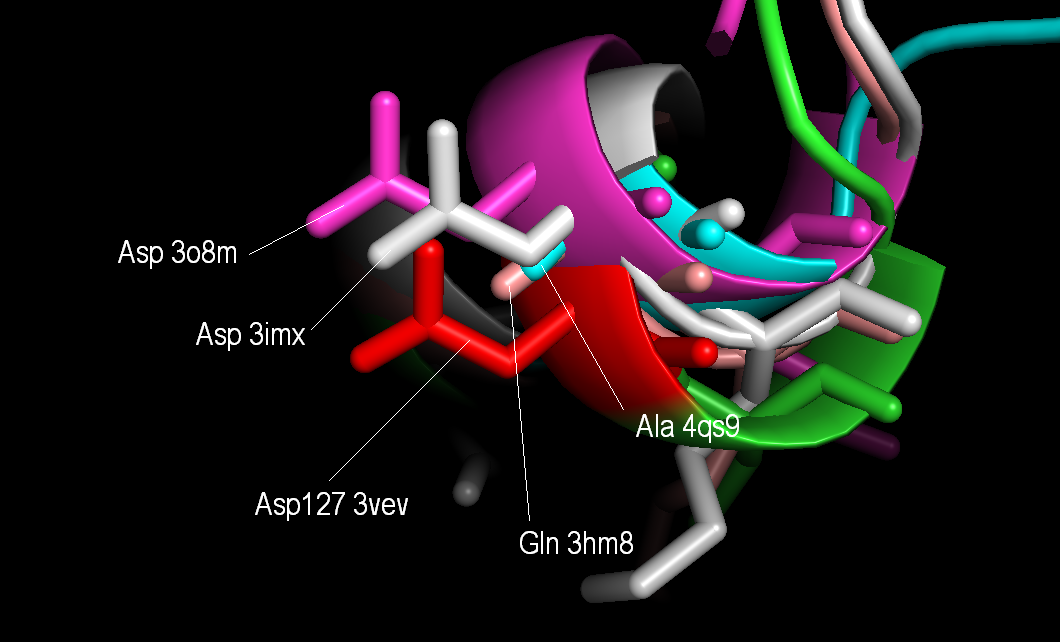
Рисунок 4А. Совмещение структур. Остаток Asp127 3vev и совмещающиеся с ним остатки.
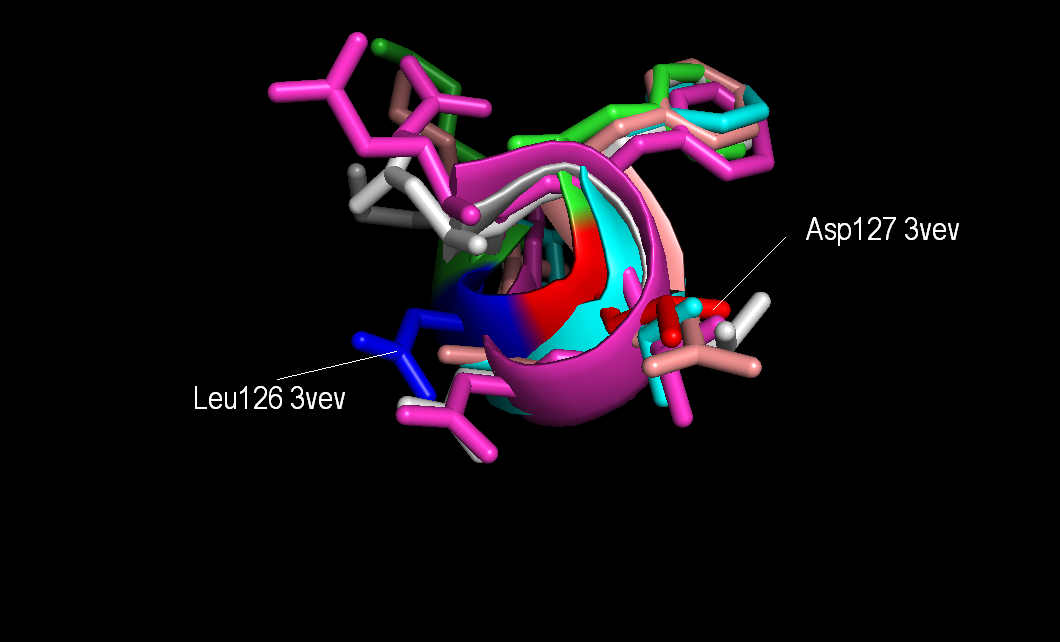
Рисунок 4В. Совмещение структур. Соседние остаток Asp127 3vev тоже совмещаются.
Рссмотрим еще один случай – участок 360-370. Нас интересуют колонки в структурном выравнивании 361-364, соответсвующие колонки в множественном выравнивании 351-353 отличаются (рисунок 5). Визуализировав это место мы видим, что там петля и, скорее всего, структуры совмещаются плохо (рисунок 6 А,В) из-за подвижности петли. В данном случае убедительнее выглядит множественное выравнивание программой Muscle, а не совмещение структур, потому что в множественном выравнивании можно выдели блок (позиции 345-353).
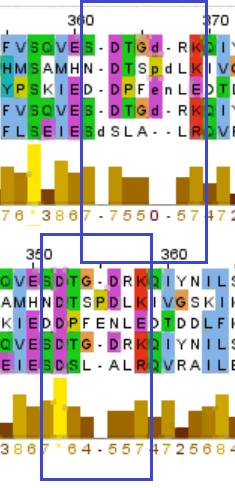
Рисунок 5. Различающиеся колонки на участке выравнивания 360-370. На рисунке 3А - структурное выравнивание, 3В - множественное выравнивание Muscle.
.png)
Рисунок 6А. Остаток аспартата 3vev (покрашен красным) совмещается с остатками Asp 3х структур, кроме 3hm8 (петля, в которую входит данный Asp покрашена синим).
.png)
Рисунок 6В. Соседний остаток Thr334 3vev (покрашен красным) также хорошо совмещается с остатками других структур, кроме Leu 3hm8.
Таким образом, на участках, где наблюдается вторичная структура совмещению структур (множественному структурному выравнивнию) можно доверять – оно в данном случае достовернее. На неупорядоченных подвижных участках (петлях, поворотах) мне кажется более правильным смотреть на обычное множественное выравнивание последовательностей.
