Поиск по сходству. BLAST, E-value
Проверка гомологичности белков, найденных поиском по сходству
Для моего белка 3-оксоакцил-[АПБ]-синтаза (BAC70085.1) в BLAST были найдены похожие белки, однако у всех процент абсолютно консервативных белков был в пределах 20-30, что не очень хорошо для качественного анализа. Поэтому для выполнения этого задания я использовала белок АДФ-рибоза пирофосфотаза (AMA73680.1). С помощью BLSAST (параметры поиска: E-value = 10, WordSize = 6) из базы данных SwissProt были найдены белки, схожие с белком АДФ-рибоза пирофосфотаза. В табл.1 приведены параметры данных белков.На рис.1 можно увидеть множественное выравнивание белков (Ссылка на файл с выравниванием), с помощью готорого я определяла гомологию их белку АДФ-рибоза пирофосфотаза. Для этого я нашла и выделила блоки достоверного выравнивания, которые содержат достаточное число консервативных позиции для всех белков, входящих в блок, и не содержат гэпы.
Первый белок (Q8KP10.1) входит в 6 блоков, а процент консервативных позиций более 50%, поэтому, скорее всего, он гомологичен моему белку.Второй белок (A9MHR4.2) входит во все 8 блоков, что говорит о его гомологичности (к тому же данный белок имеет еще и очень малое значение E-value).
Третий (Q83K58.1) и шестой (Q74SF4.1) также входят в 8 блоков, имеют E-value меньше 1е-10, и, вероятно, они тоже гомологичны моему белку.
А вот четвертый (Q9UKK9.1) и пятый (Q6D8X4.1) входят только в 4 и 6 блока соответственно, однако, при таком низком E-value, этого оказывается вполне достаточно для гомологичности белку АДФ-рибоза пирофосфотаза.
Седьмой (P45799.1), восьмой (Q65217.1) и девятый (A9BP66.1) вошли только в один блок, значит, все эти три белка не гомологичны моему (к тому же восьмой и девятый имеют высокий E-value).
Рис.1. Множественное выравнивание белков, найденных поиском по свойству
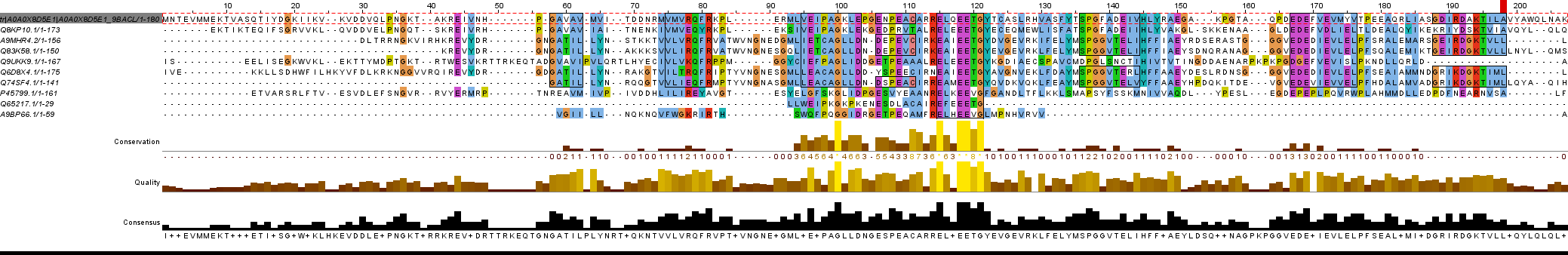
Табл.1. Параметры белков.
| ID/AC | Название белка | Coverage, % | Identity % | E-value | Гомология белку AMA73680.1 |
| Q8KP10.1 | Methanol dehydrogenase activator | 95 | 56 | 3е-66 | да |
| A9MHR4.2 | GDP-mannose pyrophosphatase NudK | 80 | 36 | 3е-19 | да |
| Q83K58.1 | GDP-mannose pyrophosphatase NudK | 78 | 34 | 9е-18 | да |
| Q9UKK9.1 | ADP-sugar pyrophosphatase | 82 | 33 | 1е-14 | да |
| Q6D8X4.1 | GDP-mannose pyrophosphatase NudK | 92 | 30 | 1е-13 | да |
| Q74SF4.1 | GDP-mannose pyrophosphatase NudK | 73 | 32 | 1е-10 | да |
| P45799.1 | ADP compounds hydrolase NudE | 88 | 22 | 8е-07 | нет |
| Q65217.1 | mRNA-decapping protein g5R | 16 | 55 | 0.21 | нет |
| A9BP66.1 | RNA pyrophosphohydrolase | 35 | 29 | 2.6 | нет |
Описание крупных перестроек между парой белков, имеющих гомологичные участки (домены)
Для рассмотрения перестроек я взяла следующие последовательности: VPseudogymnoascus sp. VKM F-4515 (FW-2607) (KFY60746.1) и Hebeloma cylindrosporum h7 (KIM47460.1). Эти белки, имеющие гомологичные домены, я искала с помощью Pfam. Для этого я открыла архитектуры последовательностей семейства ZZ и по схеме, на которой изображены домены, нашла белки с интересной архитектурой. Затем я построила карту локального сходства (рис.2) в BLAST, с параметрами E-value = 1.0E-5 и длиной слова, равной двум.Рис.2. Карта локального сходства
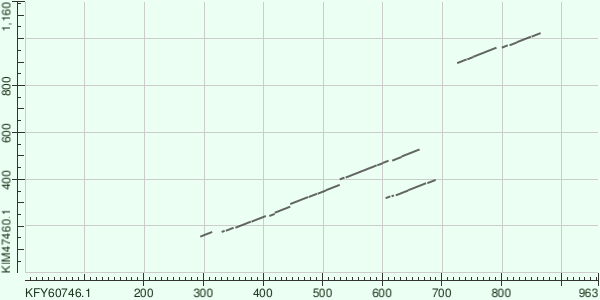
Далее я обозначила гомологичные участки одинаковыми буквами, а негомологичные - разными (рис.3). Теперь более подробно разберем крупные эволюционные перестройки. Для удобства обозначим последовательность KFY60746.1 (расположена по горизонтали) цифрой 1, а KIM47460.1 (по вертикали) цирфой 2.
Вначале последовательности не имеют гомологии, поэтому обозначены разными буквами. Далее идут два гомологичных участка - A и B. Затем в последовательности 1 идут участки С и В, которым есть гомологи в последовательности 2, в которой, однако, между В и С есть маленький участок Е. В 1 последовательности этот участок идет после C и B, и можно предположить, что в какой-то из этих последовательностей произошла транслокация участка Е. Далее идут негомологичные участки, обозначенные V и F в последовательностях 1 и 2 соответственно. И затем следуeт последний гомологичный участок - D, после которого гомология снова заканчивается. Теперь вернемся к участку В. Как объяснить наличие в обеих последователностях по два одинаковых участка, разделенных еще одним? Скорее всего, у общего предка произошла дупликация участка В, и вставка между ними участка С (или С и Е, так как мы не может определить, где изначально находился участок Е).
В итоге последовательности получились такими:
1). RABCBEVDU
2). QABECBFDI
Рис.3. Карта локального сходства с отмеченными учатсками гомологии
