Пространственная структура β-лактамазы бактерии Oceanobacillus iheyensis
С помощью программы jmol была построена трёхмерная структура галотолерантной β-лактамазы бактери Oceanobacillus iheyensis (идентификатор в базе данных PDB - 3LEZ). Трёхмерная структура сторится на основе информации о координатах атомов и том, какие атомы связаны друг с другом, которая берётся из соответствующего файла (в данном случае это 3LEZ.pdb). Кроме информации о координатах в этом файле есть также информация, которая описывает некоторые особенности белка. Эта информация представлена в блоке ниже.
TITLE CRYSTAL STRUCTURE OF A HALOTOLERANT BACTERIAL BETA-LACTAMASE
SOURCE 2 ORGANISM_SCIENTIFIC: OCEANOBACILLUS IHEYENSIS;
COMPND MOL_ID: 1;
COMPND 2 MOLECULE: BETA-LACTAMASE;
COMPND 3 CHAIN: A;
COMPND 4 FRAGMENT: MATURE BETA-LACTAMASE;
COMPND 5 EC: 3.5.2.6;
COMPND 6 ENGINEERED: YES
HETNAM CA CALCIUM ION
HETNAM CL CHLORIDE ION
HETNAM EPE 4-(2-HYDROXYETHYL)-1-PIPERAZINE ETHANESULFONIC ACID
FORMUL 2 CA 8(CA 2+)
FORMUL 10 CL CL 1-
FORMUL 11 EPE C8 H18 N2 O4 S
FORMUL 12 HOH *515(H2 O)
EXPDTA X-RAY DIFFRACTION
REMARK 2
REMARK 2 RESOLUTION. 1.25 ANGSTROMS.
Белок состоит из одной цепи. С белком связаны 8 ионов Ca2+, 1 ион Cl- и 1 молекула EPE (4-(2-гидроксиметил-1-пиперазин этансерная кислота). А также 515 молекул воды. Данные были получины с использованием метода рентгеноструктурного анализа с разрешением в 1,25 ангстрем.
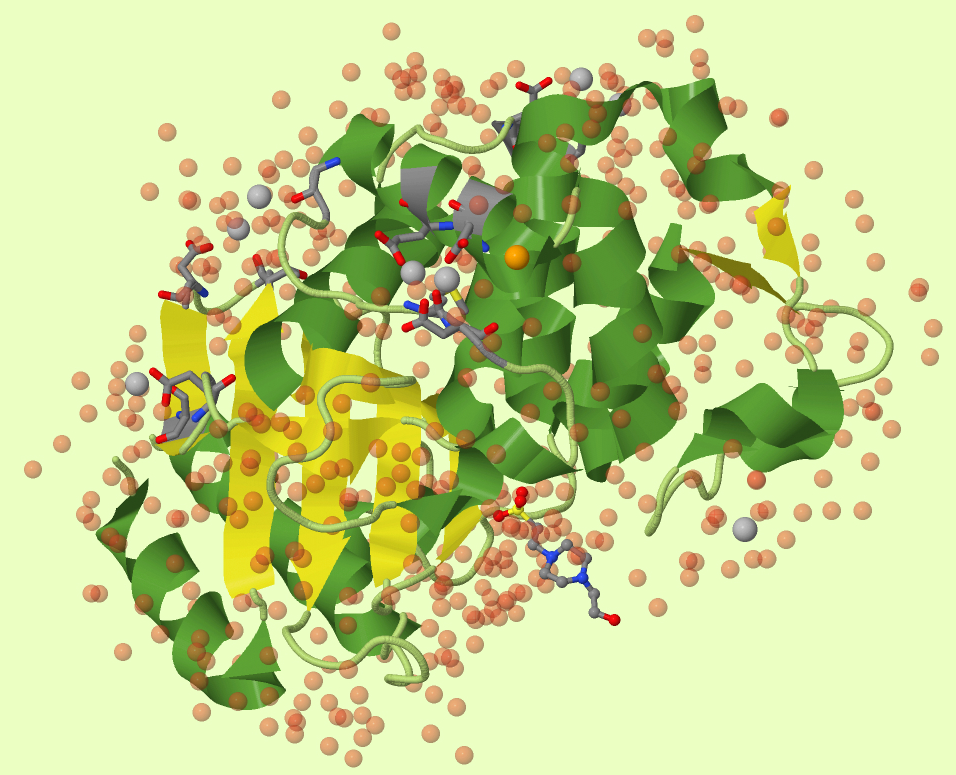
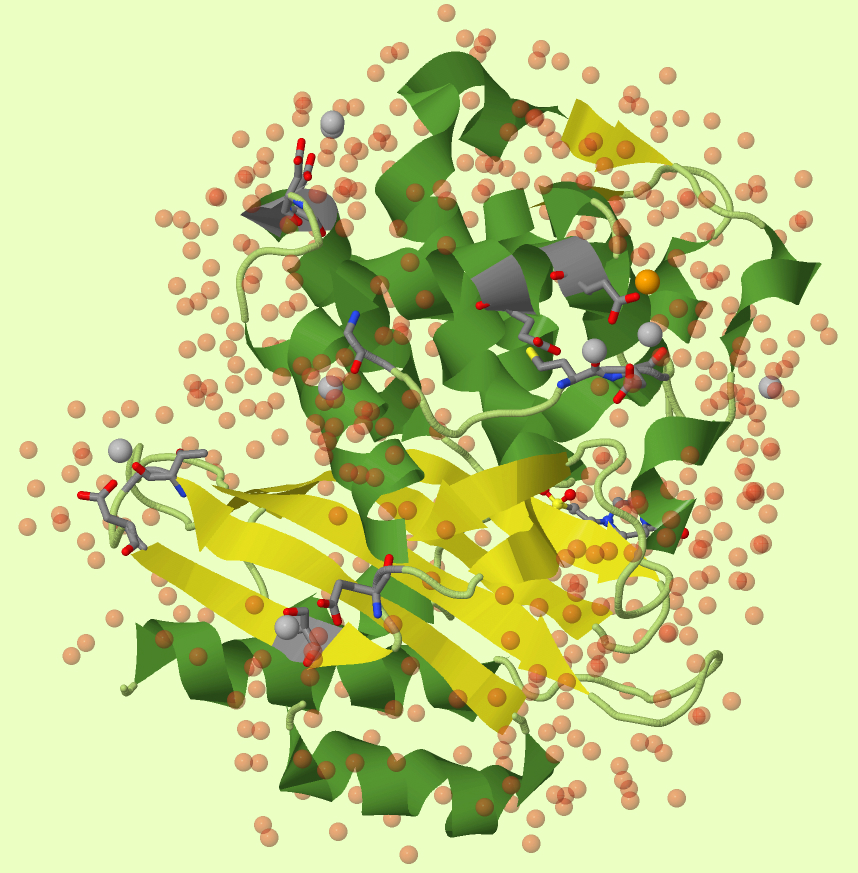
С помощью программы jmol у меня получилось создать скрипт, с помощью которого было получено следущее изображение. На рисунке 1 и рисунке 3 видно, что аминокислотная последовательность, образующая β-складчатые слои образует единый β-лист, пространственно отделённый от α-спиралей следовательно этот белок можно отнести к α/β - белкам. Используя скрипт, написанный на языке python у меня получилось выделить те аминокислоные остатки, чьи атомы учавствуют в связи с лигандами (свзяи записаны в pdb-файле под квалификатором "CONECT"), эти аминокислотные остатки показаны в виде "проволоки" (wireframe). Видно, что Ca связывают аминокислотные остатки, содержащие кислород, в большей степени - кислые аминокислоты (Аспарагиновая и Глутаминовая).
|
|
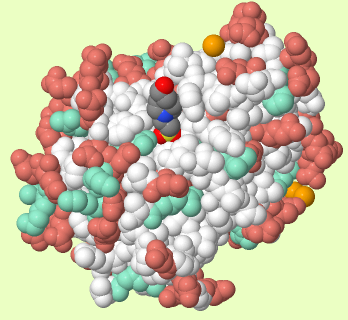
А с помощью этого скрипта можно посмотреть на расределение заряженных аминокислот по поверхности белка. Из рисунка 2, можно заключить, что больше всего на поверхности белка отрицательно-заряженных аминокислотных остатков. Что и обуславливает связывание с ионами Ca. Это подтверждается и тем, что ионы Ca располагаются над участками с высокой концентрацией отричательного заряда. Ион Cl также располагается рядом с отрицательным зарядом, что не характерно для орицательно заряженных ионов. Молекула же EPE находится в нейтральной "ямке" в белке.
|
|
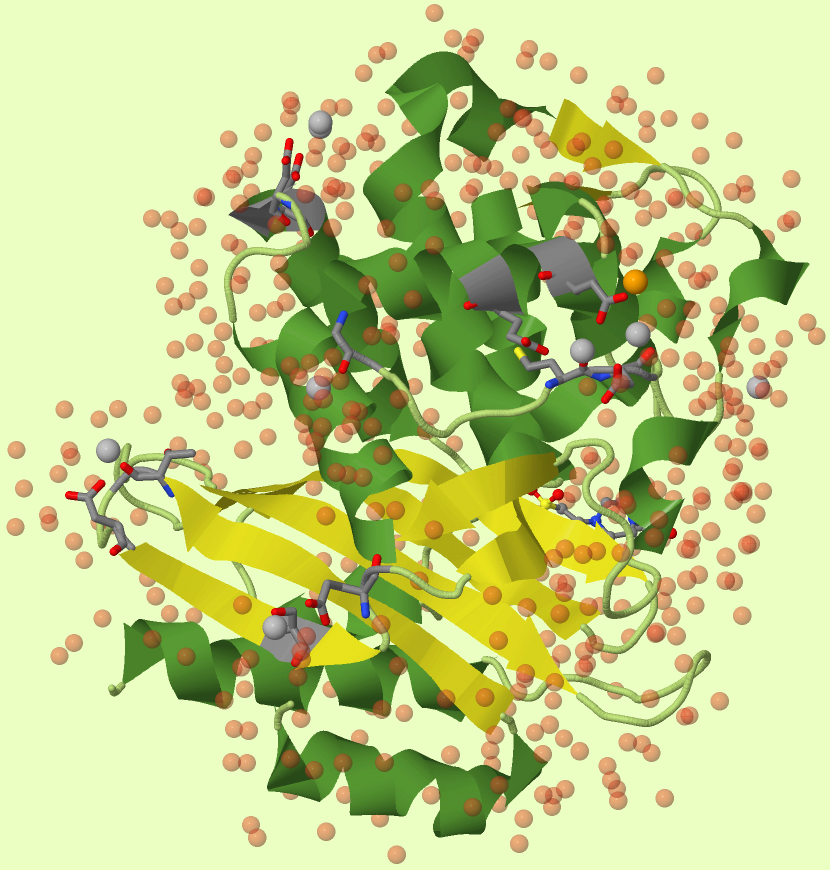
Раскрасив белок градиентом от N к C (как на рисунке 3) концу можно проследить то, как расположены относительно друг-друга элементы α-спиралей и β-листов. Для создания jmol-скрипта для градиентной окраски я использовал тот же скрипт, написанныйна языке python. (он многофункциональный и используется в течении всего блока о этом белке)
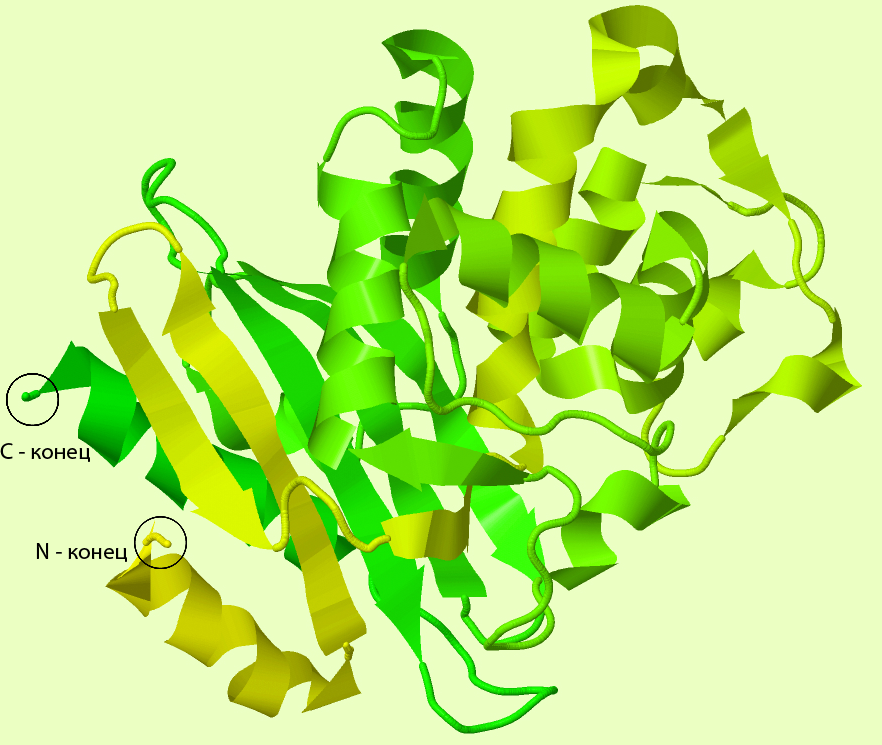 Рис.3. Градиентная раскраска белка. Изменение цвета от жёлтого к зелёному соответствует движеню от N-конца белковой цепи к C-концу.
Рис.3. Градиентная раскраска белка. Изменение цвета от жёлтого к зелёному соответствует движеню от N-конца белковой цепи к C-концу.
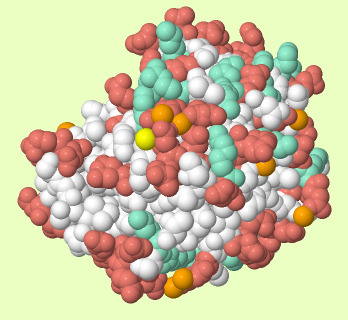
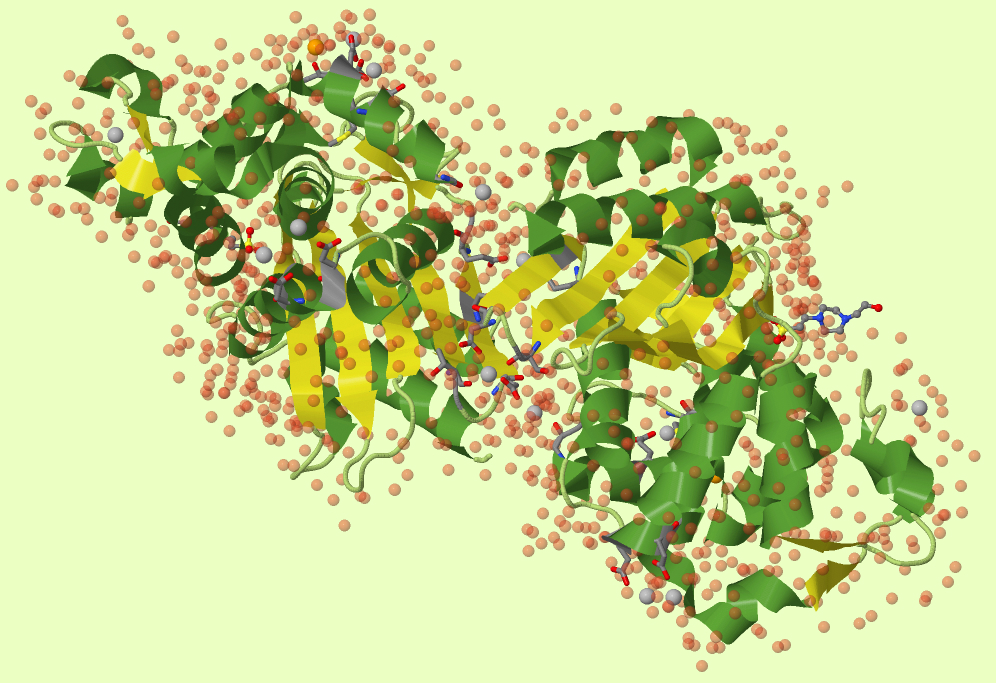
В базе данных PDB белок представлен не только тем файлом, с которым сейчас иёдт работа. В живых системах белки часто образуют димеры, тримеры и так далее, или в условиях клетки имеют немного другое расположение каких-то аминокислотных остатков. Такие структуры предлагаются как Biological Assembly. Для β-лактамазы показано две такие структуры. Как видно из рисунка 4 и рисунка 1 первая биологическая еденица внешне не отличается от исходной, а вторая - это димер, скорее всего это как раз работающая форма фермента, а активный центр находится между двумя половинками - между β-листами.
|
|
Если сравнивать аминокислотную последовательность из базы данных NCBI и PDB, то окажется, что последовательность из базы данных NCBI на 44 аминокислотных остатка длиньше, чем последовательность из базы данных PDB. Это объясняется тем, что в NCBI последовательность строится на основе гена, а в PDB на основе уже "окончательной" (прошедшей постранляционную модификацию последовательность) последовательности. Эта последоваттельность в 44 аминокислотных остатка нужна это белку для того, чтобы транспортировать его из цитоплазмы бактерии, в процессе переноса эта последовательность отрезается от белка [1].

- Использованная литература:
- [1] - An antibiotic-resistance enzyme from a deep-sea bacterium