Множественное выравнивание
Выравнивание последовательностей - это вставка "гэпов" - пробелов в оследовательности так, чтобы одинаковые участки последовательностей оказались друг под другом. Это позволяет увидеть общие мотивы для них. При множественном выравнивании мы сравниваем сразу много последовательностей и это позволяет выделить в них консервативные участки, то есть встречающиеся в каждой или почти каждой последовательности в неизменном виде. По ним мы можем судить о гомологии последовательностей.
В данном практикуме для построения выравниваний использовалась программа JalViev. Для работы я взял последовательности из файла aln17_1.fa. После построения обработки в JalView этих 19 последовательностей было полуено выравнивание, представленное на рисунке 1.
По этому выравниванию можно судить о степени гомологичности последовательностей белков. На основе этой гомологии можно построить филогенетическое дерево, показывающее степень родства последовательностей. такое дерево представлено на рисунке 2. Дерево строится на основе того, насколько сходны последовательности, так как все они имели общего предка, то у них сохранились некоторые консервативные блоки, которые практически не претерпели изменений и сходны во всех последовательностях (увидеть эти блоки можно наведя курсор на картинке на "гомологичные последовательности"). В каждой строчек есть позиции, на которой всегда стоит строго определённая буква. несколько таких позиций. Несколько таких концервативных позиуий рядом друг с другоммогут быть прерваны какой-то неконсервативной частью, но, скорее всего, это гомлогичные аминокислоты (то есть имеют общее проихождение). Напротив, если выбрать пару родственных последовательностей, но которые по "родственности" стоит далеко от других, то можно увидеть, что их последовательности очень похожи, но они сильно отличаются от других последовательностей. На таких участках часто бывают "гэпы", и совпадение букв является случайным. На рисунке 1 первые две последовательности являются родственными и очень сходными, но на выделенном участке они сильно отличаются от других строк.
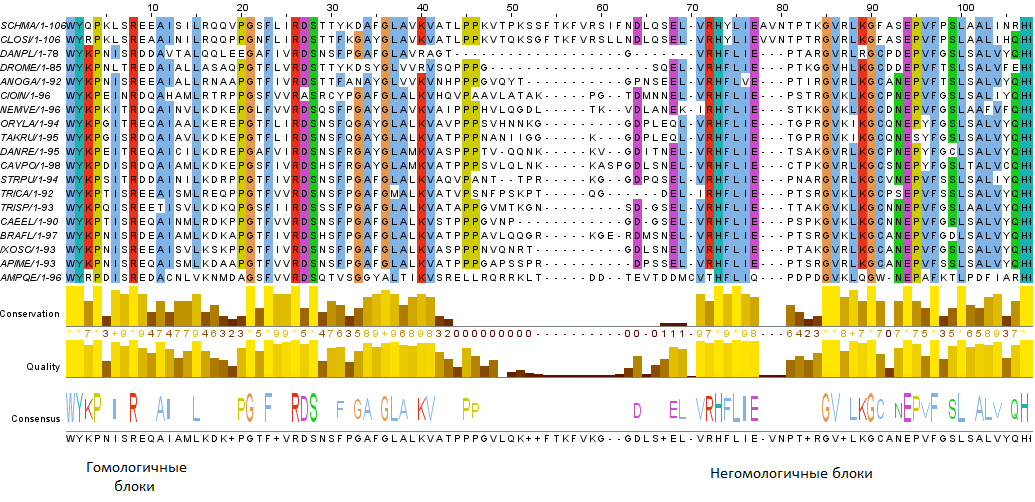 Рис. 1.Множественное выравнивание. Окраска - ClustalX, консервативность - 70%.
Рис. 1.Множественное выравнивание. Окраска - ClustalX, консервативность - 70%.
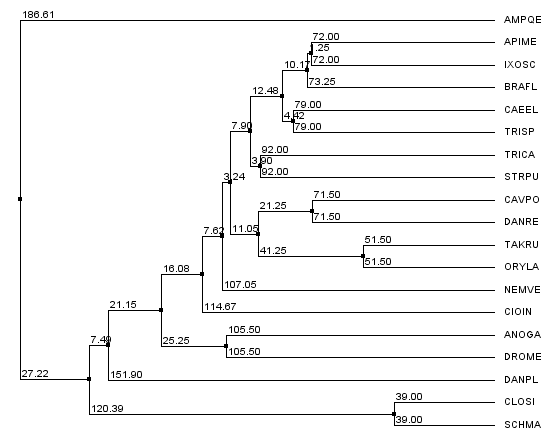 Рис. 2.Филогенетическое древо последовательностей.
Рис. 2.Филогенетическое древо последовательностей.
Для следующего выполнения следующего задания был взят первый гомологичный блок, сстоящий из 41 буквы. Полученные данные представлены в таблице 1.
| Параметр | Число позиций | % |
| Абсолютно консервативные позиции | 8 | 19,51 |
| Консервативные на 70% | 23 | 56,09 |
| Функционально консервативные позиции | 8 | 19,51 |
| Функционально консервативные на 70 % | 23 | 56,09 |
Далее нужно было рассмотреть участок между недалеко расположенными консервативными позициями, содержащий "гэпы". Такой блок в данном выравнивании единственный - с 75 по 88 позицию (содержит 14 букв, включая концевые консервативные пизиции). Этот блок предствален на рисунке 3. Всего на этом участке 51 "гэп". по три в каждой строчке, исключая первые 2. Это связано с тем, что первые две последовательности, как говорилось ранее, эволюционно находятся далеко от других последовательностей и близки друг с другом. В процессе эволюции, скорее всего, в месте между этими консервативными позициями либо произошла вставка трёх лишник аминокислот, либо делеция. Процент гэпов на данном участке (не включая консервативыне концы) получается равным 38,35%.
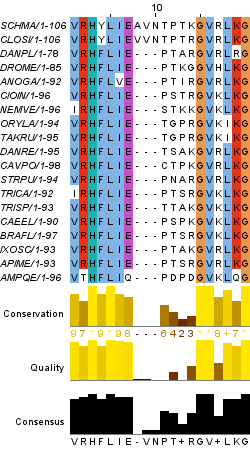 Рис. 3.Участок выравнивания, содержащий "гэпы"
Рис. 3.Участок выравнивания, содержащий "гэпы"
Далее нужно было сделать выравнивание ещё одной последовательности относительно данного множественного выравнивания вручную. результат представлен на рисунке 4.
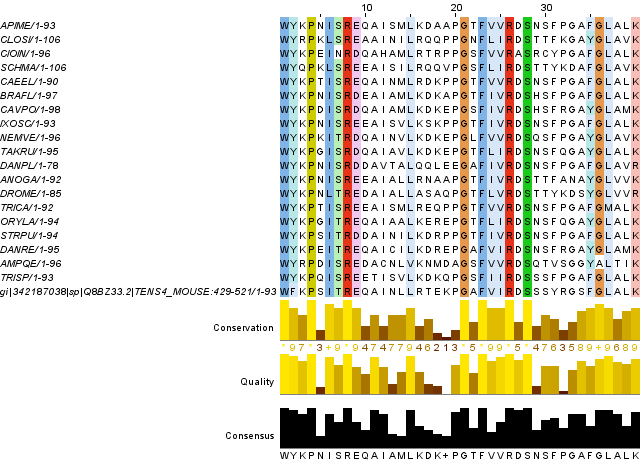
Консенсусная последовательность первого гомологического блока это:
>Consensus/1-41 Percentage Identity Consensus
WYKPNISREQAIAMLKDKEPGTFVVRDSNSFPGAFGLALK'LOGO' для данного блока представлено на рисунке 5

Для следующего задания мною были выбраны 7 белков со следующими идентификаторами из базы данных PDB: "2CX1; 3ATQ; 4JBI; 1SFX; 3NTU; 3CMB; 1DO6". В результате получилось выравнивание, совпадающие только в одной позиции, и которое является, вероятнее всего, случайным. Этот блок приведён на рисунке 6.
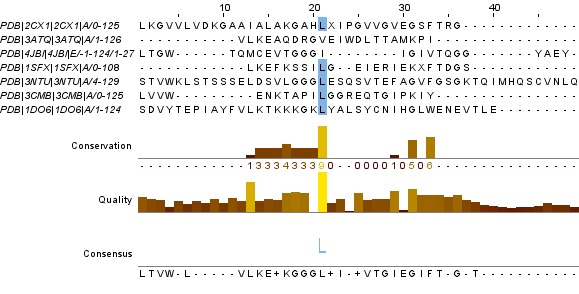 Рис. 6.Выравнивание случайных последовательностей белков.
Рис. 6.Выравнивание случайных последовательностей белков.
Также вы можете скачать мой рабочий файл в формате .jar .