BLAST
BLAST - это програма, предназначенная для поиска гомологичных последовательностей. Я использовал её, чтобы найти гомологов белка, имеющиего код доступа в базе данных Uniprot - Q8EMP8 и следущую последовательность.
Из полученных последовательностей я выбрал последовательно с кодом доступа P14559.1. Информацию о ней можно увидеть в таблице 1.
Таблица 1. Информация о найденном белке-гомологе.| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Flags: Precursor [Streptomyces albus G] | 195 | 195 | 83% | 3,00e-58 | 40% | P14559.1 |
Поле "Description" содержит название белка, поле "Max score" - это максимальный вес выравнивания фрагмента, если фрагмент один, то он численно совпадает с суммарным весом выравнивания - "Total score". "Query cover" - это процент покрытыия данного белка найденным белков. "E value" показывает вероятность ошибки, нахождения как гомолога случайной последовательности. "Ident" - это процент сходных аминокислотных остатков, отражает сходство последовательностей. "Accession" - код доступа данного белка.
дальше нужно было построить карту локального сходства для этих двух белков. Выравнивание этих белков можно увидеть в этом файле. Полученная карта локального сходства показана на рисунке 1. Как видно из полученной карты, эти беки являются практически идентичными, что подтверждает то, что у этих белков одна и та же функция - β-лактамазная активность, то есть, что, фактически, означает устойчивость к атнибиотикам, вроде пенициллина. Второй белок была найден в бактерии Streptomyces albus.
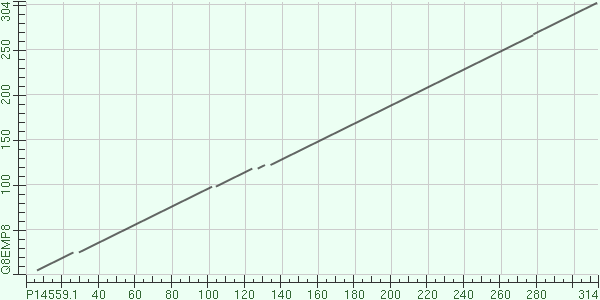 Рисунок 1. Карта локального сходства двух белков.
Рисунок 1. Карта локального сходства двух белков.
Для поиска гомологов данного белка у эукариотических организмов в поле "organism" было поставлено "eukaryotes (taxid:2759)", и по данному запросу было найдено 2 белка, информация о них представлена в таблице 2. Но, как видно из этой таблицы, эти белки имеют маленькую схожесть с белков β-лактамазой, имеют очень высокий E-value, а исходя из названий, один белок является хеликазой, а другой относится к семейству иммуноглобулинов. То есть их функция сильно отличается от функции β-лактамазы, так что эти находки, вероятнее всего случайны. Что не удивительно, так как белки, лдающие устойчивость к пенициллину эукариотическим организмам не нужны и в них не присутствуют. Поэтому я решил провести поиск по прокариотическим организмам, но в таксоне Bacillus (запрос - "Bacillus/Clostridium group (taxid:1239)"). Из полученных результатов я выбрал те, у которых E-value , 0,001. Эти белки представлены в таблице 3.
Таблица 2. Результаты поиска гомологичных белков в эукариотических организмов.| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| Select seq sp|O70133.2|DHX9_MOUSE RecName: Full=ATP-dependent RNA helicase A; Short=RHA; AltName: Full=DEAH box protein 9; Short=mHEL-5; AltName: Full=Nuclear DNA helicase II; Short=NDH II [Mus musculus] | 31.2 | 31.2 | 32% | 5.3 | 25% | O70133.2 |
| Select seq sp|Q9P232.3|CNTN3_HUMAN RecName: Full=Contactin-3; AltName: Full=Brain-derived immunoglobulin superfamily protein 1; Short=BIG-1; AltName: Full=Plasmacytoma-associated neuronal glycoprotein; Flags: Precursor [Homo sapiens] | 31.2 | 31.2 | 31% | 6.2 | 25% | Q9P232.3 |
| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| Select seq sp|P39824.2|BLAC_BACSU RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Flags: Precursor [Bacillus subtilis subsp. subtilis str. 168] | 323 | 323 | 96% | 6,00e-109 | 54% | P39824.2 |
| Select seq sp|Q45726.1|BLAC_BACTU RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Flags: Precursor [Bacillus thuringiensis] | 322 | 322 | 98% | 1,00e-108 | 51% | Q45726.1 |
| Select seq sp|P28018.1|BLA1_BACMY RecName: Full=Beta-lactamase 1; AltName: Full=Beta-lactamase I; AltName: Full=Penicillinase; Flags: Precursor [Bacillus mycoides] | 320 | 320 | 98% | 4,00e-108 | 51% | P28018.1 |
| Select seq sp|Q44674.1|BLAC_BACAM RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Flags: Precursor [Bacillus amyloliquefaciens] | 319 | 319 | 84% | 2,00e-107 | 58% | Q44674.1 |
| Select seq sp|P10424.1|BLA1_BACCE RecName: Full=Beta-lactamase 1; AltName: Full=Beta-lactamase I; AltName: Full=Penicillinase; Flags: Precursor [Bacillus cereus] | 318 | 318 | 98% | 3,00e-107 | 51% | P10424.1 |
| Select seq sp|P00808.1|BLAC_BACLI RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Contains: RecName: Full=Large exopenicillinase; Contains: RecName: Full=Small exopenicillinase; Flags: Precursor [Bacillus licheniformis] | 315 | 315 | 98% | 5,00e-106 | 54% | P00808.1 |
| Select seq sp|P06548.1|BLA3_BACCE RecName: Full=Beta-lactamase 3; AltName: Full=Beta-lactamase III; Flags: Precursor [Bacillus cereus] | 314 | 314 | 96% | 2,00e-105 | 51% | P06548.1 |
| Select seq sp|P00809.1|BLAC_BACCE RecName: Full=Beta-lactamase 1; AltName: Full=Beta-lactamase I; AltName: Full=Penicillinase; Flags: Precursor [Bacillus cereus] | 310 | 310 | 97% | 6,00e-104 | 50% | P00809.1 |
| Select seq sp|P00807.1|BLAC_STAAU RecName: Full=Beta-lactamase; AltName: Full=Penicillinase; Flags: Precursor [Staphylococcus aureus] | 202 | 202 | 96% | 2,00e-62 | 34% | P00807.1 |
Эти послеовательности были выравнены, результат выравнивания представлен на рисунке 2. При это абсолютно консервативных колонок 23,8%, функционально - 38,3%. По этому ссылке можно скачать файл проекта с этим выравниванием. Файл с выравниванием в формате .fa можно скачать по этой ссылке.
