Введение в филогенетические деревья
Для выполнения этого практикума были взяты следующие 8 видов бактерий, принадлежащих таксону cellular organisms/Bacteria/Firmicutes:
| Название | Мнемоника | Филогения |
| Bacillus anthracis | BACAN | Bacilli/Bacillales/Bacillaceae/Bacillus/Bacillus cereus group |
| Clostridium tetani | CLOTE | Clostridia/Clostridiales/Clostridiaceae/Clostridium |
| Enterococcus faecalis | ENTFA | Bacilli/Lactobacillales/Enterococcaceae/Enterococcus |
| Finegoldia magna | FINM2 | Clostridia/Clostridiales/Peptoniphilaceae/Finegoldia |
| Lactobacillus delbrueckii | LACDA | Bacilli/Lactobacillales/Lactobacillaceae/Lactobacillus |
| Listeria monocytogenes | LISMO | Bacilli/Bacillales/Listeriaceae/Listeria |
| Staphylococcus epidermidis | STAES | Bacilli\Bacillales/Staphylococcaceae/Staphylococcus |
| Streptococcus pyogenes | STRP1 | Bacilli/Lactobacillales/Streptococcaceae/Streptococcus |
Скобочная формула дерева:
((((BACAN, LISMO), STAES), (LACDA, (ENTFA, STRP1))), (CLOTE, FINM2));
С помощью этой записи в программе MEGA было построено изображение этого дерева, которое можно увидеть на рисунке 1.
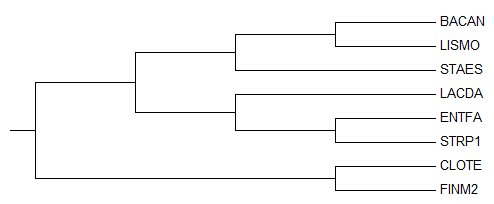 Рис.1. Изображение филогенетического дерева, полученного с помощью программы MEGA.
Рис.1. Изображение филогенетического дерева, полученного с помощью программы MEGA.
Это дерево имеет следующие нетривиальные ветви:
{BACAN, LISMO, STAES, CLOTE, FINM2} против {LACDA, ENTFA, STRP1} - Lactobacillales против всех
{BACAN, LISMO, STAES} против {LACDA, ENTFA, STRP1, CLOTE, FINM2} - Bacillales против всех
{BACAN, LISMO, STAES, LACDA, ENTFA, STRP1} против {CLOTE, FINM2} - Bacilli против Clostridia
{BACAN, LISMO} против {STAES, LACDA, ENTIFA, STRP1, CLOTE, FINM2}
{ENTIFA, STRP1} против {BACAN, LISMO, STAES, LACDA, CLOTE, FINM2}
Для дальнейшей реконструкции филогенетического древа было выбрано семейство белков - шаперонинов, с мнемоникой HSLO. Для построения дерева был использован метод "Neighbor Joining Using % Identity", которое на выходе даёт неукоренённое древо. Укоренение по методу укоренения в среднюю точку. Результат можно увидеть на рисунке 2. Сравнивая с "эталонным" деревом можно сказать, что дерево было укоренено правильно.
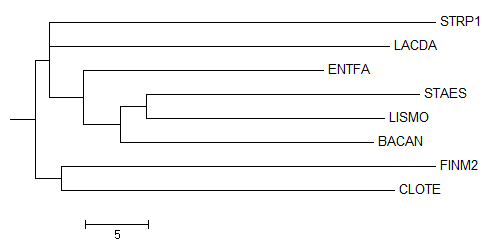 Рис.2. Изображение филогенетического дерева, укоренённое в среднюю точку.
Рис.2. Изображение филогенетического дерева, укоренённое в среднюю точку.
Это укоренение разделяет ветви {BACAN, LISMO, STAES, LACDA, ENTFA, STRP1} и {CLOTE, FINM2}, однако, как видно из рисунка 2, сам метод построения дерева не смог "разобраться" с филогенией STRP1, LACDA, ENTFA и ветви {STAES, LISMO, BACAN}