Структура нуклеиновых кислот
Для начала с помощью программы fiber пакета 3DNA были построены модели A- , B- и Z- формы ДНК, представленные соответственно на рисунках 1, 2 и 3. (ссылка на скачивание крипта для получения изображения A-формы ДНК и ссылка на скачивание скрипта для получения изображения B- и Z-формы.)
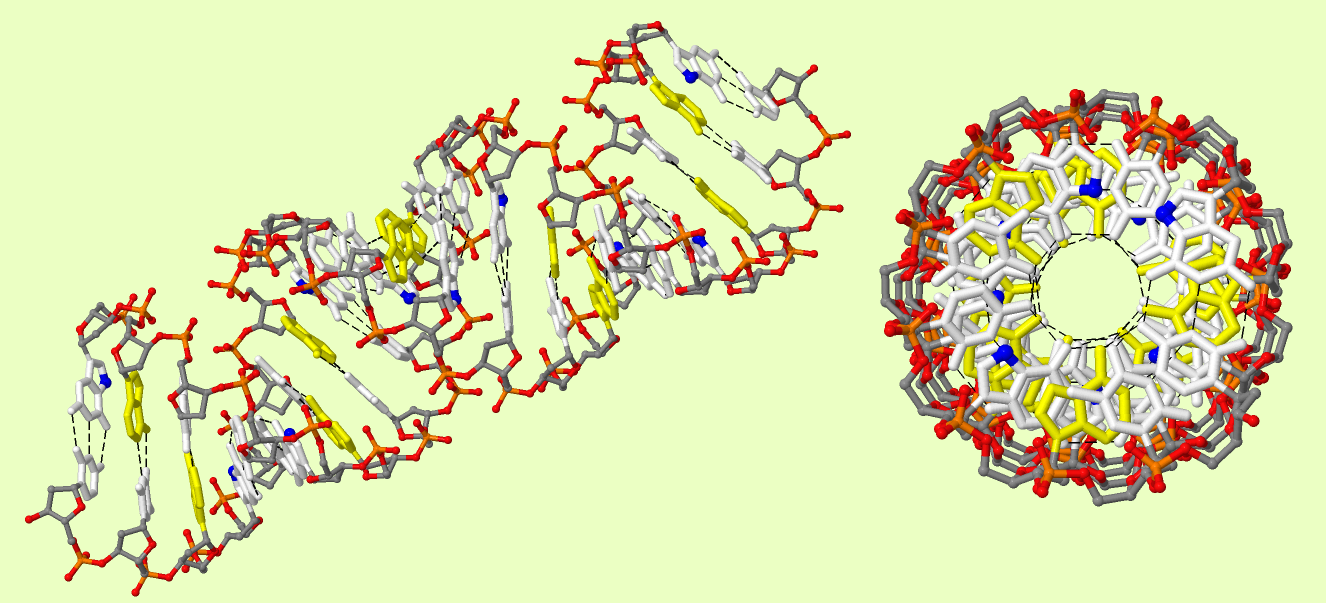 Рис.1. Трёхмерная модель А-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Отедльно жёлтым цветом воделены аденины. Синим цветом показан N7-атом в гуанинах, согласно заданию. Водородные связи показаны чёрными пунктирными линиями.
Рис.1. Трёхмерная модель А-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Отедльно жёлтым цветом воделены аденины. Синим цветом показан N7-атом в гуанинах, согласно заданию. Водородные связи показаны чёрными пунктирными линиями.
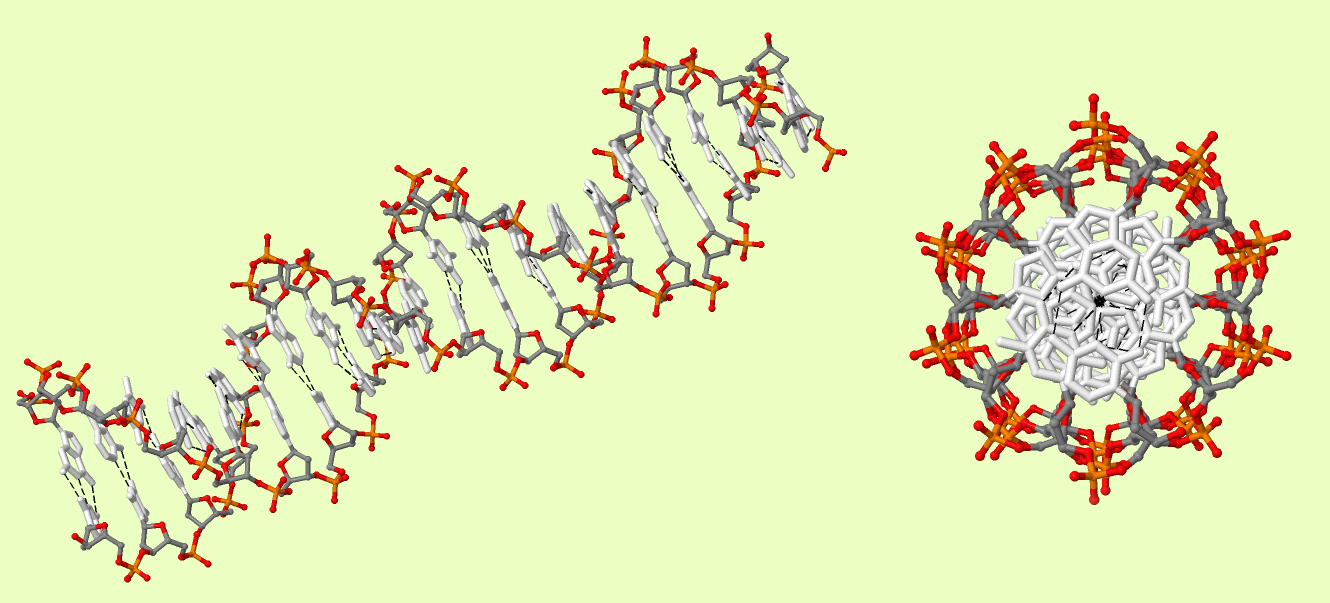 Рис.2 Трёхмерная модель B-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Водородные связи показаны чёрными пунктирными линиями.
Рис.2 Трёхмерная модель B-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Водородные связи показаны чёрными пунктирными линиями.
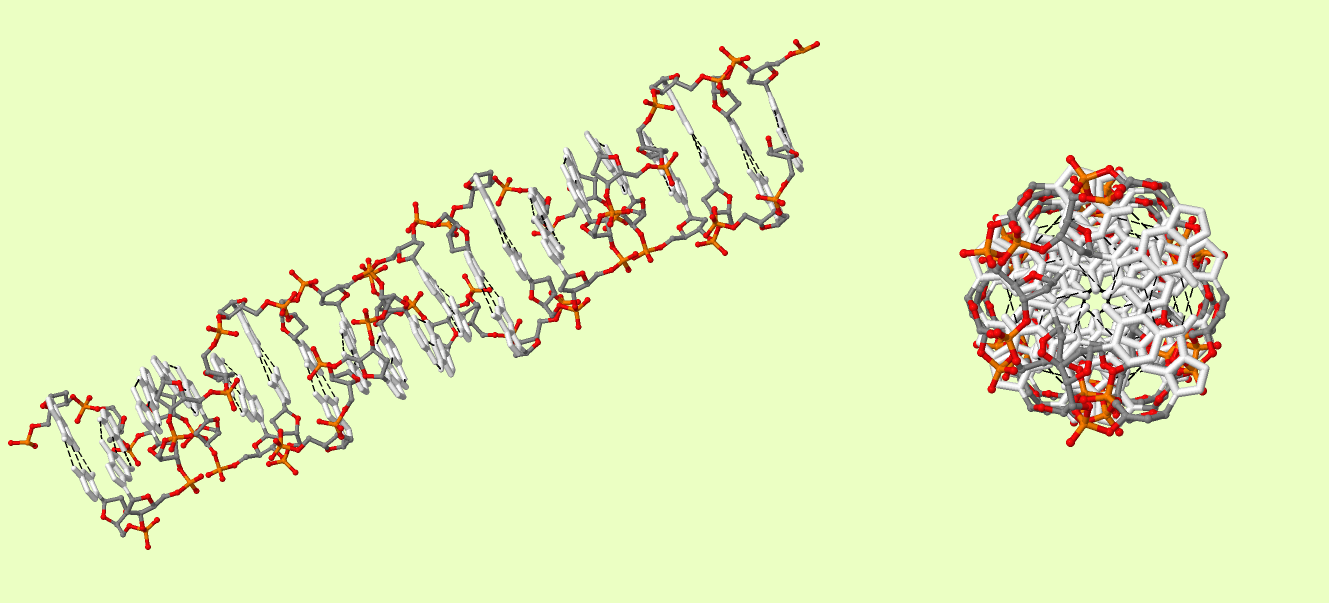 Рис.3. Трёхмерная модель Z-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Водородные связи показаны чёрными пунктирными линиями.
Рис.3. Трёхмерная модель Z-формы ДНК. Сахарофосфатный остов покрашен в соответствии с химической природой атомов (cpk), азотистые основания - в белый цвет. Водородные связи показаны чёрными пунктирными линиями.
Дальнейшая работа была проделана с pdb-файлами, содержащими комплекс ДНК-белок и тРНК. Для выполнения задания нужно было получить файлы, содержащие только цепи нуклеиновых кислот. Эти файлы можно скачать по следующим ссылкам: ДНК и тРНК. Полученные трёхмерные структуры можно увидеть на рисунке 4 и рисунке 5. Как из них видно, ни ДНК, ни РНК не имеет разрывов.
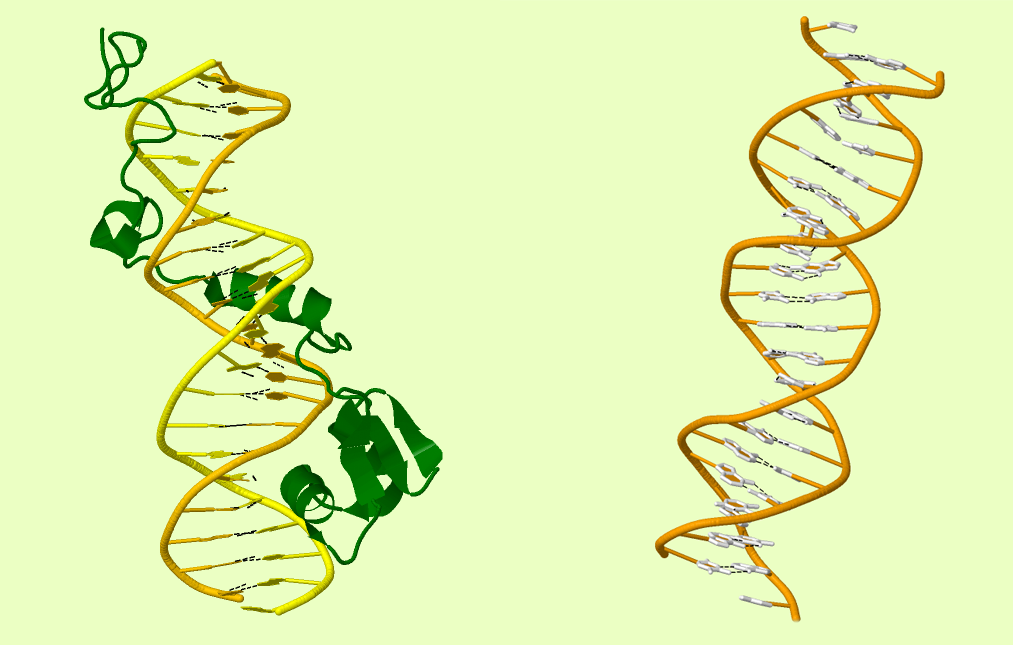 Рис.4. Трёхмерная модель комплекса ДНК с белком. Зелёным цветом выделен белок, жёлтым и оранжевым - цепи ДНК. Чёрными пунктирными линиями выделены водородные связия. Белыми проволочными моделями также выделены азотистые основания.
Рис.4. Трёхмерная модель комплекса ДНК с белком. Зелёным цветом выделен белок, жёлтым и оранжевым - цепи ДНК. Чёрными пунктирными линиями выделены водородные связия. Белыми проволочными моделями также выделены азотистые основания.
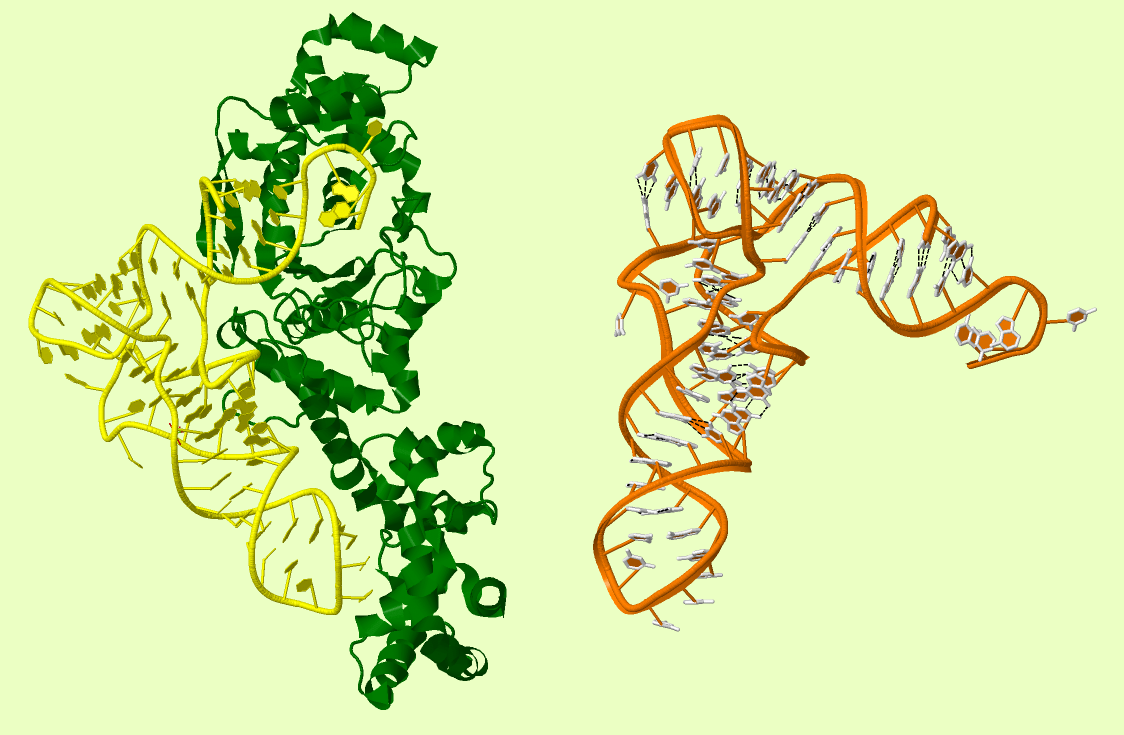 Рис.5. Трёхмерная модель транспортной РНК. Зелёным цветом выделен белок, жёлтым (оранжевым, где только РНК) цепь РНК. Чёрными пунктирными линиями выделены водородные связия. Белыми проволочными моделями также выделены азотистые основания.
Рис.5. Трёхмерная модель транспортной РНК. Зелёным цветом выделен белок, жёлтым (оранжевым, где только РНК) цепь РНК. Чёрными пунктирными линиями выделены водородные связия. Белыми проволочными моделями также выделены азотистые основания.
Далее нужно было подробнее изучить уже полученную ранее B-форму ДНК. Как видно на рисунке 6, ширина малой бороздки составляет 11.69 Å, а большой - 17.21 Å.
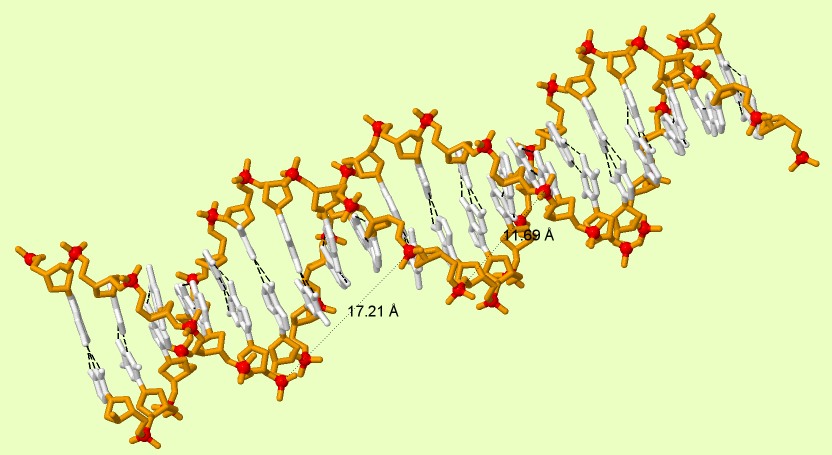 Рис.6. Большая и малая бороздка в B-форме ДНК. Белым цветом показаны азотистые основания, оранжевым - саарофосфатный остов, красным выделены атомы фосфора. Чёрными пунктирными линиями показаны водородные связи.
Рис.6. Большая и малая бороздка в B-форме ДНК. Белым цветом показаны азотистые основания, оранжевым - саарофосфатный остов, красным выделены атомы фосфора. Чёрными пунктирными линиями показаны водородные связи.
Мне нужно было рассмотреть, как расположены атомы гуанина относительно большой и малой бороздок, у меня получились следующие результаты, представленые в таблице 1. Для B-формы также построено изображение гуанина в ChemScetch с учётом напрвленности атомов, что представлено на рисунке 7.
| Атомы | Направление | Цвет |
| N1, C5, C6, N7 | Большая бороздка | красный |
| C2, N3, C4, N9 | Малая бороздка | синий |
| C8 | Промежуточное положение | чёрный |
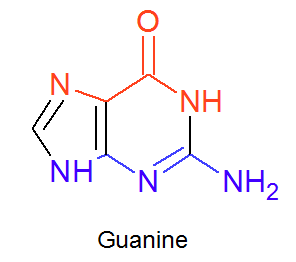 Рис. 7. Химическая структура Гуанина
Рис. 7. Химическая структура Гуанина
В Таблице 2 представлены характеристики A- , B- и Z-форм ДНК, полученные в хое работы в программе jmol.
Таблица 2. Характеристики различных форм ДНК.| A-форма | B-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 11 |
| Ширина большой бороздки | 16.97 ([C]28:B - [C]16:A) | 17.21 ([C]12:A - [A]26:B) | 18.3 ([C]8:A - [C]30:B) |
| Ширина малой бороздки | 7.98 ([G]29:B - [A]6:A) | 11.69 ([A]34:B - [T]11:A) | 8.68 ([G]19:A - [G]27:B) |
С помощью jmol были измерены торсионные углы гуанина в A- и B-формах ДНК, эти данные представлены в Таблице 3
Таблица 3. Характеристики торсионных углов гуанина в А и В формах ДНК.| угол | α | β | γ | δ | ε | ζ | χ |
| A-форма | 64,1 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| B-форма | 85,9 | 136,3 | 31,2 | 143,3 | -140,8 | -160,5 | -98,0 |
С помощью предстваленной ниже команды была получены информация о торсионных углах в A-, B- и Z-формах ДНК. Их среднее значение представлено в Таблице 4. Как видно из неё для различных нуклеотидов в A- и B- форме торсионные углы имеют примерно одно и то же значение, а торсионные угла в Z-форме для каждого нуклеотида разная. ТАк как эта Z-ДНК состояла из цитозинов и гуанинов, то в таблице представлена информация только по ним.
find_pair -t XXX.pdb stdout | analyze
| угол | α | β | γ | δ | ε | ζ | χ |
| А-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| Z-форма, цитозин | -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
| Z-форма, гуанин | 52.0 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
С помощью тех же комманд была получена таблица 5, содржащая все торсионные углы тРНК 1N78. Судя по среднему значению углов, ближе всего третичная структура этой тРНК к Ф-форме. Наиболее сильно отличающийся от других по торсионным углам нуклеотид - это 10 нуклеотид в первой цепи (Гуанин)
Таблица 5. Значение торсионных углов в структуре тРНК 1N78| Strand II | ||||||||
| № | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 | G | --- | 170.0 | 50.9 | 86.6 | -146.4 | -80.0 | -172.7 |
| 2 | G | -79.4 | -175.8 | 54.9 | 84.0 | -147.5 | -70.4 | -161.8 |
| 3 | C | -66.9 | 169.8 | 54.4 | 82.7 | -150.0 | -72.7 | -164.3 |
| 4 | C | -67.9 | 173.7 | 53.9 | 81.6 | -152.4 | -79.4 | -161.7 |
| 5 | C | -61.6 | 169.7 | 54.8 | 81.2 | -151.3 | -72.3 | -166.5 |
| 6 | C | -61.7 | 171.4 | 58.2 | 87.3 | -156.2 | -79.4 | -164.9 |
| 7 | A | -51.5 | 174.8 | 59.9 | 137.6 | --- | --- | -122.1 |
| 8 | G | --- | 142.7 | 40.0 | 81.9 | -148.5 | -73.1 | -173.8 |
| 9 | G | -61.7 | -179.6 | 48.0 | 81.3 | -153.7 | -69.3 | -170.1 |
| 10 | G | -59.4 | -178.8 | 44.5 | 81.1 | -171.6 | -78.4 | -159.2 |
| 11 | G | 160.5 | -157.2 | 168.6 | 85.9 | -107.1 | -47.3 | 176.5 |
| 12 | G | -108.7 | 90.8 | 151.8 | 88.8 | -117.5 | -68.2 | 163.3 |
| 13 | U | -68.6 | -180.0 | 45.5 | 82.5 | -125.2 | -77.6 | -162.3 |
| 14 | U | -52.7 | 155.6 | 50.9 | 81.0 | --- | --- | -153.5 |
| 15 | A | --- | 167.5 | 48.8 | 85.3 | -141.5 | -63.3 | -167.8 |
| 16 | G | -66.0 | 173.7 | 52.0 | 81.7 | -152.2 | -77.5 | -164.9 |
| 17 | G | -59.7 | 174.8 | 48.9 | 82.0 | -149.3 | -71.5 | -166.0 |
| 18 | C | -65.0 | 174.3 | 53.2 | 82.6 | -155.0 | -68.8 | -159.4 |
| 19 | C | -70.3 | 174.5 | 57.1 | 78.3 | -153.2 | -69.3 | -166.3 |
| 20 | G | -57.1 | 174.6 | 54.7 | 81.1 | -155.7 | -81.6 | -164.7 |
| 21 | A | -63.2 | 173.9 | 50.5 | 82.4 | --- | --- | -154.3 |
| 22 | G | --- | 178.1 | 52.4 | 88.3 | -148.4 | -67.7 | -178.6 |
| 23 | U | -70.4 | -175.8 | 48.0 | 81.6 | -154.2 | -71.0 | -164.7 |
| 24 | C | -65.9 | -174.2 | 48.5 | 81.5 | -152.9 | -66.4 | -159.7 |
| 25 | U | 140.4 | -155.7 | -167.0 | 83.4 | -153.3 | -77.5 | -171.5 |
| 26 | A | -57.7 | -174.8 | 45.3 | 79.8 | -139.4 | -66.7 | -170.7 |
| 27 | G | -43.4 | 161.1 | 58.8 | 129.6 | --- | --- | -119.6 |
| 28 | G | --- | -131.3 | 48.7 | 114.2 | --- | --- | -64.1 |
| Strand II | ||||||||
| № | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 | C | 157.9 | -143.8 | 161.4 | 87.5 | --- | --- | -168.9 |
| 2 | U | -70.0 | 176.4 | 52.4 | 83.8 | -168.0 | -94.2 | -160.5 |
| 3 | G | -47.2 | 160.9 | 54.1 | 82.8 | -149.9 | -65.8 | -174.1 |
| 4 | G | -64.4 | -178.5 | 51.0 | 84.1 | -153.2 | -79.1 | -169.2 |
| 5 | G | -67.4 | 178.8 | 47.4 | 79.5 | -163.2 | -65.5 | -166.9 |
| 6 | G | -64.8 | 178.2 | 47.2 | 83.1 | -160.2 | -67.8 | -159.0 |
| 7 | U | -73.6 | 179.7 | 54.5 | 83.4 | -157.5 | -67.0 | -158.2 |
| 8 | C | -65.0 | 177.1 | 55.6 | 84.4 | -156.9 | -60.6 | -163.4 |
| 9 | C | -63.4 | 168.9 | 55.7 | 82.2 | -154.5 | -65.1 | -162.6 |
| 10 | C | -73.5 | -169.5 | 49.6 | 84.4 | -160.3 | -76.4 | -155.6 |
| 11 | C | -70.7 | -175.7 | 51.4 | 83.8 | -159.3 | -67.7 | -163.7 |
| 12 | C | --- | -150.3 | 47.5 | 82.2 | -158.3 | -74.2 | -165.5 |
| 13 | A | --- | -96.6 | 179.1 | 81.6 | --- | --- | -149.1 |
| 14 | G | --- | -149.9 | -64.9 | 108.9 | --- | --- | -115.4 |
| 15 | C | -61.1 | 167.7 | 51.8 | 85.0 | --- | --- | -162.5 |
| 16 | C | -63.2 | 173.9 | 53.7 | 82.8 | -141.4 | -70.0 | -159.9 |
| 17 | C | -66.3 | 172.8 | 55.1 | 83.9 | -154.5 | -70.5 | -164.9 |
| 18 | G | -64.4 | 178.8 | 53.5 | 83.2 | -158.3 | -72.1 | -165.6 |
| 19 | G | -72.3 | -179.5 | 52.5 | 79.0 | -153.2 | -71.9 | -169.1 |
| 20 | C | -64.4 | 178.2 | 50.5 | 78.2 | -146.9 | -73.6 | -159.7 |
| 21 | G | -67.1 | 169.9 | 50.2 | 81.8 | -148.3 | -65.8 | -164.9 |
| 22 | C | -60.0 | 176.0 | 52.8 | 84.1 | -144.6 | -61.6 | -163.0 |
| 23 | A | -63.5 | 176.3 | 50.3 | 81.0 | -152.1 | -76.5 | -164.6 |
| 24 | G | -54.3 | 170.7 | 53.6 | 81.0 | -153.0 | -69.0 | -166.0 |
| 25 | G | --- | 142.1 | 45.6 | 80.2 | -147.5 | -81.7 | -172.6 |
| 26 | U | --- | -156.2 | 59.0 | 85.8 | --- | --- | -152.1 |
| 27 | C | --- | 108.0 | 172.3 | 133.5 | --- | --- | -123.6 |
| 28 | C | --- | 162.2 | 52.6 | 83.3 | --- | --- | -160.1 |
| Среднее знаечние | 68,75116279 | 166,2211538 | 63,11346154 | 86,11923077 | 150,6325581 | 71,29069767 | 160,7673077 | |
| Strand I | ||||||||
| № | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 | T | --- | 166.7 | 60.3 | 146.6 | -171.5 | -102.4 | -105.7 |
| 2 | C | -50.8 | -174.0 | 31.0 | 150.4 | 175.8 | -114.2 | -85.3 |
| 3 | T | -16.9 | -174.6 | 13.5 | 150.0 | -161.2 | -114.5 | -99.4 |
| 4 | T | 18.2 | -162.5 | -58.8 | 151.2 | -159.9 | -95.2 | -95.8 |
| 5 | G | -60.2 | 175.4 | 34.8 | 153.6 | -122.6 | -179.0 | -65.5 |
| 6 | G | -67.3 | 137.9 | 49.4 | 147.5 | -149.2 | -150.8 | -89.9 |
| 7 | G | -71.5 | 157.8 | 56.9 | 147.8 | -174.3 | -92.2 | -122.1 |
| 8 | T | -55.9 | -159.4 | 25.3 | 144.7 | 161.1 | -82.0 | -95.7 |
| 9 | C | 151.3 | 150.4 | -171.3 | 135.6 | -154.7 | -113.1 | -129.7 |
| 10 | T | -38.9 | -174.8 | 29.9 | 144.0 | -170.3 | -109.5 | -105.1 |
| 11 | A | -48.8 | -178.3 | 40.5 | 147.1 | -166.1 | -113.6 | -93.0 |
| 12 | C | 22.8 | -169.9 | -55.1 | 153.1 | -178.1 | -115.7 | -91.8 |
| 13 | C | 27.5 | -161.8 | -61.8 | 149.8 | -159.9 | -93.1 | -124.5 |
| 14 | G | -55.8 | -169.5 | 37.6 | 148.3 | 170.5 | -108.5 | -99.3 |
| 15 | T | -43.8 | -171.3 | 39.0 | 147.8 | -168.4 | -120.7 | -103.4 |
| 16 | T | 26.1 | -168.3 | -53.3 | 154.4 | 179.4 | -89.1 | -107.1 |
| 17 | T | -50.1 | -171.0 | 29.7 | 156.8 | -166.8 | -154.0 | -84.6 |
| 18 | A | -44.9 | 159.2 | 47.7 | 146.3 | 177.3 | -90.7 | -92.8 |
| 19 | A | -37.0 | -155.4 | 25.0 | 151.9 | -162.2 | -112.0 | -86.8 |
| 20 | T | 24.7 | -126.9 | -75.8 | 161.3 | --- | --- | -119.5 |
| Strand II | ||||||||
| № | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 | A | 16.3 | -176.4 | -51.6 | 159.8 | --- | --- | -94.6 |
| 2 | G | -52.4 | 164.8 | 49.0 | 151.0 | -161.1 | -112.2 | -107.6 |
| 3 | A | -38.5 | 160.2 | 37.6 | 152.6 | -163.9 | -149.8 | -85.8 |
| 4 | A | -48.7 | 157.8 | 48.3 | 145.1 | -168.7 | -123.3 | -83.3 |
| 5 | C | -41.4 | 175.4 | 31.3 | 144.7 | -156.6 | -144.8 | -86.6 |
| 6 | C | -27.9 | -172.7 | 17.8 | 142.9 | -164.0 | -105.2 | -105.6 |
| 7 | C | 41.2 | 174.7 | -57.0 | 151.5 | -176.1 | -98.0 | -112.2 |
| 8 | A | -35.2 | 167.6 | 23.9 | 147.1 | -172.2 | -120.7 | -111.5 |
| 9 | G | -49.4 | -177.4 | 29.3 | 152.5 | -166.5 | -150.0 | -85.4 |
| 10 | A | -33.9 | 175.0 | 46.1 | 149.0 | -175.2 | -99.2 | -96.8 |
| 11 | T | -48.9 | -167.7 | 35.9 | 146.1 | -171.5 | -123.0 | -99.1 |
| 12 | G | 32.9 | -177.8 | -52.9 | 151.4 | 165.5 | -100.4 | -125.1 |
| 13 | G | -42.4 | -161.1 | 33.1 | 154.1 | 177.5 | -130.9 | -94.6 |
| 14 | C | 32.0 | -161.0 | -57.9 | 150.1 | -178.5 | -93.9 | -100.8 |
| 15 | A | -21.7 | 162.3 | 31.9 | 147.7 | -170.7 | -111.2 | -96.5 |
| 16 | A | -35.0 | -173.9 | 24.6 | 151.0 | 170.0 | -131.3 | -84.8 |
| 17 | A | -36.7 | 174.5 | 42.1 | 147.1 | 175.8 | -101.8 | -85.5 |
| 18 | T | -31.1 | 178.3 | 26.2 | 144.4 | -176.8 | -119.8 | -93.6 |
| 19 | T | 6.0 | -151.1 | -80.2 | 143.3 | -167.5 | -112.0 | -108.4 |
| 20 | A | --- | 177.9 | 47.4 | 155.3 | -132.9 | -137.0 | -85.2 |
| Среднее значение | 42,86388889 | 166,8 | 44,04722222 | 148,6638889 | 167,1083333 | 115,9833333 | 98,19444444 | |
С помощью find_pair также можно узнать о структуре водородных связей в молекуле нуклеиновой кислоты. В таблице 7 показаны стебли в третичной структуре тРНК, в таблице 8 - неканонические пары оснований, а в таблице 9 - информация о дополнительных водородных связях в той же молекуле тРНК.
Таблица 7. Стебли в третичной структуре тРНК| № | Нуклеотиды первой цепи | Нуклеотиды второй цепи | Длина стебля (пар нуклеотидов) |
| 1 | C:.501 - C:.507 | 572_:C - 566_:C | 7 |
| 2 | C:.549 - C:.553 | 565_:C - 561_:C | 5 |
| 3 | C:.538 - C:.543 | 532_:C - 527_:C | 6 |
| 4 | C:.510 - C:.513 | 525_:C - 522_:C | 4 |
| Нуклеотид первой цепи | Нуклеотид второй цепи | Тип пары |
| C:.502 | 571_:C | G - U |
| C:.555 | 518_:C | U - G |
| C:.538 | 532_:C | A - C |
| C:.544 | 526_:C | A - G |
| C:.513 | 522_:C | U - G |
| Нуклеотид первой цепи | Нуклеотид второй цепи |
| C:.554 | 558_:C |
| C:.555 | 518_:C |
| C:.544 | 526_:C |
| C:.514 | 508_:C |
| C:.515 | 548_:C |
Также помощью пакета 3DNA можно исследовать стекинг-взаимодействия. На рисунке 8 изображена вторая пара нуклеотидов, которая имеет самый большой процент пересрывания азотистых оснований. Избражение сгенерировано с помощью программы stack2img.
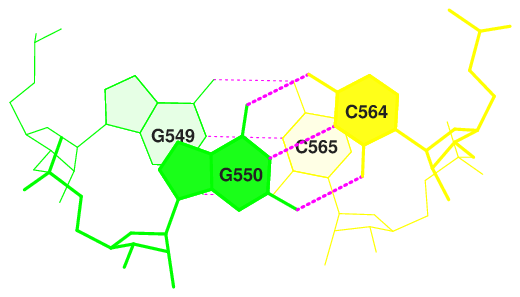 Рис. 8. Схема стекинг-взаимодействия второй пары нуклеотидов
Рис. 8. Схема стекинг-взаимодействия второй пары нуклеотидов