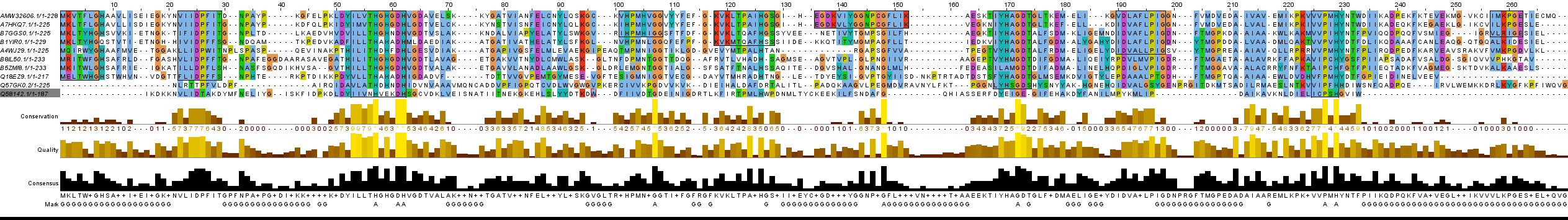
| AC | RecName | Coverage, % | Identity, % | E-value | Homology |
| AMW32606 | metal-dependent hydrolase [Fervidobacterium islandicum] | — | — | — | — |
| A7HKQ7 | UPF0173 metal-dependent hydrolase Fnod_0635 | 98 | 81 | 1e-139 | ✔ |
| B7GGS0 | UPF0173 metal-dependent hydrolase Aflv_0488 | 99 | 50 | 6e-71 | ✔ |
| B1YIR0 | UPF0173 metal-dependent hydrolase Exig_1934 | 99 | 45 | 1e-60 | ✔ |
| A4WJ29 | UPF0173 metal-dependent hydrolase Pars_0810 | 99 | 38 | 9e-50 | ✔ |
| B8IL50 | UPF0173 metal-dependent hydrolase Mnod_3315 | 98 | 34 | 8у-41 | ✔ |
| B5ZMI8 | UPF0173 metal-dependent hydrolase Rleg2_1519 | 98 | 34 | 4e-30 | ✔ |
| Q18EZ9 | UPF0173 metal-dependent hydrolase HQ_3368A | 91 | 34 | 5e-23 | ✔ |
| Q57GK0 | Probable L-ascorbate-6-phosphate lactonase UlaG | 88 | 22 | 0,007 | ✘ |
| Q58142 | Uncharacterized protein MJ0732 | 78 | 23 | 5,0 | ✘ |