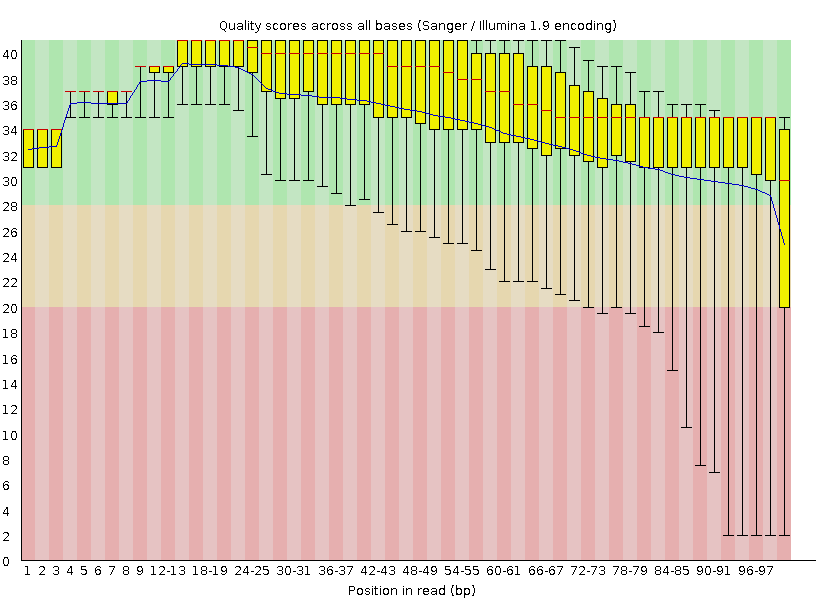
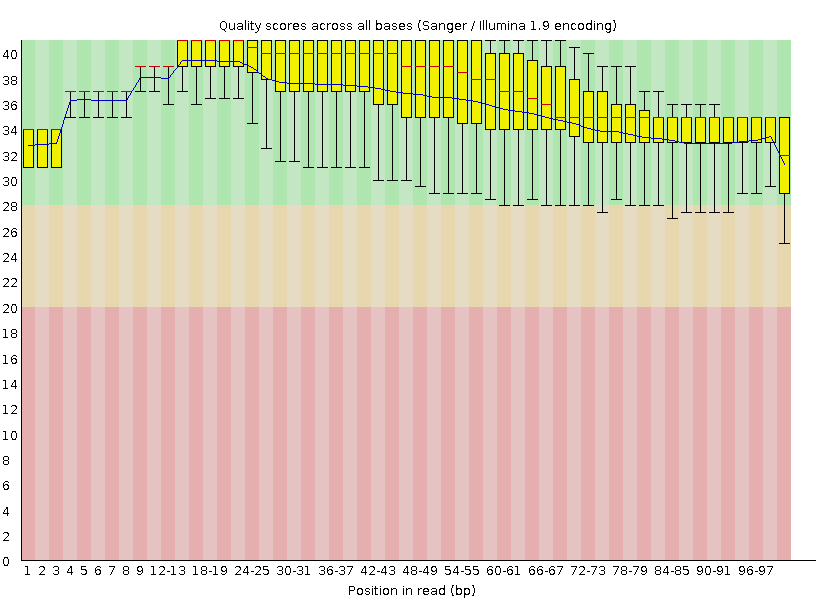
| качество прочтений до очистки | качество прочтений после очистки |
 |  |
| количество чтений до очистки | количество чтений после очистки |
| 4661 | 4472 |
4472 reads; of these:
4472 (100.00%) were unpaired; of these:
17 (0.38%) aligned 0 times
4455 (99.62%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.62% overall alignment rate
| хромосома | тип полиморфизма | координата | в референсе | в ридах | качество покрытия | покрытие |
| chr20 | 33903158 | замена | G | A | 3.01394 | 1 |
| chr20 | 33913681 | замена | A | C | 6.20226 | 1 |
| chr20 | 33918910 | замена | C | T | 73.0074 | 15 |
| Координата | SNP | Quality и DP | refgene | dbsnp | 1000 genomes | Gwas | Clinvar |
| 34025983 | замена A на G | 176.016 | upstream hom | rs143383 | 0.3718 | - | остеоартрит |
| 33913681 | замена A на C | 6.20226 | intronic hom | rs6060372 | 0.326 | - | - |
| 48522330 | замена G на A | 196.009 | exonic het | rs495337 | 0.3976 | псориаз | - |