Задание 1. Поиск гена δ-субъединицы АТФ-синтазы
Организм: Немецкая овчарка (Canis lupus familiaris). Сборка: RefSeq GCF_011100685.1 (UU_Cfam_GSD_1.0).
Цель — найти в геноме Canis lupus familiaris ген, кодирующий δ-субъединицу АТФ-синтазы. В результате была найдена запись с идентификатором XP_038423949.1. Аминокислотная последовательность сохранена в файле
- Идентификатор нуклеотидной записи, к которой относится ген: NC_049241.1
- ДНК-последовательность, непосредственно содержащая кодирующую белок часть гена δ-субъединицы АТФ-синтазы в пределах CDS (без вырезания экзонов) находится в файле

На рисунке показан участок хромосомы 20, содержащий ген ATP5F1D, а также его ближайшие соседние гены.
Идентификатор белка: XP_038423949.1
Идентификатор нуклеотидной записи: NC_049241.1
Координаты участка, загруженного через Visible Range: 58027996–58031287 (хромосома 20, сборка UU_Cfam_GSD_1.0)
Координаты самого гена: 58028376-58030908, его последовательность
FASTA-файл с кодирующей областьюЗадание 2. Поиск BLAST для ДНК-фрагмента
Выбранный таксон: Пауки (Araneae, taxid:6893).
Это таксон первичноротых, филогенетически далекий от нашего организма - собаки (вторичноротых).
blastn
blastn предназначен для выявления нуклеотидных гомологов, то есть таких генов, у которых сохранены консервативные участки ДНК. Однако при значительных эволюционных расхождений (как в нашем случае с Canis lupus familiaris и Araneae) нуклеотиды сильно дивергируют, поэтому BLASTN не находит статистически значимых совпадений. Это ожидаемо, т.к. для далеких организмов поиск гомологов эффективнее реализуется на белковом уровне - TBLASTX (см. ниже).Использованная база данных: RefSeq Genomes.
Число сборок, входящих в таксон Araneae: 4
Параметры поиска:
– длина слова (word size): 11;
– остальные параметры — по умолчанию.
Уменьшение длины слова позволяет находить более короткие совпадения.
Высвечивается одна вертикальная полоса из коротких зелёных и синих блоков — это небольшие участки низкого сходства, встречающиеся примерно в одном и том же месте последовательности.
tblastn
tblastn — инструмент для анализа белковых последовательностей, который используется для поиска сходных последовательностей в базе данных.
Использованная база данных: RefSeq Genomes.
Остальные параметры по умолчанию.
Число сборок в таксоне Araneae: 4 .
tblastn нашёл 4 значимых гомолога у пауков. Все совпадения имеют низкие e-value, что указывает на статистическую значимость.
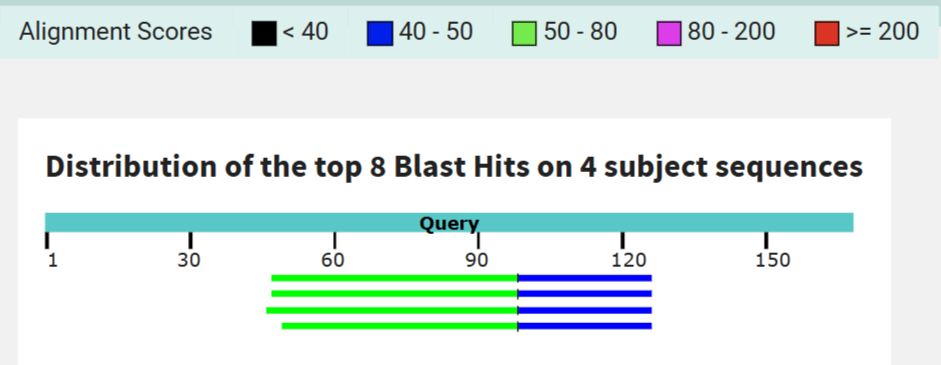
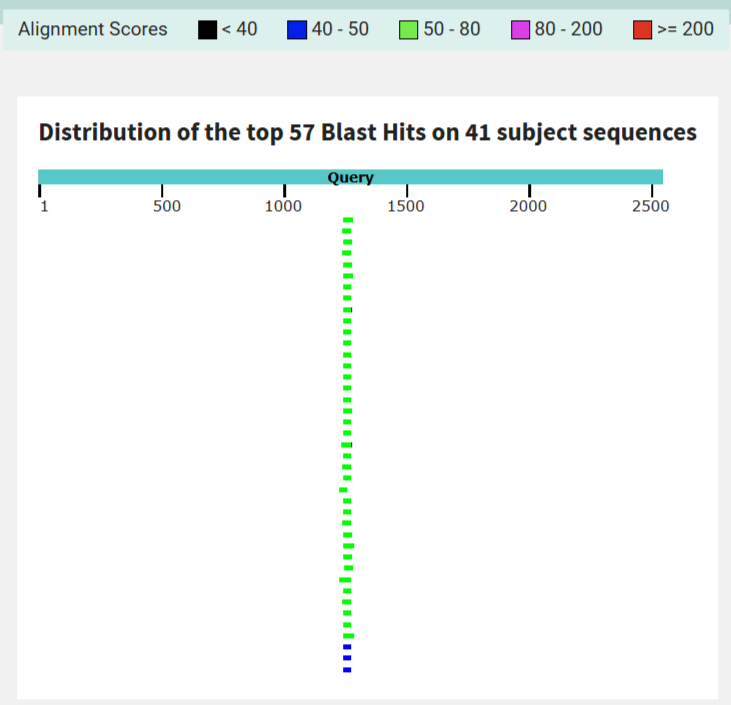
График распределения лучших попаданий tblastn показывает, что все найденные последовательности выравниваются с нашим белком примерно на половину его длины (около 46–48%). Окраска сегментов соответствует высоким alignment score, характерным для дальних, но реальных гомологий.
Задание 3. Поиск основных рибосомальных РНК по далекому гомологу
Команда подающаяся на вход для индексирования последовательности организма для последующей работы локального BLAST:
makeblastdb -in .\GCA_011100685.1_UU_Cfam_GSD_1.0_genomic.fna -dbtype nucl -out dog_db
После выполнения команды были созданы файлы dog_db.nhr, dog_db.nin, dog_db.nsq, что означает корректное формирование локальной нуклеотидной базы.
Далее была скачена последовательность рРНК Escherichia coli и проведен локальный BLAST отдельно для каждой рРНК по полученной базе данных с помощью метода blastn, потому что нам нужно искать нуклеотидные рРНК в нуклеотидном геноме, megablast не подходит т.к. он ищет очень близкие последовательности.
Поиск проводился отдельно для 16S и 23S рРНК:
Команда для поиска по 16s рРНК:
blastn -task blastn -query rrnaE.coli16S.txt -db dog_db -out dog_16S.txt -evalue 0.01 -outfmt 7
Выдача:
BLAST нашёл 15 хитов, все выравнивания имеют: 1) длина выравнивания всего 43 нт 2) идентичность ≈ 88% 3) все хиты соответствуют одному и тому же участку бактериальной 16S: 1494–1536 4) E-value ≈ 8e-05
В геноме собаки обнаружены многочисленные повторяющиеся участки, похожие на маленький консервативный фрагмент бактериальной 16S рРНК.
Команда для поиска по 23s рРНК:
blastn -task blastn -query rrnaE.coli23S.txt -db dog_db -out dog_23S.txt -evalue 0.01 -outfmt 7
Выдача:
BLAST нашёл 71 хит, среди них есть немного более длинные совпадения: 1) фрагменты 175 нт, 93 нт, 86 нт, 69 нт 2) идентичность 73–81% 3) совпадают с разными частями бактериальной 23S (участки ~200–300 и ~2400–2600 нт) 4) E-value варьируется
При выравнивании 23S рРНК Escherichia coli с геномными последовательностями собаки алгоритм blastn обнаружил 71 гомологичный фрагмент. Все найденные хиты соответствуют нескольким коротким консервативным участкам 23S рРНК E. coli, которые повторяются во всех найденных выравниваниях: 198–290; 1898–1983; 2233–2300; 2442–2613.
IMG_5149.JPEGНа рисунке 4 показан пример гомолога 16S рРНК E. coli, найденного BLAST в геноме собаки на скаффолде JAAHUQ010002733.1. Участок 16S рРНК, соответствующий позициям 1494–1536, выровнялся с областью 23220–23262 собачьего генома.
Задание 4. Карты локального сходства
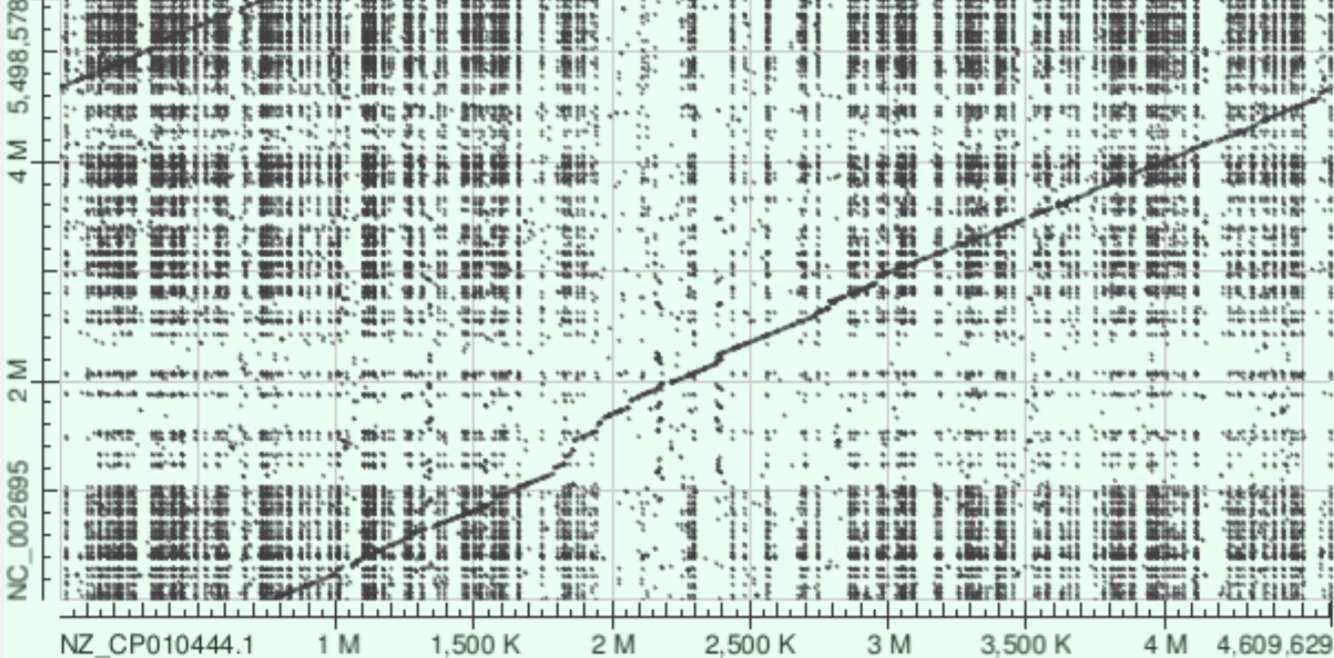
Для выполнения задания были выбраны два разных штамма кишечной палочки: Escherichia coli K-12 MG1655, Escherichia coli O157:H7 Sakai.
Результаты по megablast
Видна одна чёткая главная диагональ — это большие консервативные блоки, сохранившиеся между штаммами. В верхней части карты заметен небольшой параллельный участок диагонали. Скорее всего его нахождение связано с тем, что оба генома кольцевые, но записаны с разными точками начала.
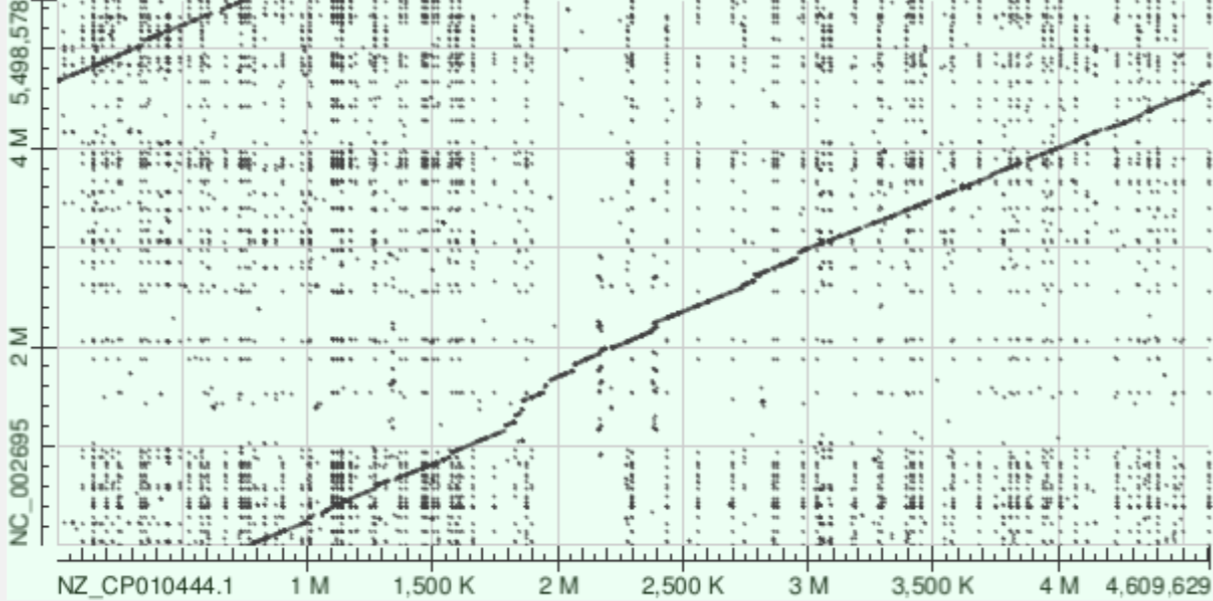
Результаты по blastn
Карта BLASTN выглядит значительно более «шумной» по сравнению с предыдущей: по всей площади графика заметно большое количество мелких локальных совпадений. Это ожидаемо, потому что blastn находит гораздо больше коротких, менее консервативных участков гомологии. На главной диагонали по-прежнему хорошо виден основной сигнал сходства между двумя штаммами E. coli. Множество близлежащих точек соответствуют небольшим повторам, фаговым вставкам, которые встречаются в разных местах обоих геномов.