Построение парных выравниваний. Поиск по сходству
Задания выполнялись для белка N-acetylmannosamine-6-phosphate 2-epimerase, Clostridium perfringens (AC:WP_003452659.1)
Выборка гомологов
Я зарегистрировалась на сайте NCBI, вошла в личный кабинет. Затем я запустила программу Protein BLAST,
предназначенную для поиска участков сходства в последовательностях белков. Был выбран алгоритм
blastp (protein-protein BLAST), база данных - Reference proteins (refseq_protein).
Сначала я использовала параметры по умолчанию (100 последовательностей на выходе и без таксономического
ограничения), но оказалось, что в базе данных слишком много гомологов данного белка. При выдаче 100
у самой худшей находки E-value составил 2e-92, при выдаче 1000 - 5e-70, что свидетельствовало о том,
что необходимо запросить больше последовательностей. Также я ограничила поиск таксоном Bacteria.
Ссылка на итоговые параметры поиска
Итоговое число находок в рамках таксона Bacteria - 4143 (включая исходную последовательность).
Среди них достаточно много находок,
гомологичных исследуемогу белку по
всей длине (query cover > 80%) - 3607.
Используя условный критерий гомологичности (E-value<0.001 и Query cover не менее 70%), можно заключить,
что в базе данных среди таксона Bacteria найдено 3633 гомоглогов целой исходной последовательности.
Применительно к данному случаю такой критерий я считаю разумным. Последовательности, не удовлетворяющие
этому критерию, не показались мне гомологичными.
В таблице представлено сравнение по ключевым параметрам нескольких находок: лучшей - 1, худшей - 5 и из
середины списка - 2, 3 и 4.
| Организм | Длина выравнивания | Bit Score | E-value | Идентичные остатки,% | Сходные остатки, % | Выравнивание | |
|---|---|---|---|---|---|---|---|
| 1 | Clostridium perfringens | 221 | 449 bits(1155) | 3e-160 | 99% | 100% | Ссылка |
| 2 | Peptoclostridium difficile | 216 | 301 bits(772) | 7e-102 | 69% | 80% | Ссылка |
| 3 | Cyanothece sp. PCC 7822 | 208 | 182 bits(461) | 1e-54 | 46% | 63% | Ссылка |
| 4 | Klebsiella pneumoniae | 216 | 147 bits(370) | 7e-41 | 39% | 62% | Ссылка |
| 5 | Nocardiopsis dassonvillei | 45 | 36.6 bits(83) | 9.9 | 44% | 60% | Ссылка |
Сохраненная выборка 29 последовательностей в формате fasta
Множественное выравнивание последовательностей из полученной выборки
С помощью Jalview и Muscle я построила множественное выравнивание полученной выборки. Вертикальные
блоки, удовлетворяющие введенному ранее техническому определению, отмечены символом B. Блоки, не
являющиеся вертикальными, но объединяющие значительное количество последовательностей, выделены в группы
и обозначены символом H. Видно, что таких блоков достаточно много, что свидетельствует о гомологичности
рассматриваемых последовательностей.
На C-концах многих последовательностей присутствовали длинные
невыровненные участки, различные у разных находок, что не свидетельствует об отсутствии гомологии
между последовательностями в целом. Эти участки были удалены. На N - концах также присутствовали подобные
участки, но значительно короче, что может говорить о важности консервативности N-конца для функционирования
данного белка.

Ссылка на JalView проект, множественное выравнивание
Глобальное и локальное парные выравнивания
Для выполнения этого задания необходимо было воспользоваться программами Needle и Water через сервер
Kodomo с помощью PuTTy. В качестве входных данных они требуют две последовательности в fasta-формате,
величины штрафов за первый гэп (по умолчанию 10.0) и за продление гэпа (по умолчанию 0.5), а также имя выходного файла.
Needle строит глобальные выравнивания двух последовательностей (выравнивания полных
последовательностей белков относительно друг друга, имеющее по возможности максимальный счет).
Water строит локальные выравнивания (выравнивания неполных последовательностей, имеющие максимальный счет).
По умолчанию needle выдает файлы формата .needle, а water - файлы формата .water,
содержащие выравнивание и аннотацию.
Особенность файлов данного вида в том, что в них указывается не только прямое совпадение, но и сходство
аминокислот (проставляются соответственно одна или две точки).
Чтобы получить выходной файл в fasta-формате, необходимо в качестве опции запуска указать -aformat3.fasta.
Использованные последовательности - WP_003452659.1 (мой белок) и WP_052908071.1.
Глобальное парное выравнивание, полученное с помощью Needle

Глобальное парное выравнивание, полученное из множественного

Локальное парное выравнивание, полученное с помощью Water

Локальное парное выравнивание, полученное с помощью BLAST
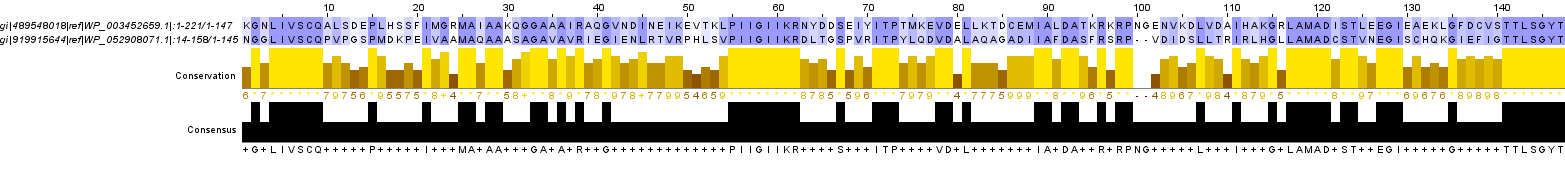
4 выравнивания в одном окне

Ссылка на JalView проект, 1 окно с 4 группами
Выравнивание выравниваний
Четыре полученных выравнивания: глобальное (выданное needle), глобальное (полученное из множественного),
локальное (выданное water) и локальное (выданное BLAST) были помещены в одно окно JalView и
объединены в 4 соответствующие группы. Затем было произведено выравнивание четырех выравниваний таким образом,
чтобы одинаковые колонки из разных выравниваний оказались друг под другом.
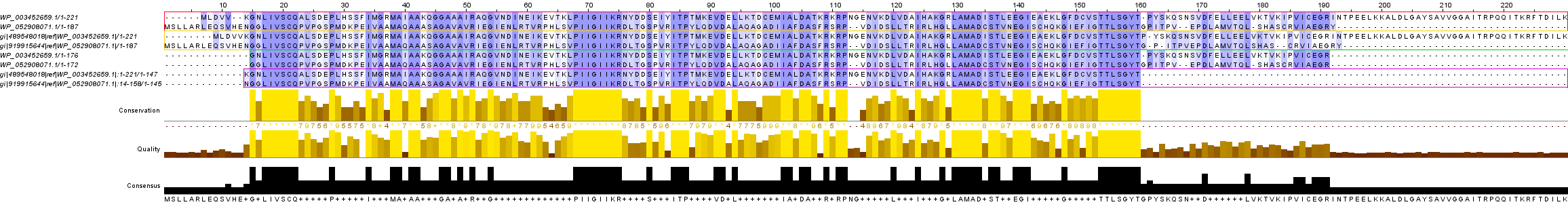
Ссылка на JalView проект с выравниванием выравниваний
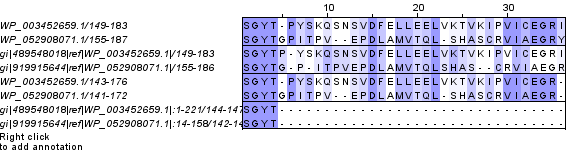
Было замечено, что встречаются участки, на которых выравнивания различаются. Так, больше всего различий было обнаружено между глобальными выравниваниями, одно из которых было получено с помощью Needle, а другое из множественного. Выравнивания, полученные с помощью Needle и Water совпали полностью, не считая, естественно, обрезанных Water участков. Локальные выравнивания Water и Blast различаются по длине, но на всех общих участках полностью совпадают.
На рисунке проиллюстрирован участок несовпадения выравниваний.
Парные выравнивания последовательностей заведомо негомологичных белков
Я использовала последовательность своего белка и белка WP_010878069.1 (cysteine desulfurase [Archaeoglobus fulgidus]).
С помощью Needle и Water были получены глобальное и локальное выравнивания,
котрые затем были помещены в одно окно JalView и выровнены относительно друг друга.
Выравнивания, полученные разными программами, в большей степени совпадают (не считая обрезанных
water N/C-концов), однако несовпадающих колонок тоже достаточно, значительно больше, чем в
случае с гомологичными последовательностями из предыдущего задания. Это свидетельствует о
том, что полученные выравнивания неточны и найденные в каждом из выравниваний совпадющие позиции
не всегда информативны.

Ссылка на JalView проект с выравниваниями негомологичных последовательностей