Анализ множественных выравниваний
Вертикальные блоки
Вертикальный блок мы определили как участок множественного выравнивания, обладающий следующими свойствами:
- Не содержит гэпов
- Длина - не менее 4 колонок
- Первая и последняя колонки - абсолютно консервативны или абсолютно функционально консервативны
- Процент абсолютно консервативных колонок больше 35%
- Не содержит более 10 колонок, не являющихся абсолютно консервативными или абсолютно функционально консервативными, подряд
- Не может быть расширен без нарушения предыдущих свойств

Ссылка на JalView проект, задание 1
Один блок из части последовательностей
Две из пяти данных последовательностей показались мне более схожими между собой. Я объединила
их в одну группу и раскрасила так же, как выравнивание. Символом H выделен участок, на котором
в пределах группы все остатки с очень высокой вероятностью гомологичны ввиду абсолютной консервативности
колонок, а за пределами группы эта вероятность снижается, главным образом из-за наличия в
других трех последовательностях гэпа и нескольких колонок, не являющихся абсолютно функционально
консервативными.

Ссылка на JalView проект, задание 2
Число и процент консервативных позиций
Параметры были подсчитаны для вертикального блока с координатами 66-73. Всего в нем 8 колонок.
| Позиции | Число | Процент |
|---|---|---|
| Абсолютно консервативные | 3 | 37,5% |
| Абсолютно функционально консервативные | 6 | 75,0% |
Число и процент позиций с гэпами в самом длинном участке за пределами блоков
Самый длинный участок выравнивания, не входящий в состав блоков, обозначен символом X.
Общее число колонок в нем - 35. Число позиций с гэпами - 2. Процент позиций с гэпами - 5,71%.

Ссылка на JalView проект, процент гэпов
Консенсусная последовательность и LOGO одного блока
Консенсусная последовательность (consensus sequence)
— усредненная нуклеотидная или аминокислотная последовательность, которая регулярно встречается
в данном генетическом элементе. В каждом из ее положений находятся аминокислоты или нуклеотиды, чаще всего
встречающиеся в данной позиции во всех известных родственных последовательностях.
Консенсусные последовательности создаются на основе анализа массивов выровненных последовательностей и часто
принимаются за эталон в сравнительном анализе. [1]
Для получения данной последовательности для блока 66-73 я воспользовалась программой cons на
сервере http://emboss.bioinformatics.nl/. Она
принимает на вход совокупность выровненных последовательностей и на выход выдает требуемую
консенсусную последовательность в выбранном формате (Fasta, gbk и др.).
Полученный на выходе fasta-файл: consensus.fasta .
Для получения LOGO блока был использован сервис
http://weblogo.threeplusone.com/, который принимает на вход совокупность выровненных последовательностей
и автоматически выдает искомое изображение в указанном разрешении. По умолчанию аминокислотные последовательности
окрашиваются по гидрофобности. Гидрофобные аминокислоты выделяются черным, гидрофильные синим, а
нейтральные зеленым.
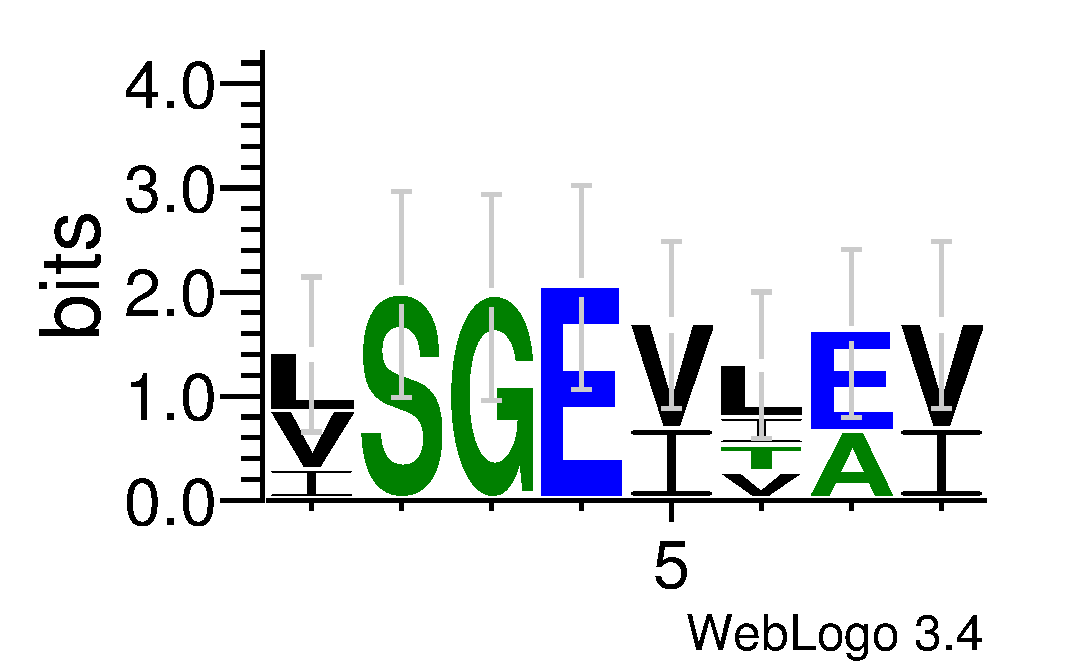
Паттерн блока
Паттерн для блока 66-73: [LVI]-S-G-E-[VI]-[LITV]-[EA]-[VI].
Выравнивание с негомологичной последовательностью
Для данного задания я использовала последовательность белка пыльцы-аллерегена (Uniprot ID:
ALL1_OLEEU), очевидно не гомологичную пяти последовательностям, исследуемым ранее. Редактируя
выравнивание, я нашла некоторое количество "консервативных" колонок, но тем не менее видно, что
качество выравнивания очень низкое.

Процент консервативных позиций в блоках после добавления негомологичной последовательности, естественно, снизился.
- В первом блоке нашлась одна абсолютно консервативная колонка (25%) против двух (50%) до добавления, одна абсолютно функционально консервативная (25%), против трех (75%) до добавления.
- Во втором блоке - 0 абсолютно консервативных (0%) и 2 абсолютно функционально консервативных (25%) против 3 (33,3%) и 7 (88,9%) до добавления.
- В третьем блоке - 1 абсолютно консервативная (9,1%) и 2 абсолютно функционально консервативные (18,2%) против 4 (36,4%) и 5 (45,5%) до добавления.
- В четвертом блоке - 0 абсолютно консервативных колонок (0%) и 2 абсолютно функционально консервативные колонки (25%) против 3 (37,5%) и 6 (75%) до добавления.
- В пятом блоке - 0 абсолютно консервативных колонок (0%) и 1 абсолютно функционально консервативная колонка (11,1%) против 4 (44,4%) и 5 (55,6%) до добавления.
Выравнивание заведомо неродственных белков
Я выбрала 5 случайных белков из списка и создала "выравнивание" согласно предыдущей схеме.
| Uniprot ID | Uniprot AC | Название белка | Организм |
|---|---|---|---|
| ISCS2_ARCFU | O29689 | Cysteine desulfurase IscS 2 | Archaeoglobus fulgidus DSM 4304 |
| Q8AAN6_BACTN | Q8AAN6 | Phenylacetate-coenzyme A ligase | Bacteroides thetaiotaomicron VPI-5482 |
| C6XF58_LIBAP | C6XF58 | Periplasmic solute binding protein | Candidatus Liberibacter asiaticus str. psy62 |
| Q9RUU2_DEIRA | Q9RUU2 | DNA helicase RecQ | Deinococcus radiodurans R1 |
| PHNS_DESVM | P21853; B8DPE1 | Periplasmic [NiFe] hydrogenase small subunit | Desulfovibrio vulgaris str. 'Miyazaki F' |
В полученном "выравнивании" не нашлось ни одного участка, удовлетворяющего введенному нами определению блока. Из 737 колонок колонок только 12 можно назвать абсолютно функционально консервативными (1,6%), из них только 2 (0,3%) абсолютно функционально консервативные.
В целом, выравнивание очень низкого качества: в нем мало консервативных позиций и много гэпов. Все это свидетельствует об отсутствии гомологии.

Ссылка на JalView проект, выравнивание негомологичных последовательностей
[1]. База знаний по биологии человека