Сравнение структур 3-х форм ДНК
С помощью инструментов пакета 3DNA были построены модели
структур A-, B- и Z-формы ДНК. Для визуального определения больших и малых бороздок
структуры были открыты в JMol. В каждой из структур было выбрано заданное мне основание -
тимин, и для него были определены атомы, обращенные в сторону большой или малой бороздки.
| Атомы тимина в трех формах ДНК | |||
|---|---|---|---|
| A-форма | B-форма | Z-форма | |
| В сторону большой бороздки обращены атомы: | T31.N3, T31.C4, T31.C5, T31.C5M, T31.C6, T31.C7, T31.O4 | T31.N3, T31.C4, T31.C5, T31.C5M, T31.C6, T31.C7, T31.O4 | - |
| В сторону малой бороздки обращены атомы: | T31.C2, T31.O2 | T31.C2, T31.O2 | - |
| Остальные атомы основания: | T31.N1 | T31.N1 | - |
С помощью MarvinSketch были получены изображения основания, где красным цветом выделены атомы, смотрящие в сторону большой бороздки, а синим в сторону малой.
| Изображения оснований в MarvinSketch | ||
|---|---|---|
| A-форма, Т31 | B-форма, Т31 | |
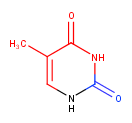 |  | |
Сравнение основных спиральных параметров разных форм ДНК
| A-форма | B-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16.97 ([DC]36:B.P #720 - [DC]8:A.P #146) | 17.21 ([DC]4:A.P #64 - [DA]34:B.P #679) | 16.08 ([DC]36:B.P #720 - [DC]12:A.P #228) |
| Ширина малой бороздки (Å) | 7.98 ([DT]3:A.P #44 - [DC]32:B.P #638) | 11.69 ([DC]36:B.P #720 - [DG]9:A.P #165) | 7.2 ([DG]11:A.P #206 - [DG]41:B.P #821) |
Сравнение торсионных углов в структурах А- и В-форм
Для начала торсионные углы нуклеотида [DT]31:B в A- и B-формах были измерены с помощью JMol.
| Торсионные углы разных форм ДНК в градусах | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
В следующем задании было необходимо сравнить торсионные углы в структурах А-, В- и Z-форм ДНК с
помощью пакета 3DNA. Поскольку на данный момент он работает только со старым PDB форматом,
нужные файлы были предварительно переведены в старый формат программой remediator.
Анализ структур нуклеиновых кислот (А-, В- и Z-форм ДНК, тРНК с PDB ID 1i9v и ДНК с PDB ID
1mdm) был проведен с помощью программ find_pair и analyze, в результате
работы которых был получен ряд файлов с описанием различных структурных параметров. Интересующие
нас данные, а именно значения торсионных углов и параметры водородных связей, находятся в файлах
формата XXXX.out.
Средние значения каждого из торсионных углов (без рассмотрения краевых нуклеотидов)
были найдены в программе Excel (ссылка на файл с расчетами).
| Средние значения торсионных углов в градусах | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| Z-форма (гуанин) | 51.9 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
| Z-форма (цитозин) | -139.5 | -136.8 | 50.9 | 137.6 | -96.5 | 82.0 | -154.3 |
| тРНК (PDB ID 1i9v) | -55.4 | 107.1 | 64.7 | 84.2 | -144.2 | -75.5 | -148.5 |
| ДНК (PDB ID 1mdm) | -47.3 | 0.6 | 33.4 | 139.6 | -70.3 | -95.4 | -108.9 |
Справнив полученные данные для трех форм ДНК, можно сделать вывод, что торсионные углы в А- и В-формах различаются, но в целом более менее схожи (наибольшие различия обнаружены в углах ζ - 85.4 градуса и δ - 64.2 градуса, наименьшее - в угле ε - 7.0 градусов). В случае сравнения Z-формы с формами А и В наблюдаются уже гораздо более значительные отличия практически по всем позициям. Для гуанина в наибольшей степени отличаются углы γ и χ, для цитозина - β и ζ.
Заданная структура тРНК наиболее схожа с А-формой.
Наиболее "деформированный" нуклеотид в тРНК - 24 цитозин первой цепи. Он больше всех остальных отклоняется по торсионному углу α , а также в значительной степени по углам β и γ.
Структура водородных связей
Определить номера нуклеотидов, образующих стебли (stems) во вторичной структуре заданной тРНК
можно на основании данных файла 1i9v.out. Такие нуклеотиды в нем идут
по порядку. Изображение РНК было получено с помощью JMol. Красным цветом выделен акцепторный
стебель, зеленым - Т-стебель, желтым - D-стебель, фиолетовым - антикодоновый стебель.
Остальные части окрашены в белый.
В таблице справа в те же цвета окрашены соответствующие строки с перечислением номеров атомов.
| Красный - акцепторный стебель Зеленый - Т-стебель Желтый - D-стебель Фиолетовый - антикодоновый стебель Белый - остальные структуры |
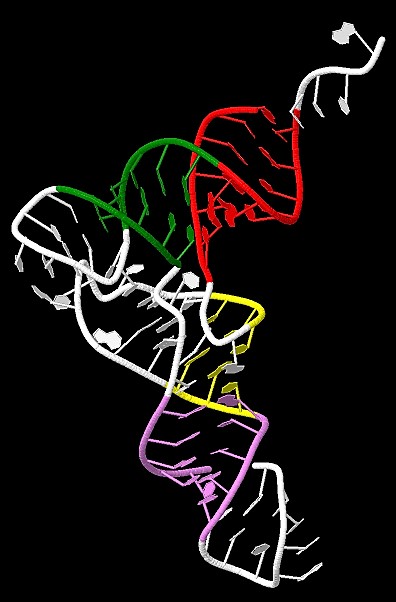 |
 |
Помимо стеблей в данной тРНК есть дополнительные водородные связи, стабилизирующие ее третичную структуру. Они могут образовываться в комплементарных парах
- A:54:[5MU]-[A]:58:A
- A:36:[A]-[U]:33:A
- A:14:[A]-[U]:8:A
- A:15:[G]-[C]:48:A
- A:19:[G]-[C]:56:A
- A:...4_:[..G]G-**--U[..U]:..69_:A
- A:..54_:[5MU]t-**--A[..A]:..58_:A
- A:..38_:[..A]A-**--C[..C]:..32_:A
- A:..44_:[..A]A-**--G[..G]:..26_:A
Стекинг-взаимодействия
Для нахождения возможных стекинг-взаимодействий в файле 1i9v.out
были найдены данные о величине площади "перекрываний" 2-х последовательных пар азотистых
оснований.
Пара с наибольшим значением:
- 12. Gt/AC 8.29( 3.00) 0.00( 0.00) 0.00( 0.00) 3.56( 0.99) 11.85( 3.99)
- 13. tA/UA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
- 14. AA/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
- 15. AC/GC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
- 20. AG/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
- 26. GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
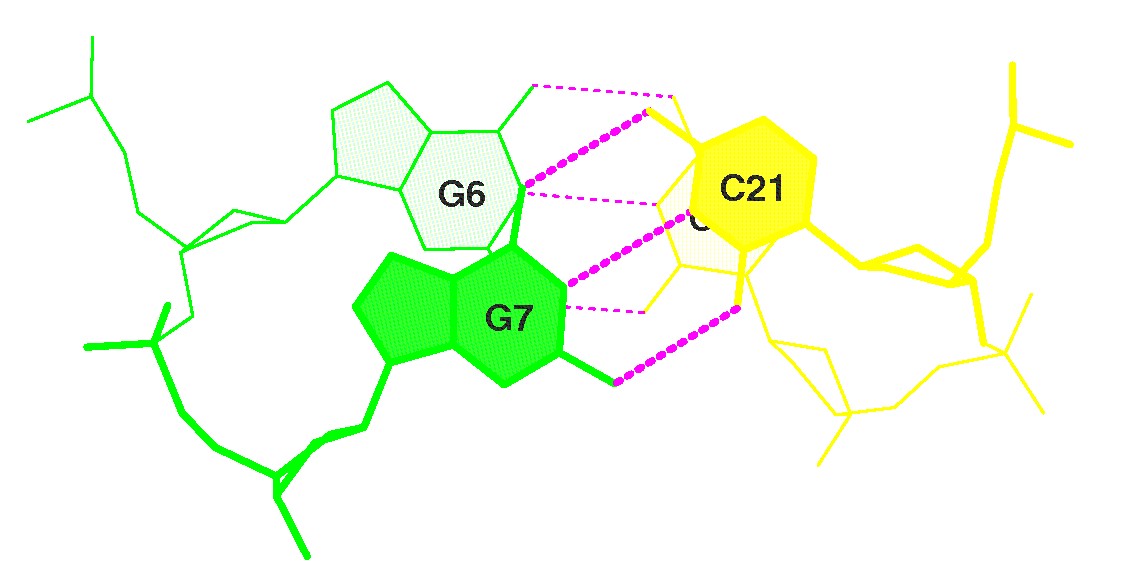
| Стэкинг-взаимодействия между парами нуклеотидов тРНК c наибольшим перекрытием | |
|---|---|
| stack2im | JMol |
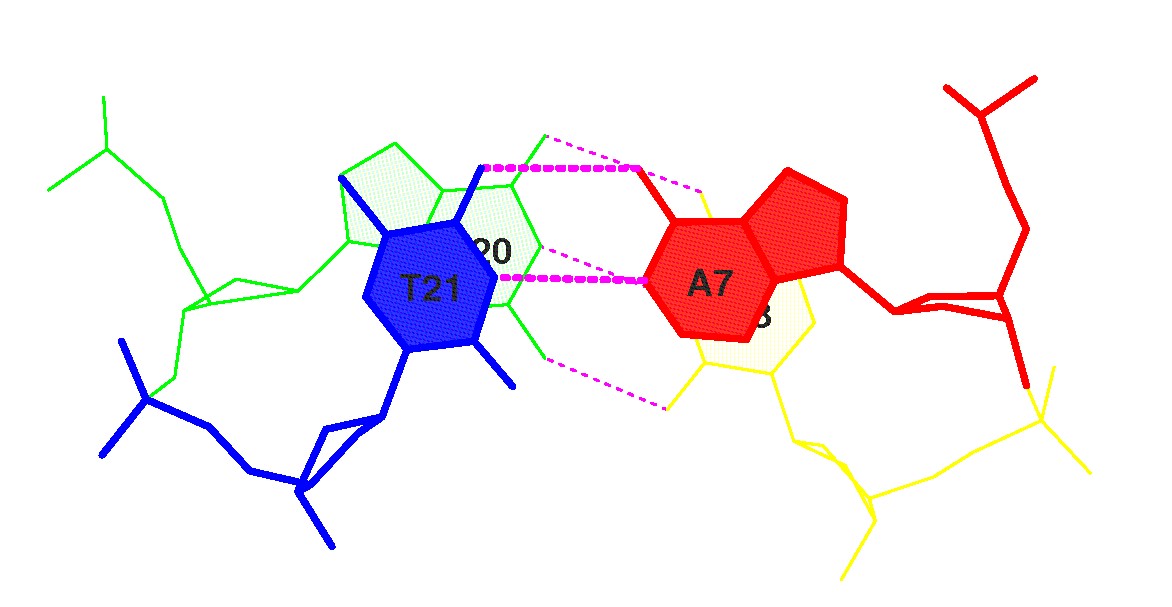 |
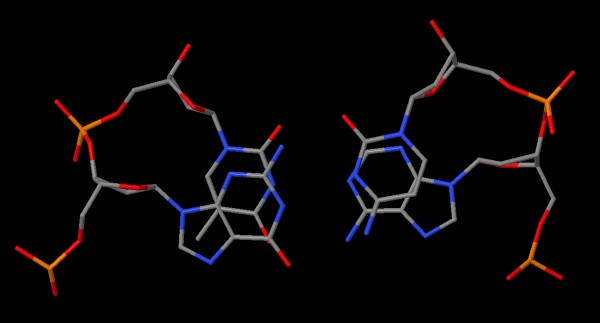 |
| Пары нуклеотидов тРНК c меньшим перекрытием | |
|---|---|
| stack2im | JMol |
 |
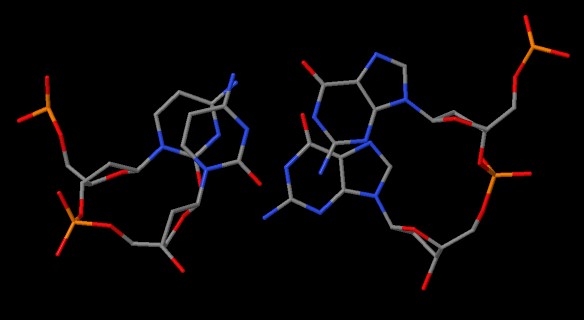 |