Предсказание генов прокариот
 |
| C. perfringens, окраска по Грамy |
Clostridium perfringens - вид грамположительных, строго анаэробных спорообразующих бактерий рода клостридий. Санитарно-показательный организм. Является возбудителем пищевых токсикоинфекций человека и одним из возбудителей газовой гангрены. Синтезирует протеиназы, лецитиназу, коллагеназу, гиалуронидазу и другие ферменты агрессивности.
Открыта в 1892 году Уэлчем и Нетталом. [1].
Работа с EMBOSS
Необходимые файлы с генами я получила с помощью пакета EMBOSS.
Командой seqret embl:cp013043 plasmid.fasta был получен файл
plasmid.fasta для последующей работы в Prodigal.
Командой seqret embl:cp013043 -feature plasmid.gff был получен файл
plasmid.gff c сохраненными особенностями для последующего
извлечения информации о реальных белок-кодирующих последовательностях.
Предсказание Prodigal
Для предсказания генов в выданной плазмиде я скачала программу Prodigal и запустила ее из
командной строки командой:

Параметры -i и -o аннотируют входной и выходной файлы соответсвенно, а параметр -f
указывает на желаемый формат выходного файла. Согласно рекомендациям был использован минималистичный
формат sco для удобства дальнейшей работы.
Полученный файл: plasmid.pro.
Обработка и сравнение файлов
Для сравнения предсказанных генов необходимо было сопоставить информацию, содержащуюся в файлах
plasmid.pro и plasmid.gff.
Для удобства была проведена предварительная обработка файлов, которая привела их к единому простому
формату. Это было сделано с помощью Python.
Скрипт attempt.py
перевел файл plasmid.gff в
true_cds.txt, содержащий только координаты и ориентацию белок-кодирующих генов,
аннотированных Genbank.
Скрипт attempt2.py перевел файл
plasmid.pro в pro_cds.txt, содержащий ту же информацию,
но предсказанную Prodigal.
Далее был написан скрипт final_attempt.py, сравнивающий
true_cds.txt и pro_cds.txt.
Общий алгоритм работы следующий:
скрипт принимает на вход файл с реальными и предсказанными генами, читает их по строкам, разбивая
каждую по знаку табуляции. В каждой строке первая позиция - координаты начала, вторая - координаты
конца, третья - ориентация. Заводятся счетчики для полностью совпавших генов, генов, совпавших
только по C-концу и только по N-концу. Для каждого файла происходит присвоение значений координат
N- и C- концов. Если в третьей позиции стоит "+", то последоваетельность координат не меняется, если "-", то
заменяется на противоположную. Затем для каждой строчки каждого файла происходит сравнение значений
координат по C- и N- концам. Если обе координаты совпадают, то к счетчику полностью совпадающих генов
прибавляется единица. В противном случае проверяется, не совпадают ли координаты по другим концам
(не одновременно), и в случае успеха, к соответсвующим счетчикам прибавляется единица.
В конце из длины файла с реальными генами (т.е. общего количества всех реальных генов) вычитаются
значения, поученные на всех счетчиках, что дает нам количество генов, которые совсем не были
предсказаны. В итоге на экран выводятся значения: общее число генов, количество и процент полностью
предсказанных генов, количество и процент генов, предсказанных с ошибкой по С-концу,
количество и процент генов, предсказанных с ошибкой по N-концу, количество и процент генов,
не предсказанных вовсе.
Результаты
| Предсказание генов в базе данных и программой Prodigal | |
|---|---|
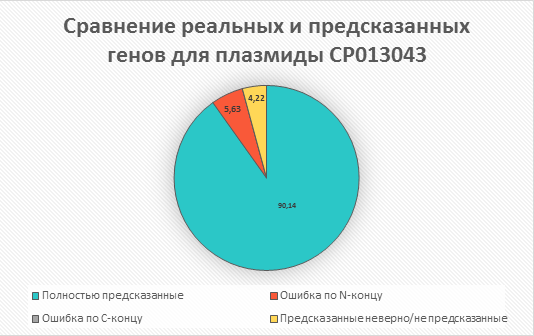
| Число генов, совпадающих обоими концами | 64 |
| Процент генов, совпадающих обоими концами | 90,14% |
| Число генов с неверным N-концом | 4 |
| Процент генов с неверным N-концом | 5,63% |
| Число генов с неверным C-концом | 0 |
| Процент генов с неверным C-концом | 0,00% |
| Число несовпавших генов | 3 |
| Процент несовпавших генов | 4,22% |
Причины несовпадения
В целом для плазмиды CP013043 предсказания Prodigal оказались довольно точными - полностью верно
предсказаны более 90% генов. Ошибок в предсказаниях по C-концу не встретилось, однако было выявлено
4 ошибки в N-концах. Попробуем на конкретных случаях понять причину этих ошибок.
Расссмотрим ген с координатами 45540-46907 (+). Продуктом данного гена является белок, инициирующий репликацию плазмиды.
В реальности он оказался короче, чем было предсказано Prodigal. Программа включила 9 лишних нуклеотидов на N-конце.
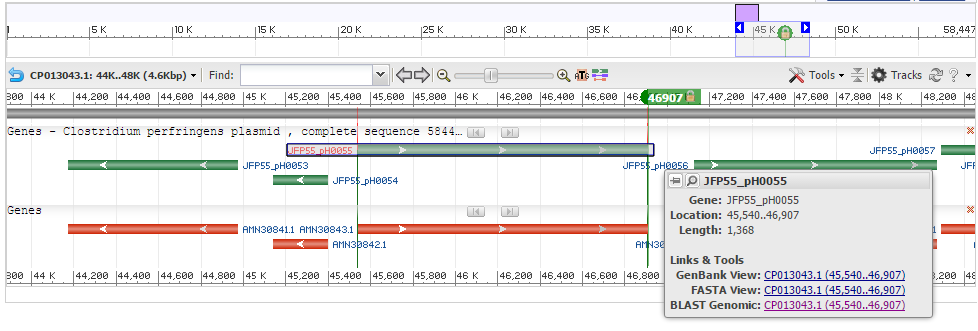 |
| Изображение гена JFP55_pH0055 в геномном браузере |
| The Bacterial, Archaeal and Plant Plastid Code |
|---|
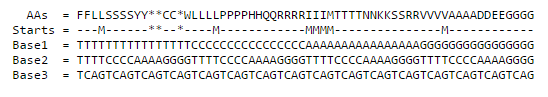 |
| Комментарий из источника: таблица 11 используется для Бактерий, Архей, прокариотических вирусов и хлоропластных белков. Наиболее эффективна инициация по стартовому кодону AUG. Для Архей и Бактерий были задокументированы стартовые кодоны GUG и UUG. В E. coli, UUG служит инициатором для приблизительно 3% белков, а CUG известен как инициатор одного закодированного в плазмиде белка (RepA). В дополнение к инициации через NUG, в редких случаях у Бактерий может наблюдаться инициация трансляции кодоном AUU, как, например, в случае poly(A) полимеразы PcnB и гена InfC, кодирующего фактор инициации трансляции IF3. |
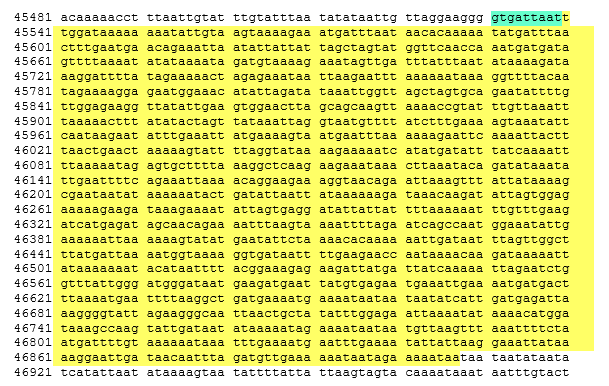 |
| Рисунок 1. Реальные и предсказанные границы гена JFP55_pH0055 |
При трансляции гена использовалась таблица генетического кода 11 (The Bacterial, Archaeal and Plant Plastid Code).
Как видно из этой таблицы, для исследуемой мной плазмиды возможны вариации в стартовых кодонах, что и могло повлечь за собой ошибку предсказания Prodigal.
На рисунке 1 желтым цветом выделены реальные границы гена, а зеленым - лишние предсказанные нуклеотиды. В зеленой области мы видим сразу 2 потенциальных стартовых кодона - gtg и att. Это, вероятно, и заставило программу ошибочно включить данный участок. В реальности же стартовым кодоном здесь является ttg.
Рассмотрим теперь ген с координатами 32407-32721 (-). Он был также неверно предсказан по N-концу, причем
Prodigal включил сразу 63 лишних нуклеотида. Продукт гена - гипотетический белок. Для трансляции использовалась та
же таблица 11.
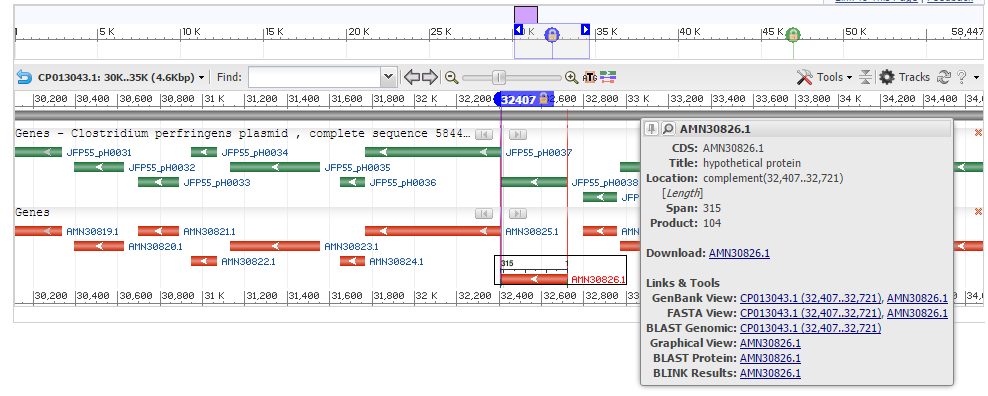 |
| Изображение гена (CDS) AMN30826.1 в геномном браузере |
 |
| Рисунок 2. Реальные и предсказанные границы гена JFP55_pH0055 |
Предполагаемая причина ошибки Prodigal сходна. Как видно из рисунка 2, кодон, с которого начинается ошибочно включенный фрагмент, представляет из себя наиболее распространенный стартовый кодон - aug (на рисунке - tac и обратный порядок, так как комплементарная цепь). Вероятно Prodigal посчитал именно его началом гена, хотя реальный ген тоже начинается с aug.
Сравнение предсказаний генов в GenBank и по данным Prodigal для геномной записи
В первом семестре мне был выдан организм Clostridium perfringens ATCC 13124 (полный геном -
NC_008261.1). По той же схеме, что и в предыдущем
задании, было произведено сравнение реальных и предсказанных генов для этого организма.
На вход программе final_attempt.py подавались обработанные файлы
organism_true_cds.txt и
organism_pro_cds.txt.
Эти же действия были выполнены для генома модельного организма Escherichia coli (полный геном -
NC_000913). Входные файлы -
ecoli_true_cds.txt и
ecoli_pro_cds.txt.
Получились следующие результаты:
| Предсказание в базе данных и програмой Prodigal для C.perfringens ATCC 13124 | |
|---|---|
| Число генов, совпадающих обоими концами | 2659 |
| Процент генов, совпадающих обоими концами | 94,49% |
| Число генов с неверным N-концом | 137 |
| Процент генов с неверным N-концом | 4,86% |
| Число генов с неверным C-концом | 18 |
| Процент генов с неверным C-концом | 0,81% |
| Число несовпавших генов | 0 |
| Процент несовпавших генов | 0,00% |
| Предсказание в базе данных и программой Prodigal для E.coli | |
|---|---|
| Число генов, совпадающих обоими концами | 3831 |
| Процент генов, совпадающих обоими концами | 87,35% |
| Число генов с неверным N-концом | 318 |
| Процент генов с неверным N-концом | 7,25% |
| Число генов с неверным C-концом | 75 |
| Процент генов с неверным C-концом | 1,71% |
| Число несовпавших генов | 162 |
| Процент несовпавших генов | 3,69% |
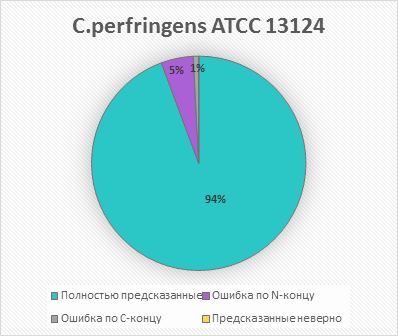
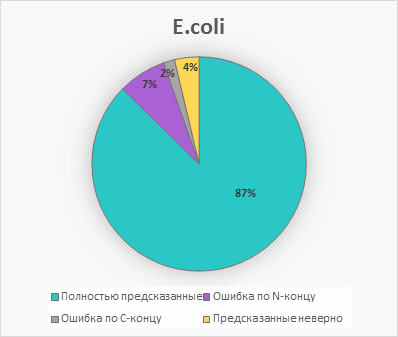
Как видно из полученных данных, предсказание для C.perfringens ATCC 13124 оказалось несколько точнее. Это поначалу показалось мне удивительным, так как я ожидала появления более точного результата именно для E.coli - популярного модельного организма. Тем не менее причина такого явления, как мне кажется, может заключаться в том, что геном E.coli почти в 2 раза длиннее, чем геном C.perfringens ATCC 13124 (3831 генов vs 2659 генов), следовательно, количество потенциальных мест для ошибок Prodigal больше.
Еще одна возможная причина - более правильно аннотированные гены для E.coli. Так как этот организм хорошо изучен, границы многих генов могли быть уточнены экспериментальными данными и исправлены относительно "сырого" предсказания. C.perfringens ATCC 13124 изучен менее подробно, поэтому некоторые гены в записи Genbank могут быть аннотированы лишь на основании предположений.
Источники