Предсказание генов эукариот
Описание контига NW_006890163
 |
| Callorhinchus milii, [2] |
|
Kingdom: Animalia
Phylum: Chordata Class: Chondrichthyes Subclass: Holocephali Order: Chimaeriformes Family: Callorhinchidae Genus: Callorhinchus Species: C. milii |
Callorhinchus milii относится к химерообразным, является филогенетическим реликтом и модельным организмом среди хрящевых рыб. Предполагается, что ДНК рыбы-слона может помочь разобраться, что повлияло на образование костяного скелета, поэтому Callorhinchus milii стала первой хрящевой рыбой, чей геном был полностью расшифрован [1].
В январе 2014 в журнале Nature была опубликована статья Why sharks have no bones?, авторы которой утверждают, что у Callorhinchus milii отсутсвует семейство генов, регулирующих процесс преобразования хряща в кость, и предполагают, что начало данному семейству генов дала дупликация, повлекшая за собой появление первых костных позвоночных [3].
Обитают Callorhinchus milii в Австралии на глубине 200-500 м, имеют размеры от 0,5-1,5 м. Наиболее выдающаяся морфологическая особенность - специальный вырост (хобот), который они используют для поиска пищи на дне. Рыба-слон плавает вблизи дна, удерживая подбородок в нескольких миллиметрах от его поверхности и качая головой из стороны в сторону. Все это происходит в условиях темноты или мутной воды. Поэтому у Callorhinchus milii весьма необычная чувствительная система, которая состоит из "фонарика" — группы клеток на хвосте, производящих электрические импульсы 80 раз в секунду, и "камеры" — чувствительных к электрическому полю клеток на коже подбородка. Они формируют картину окружающего мира по искажениям поля [4].
Контиг NW_006890163 имеет длину 2618064 bp, 73 гена и 40 белок-кодирующих последовательностей (CDS).
Последовательность контига в формате fasta: contig.fasta
Для гена slc37a1 базой данных предсказан альтернативный сплайсинг. Координаты этого гена - complement(751812..786779), длина - 34968, число экзонов -22. На рисунке 1 представлено геномное окружение этого гена, различные варианты транскрибируемых с него мРНК и соответсвующие им изоформы белков.
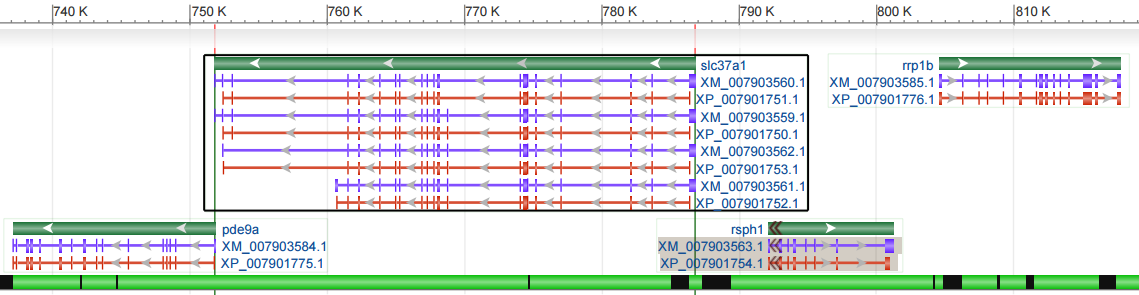 |
| Рис.1: Геномное окружение гена slc37a1 |
Видно, что для данного гена возможны 4 альтернативных варианта мРНК (синие полосы), причем некоторые из них разной длины. Данным мРНК соответсвуют 4 изоформы белка (красные полосы), являющегося глицерол-3-фосфат транспортером.
Этот белок принадлежит к суперсемейству MFS - major facilitator superfamily. Белки данного семейства способствуют транспорту различных субстратов (ионов, сахарофосфатов, лекарств, нейротрансмиттеров, нуклеозидов, аминокислот, пептидов) через цитоплазматические и внутренние мембраны по хемиоосмотическому градиенту. Унипортеры транспортируют единичные молекулы, а симпортеры и антипортеры - две молекулы субстрата в одном или противоположных направлениях соответственно. Белки MFS обычно состоят из 400-600 аминокислот и имеют 12 трансмембранных альфа-спиралей (TMs), связанных гидрофобными петлями. N- и С-концевые участки данных белков мало сходны и могут быть результатом генной дупликации или слияния генов.
Рисунок 2 показывает один из вариантов транскрибируемых мРНК ( XM_007903559.1 - Callorhinchus milii solute carrier family 37 (glucose-6-phosphate transporter), member 1 (slc37a1), transcript variant X1) и соответсвующую ему изоформу белка ( XP_007901750.1 - glycerol-3-phosphate transporter isoform X1 ).
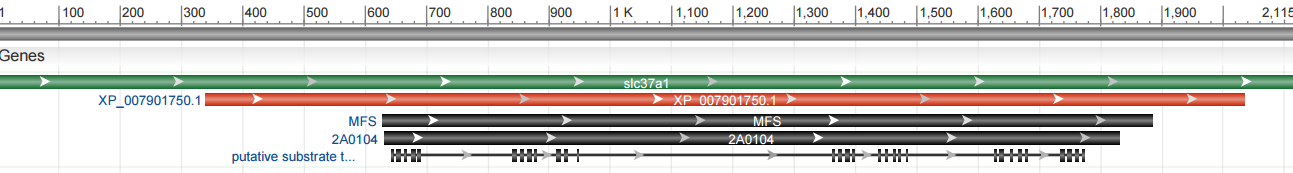 |
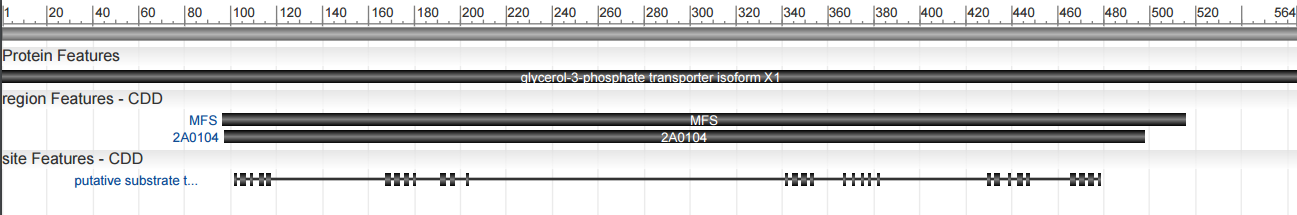 |
| Рис.2: Один из вариантов мРНК (transcript variant X1) и соответсвующая ей изоформа белка (isoform X1) |
На рисунке 3 представлено графическое изображение доменов, входящих в состав изоформы 1 (XP_007901750.1).
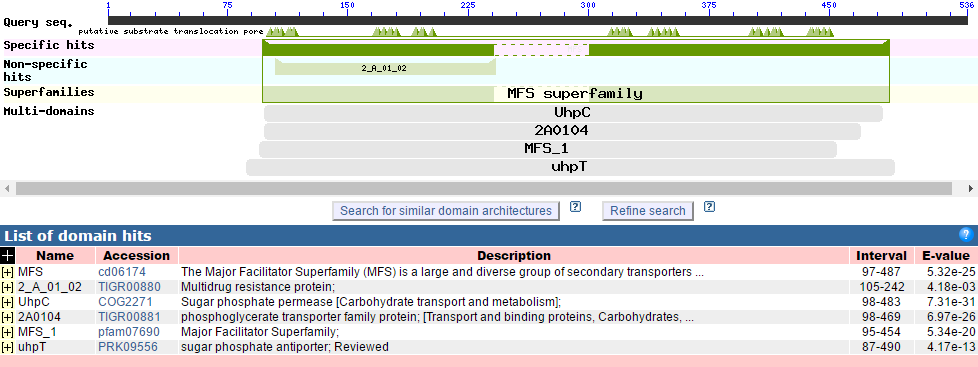 |
| Рис.3: Доменный состав изоформы XP_007901750.1 |
Предсказание AUGUSTUS
Далее дя выданного мне контига были предсказаны гены и белок-кодирующие области с помощью сервера
AUGUSTUS в режиме prediction.
Параметры запуска представлены ниже:
 В качестве параметров модели было необходимо использовать организм, наиболее близкий к моему, однако среди
предложенных программой оказался сам мой организм - Callorhinchus milii. Сначала я выбрала именно его, но
программа отказалась работать корректно, выдав всего 3 файла из 6, причем с пустым содержимым. Тогда я взяла
наиболее близкий организм из оставшихся - Danio rerio.
В качестве параметров модели было необходимо использовать организм, наиболее близкий к моему, однако среди
предложенных программой оказался сам мой организм - Callorhinchus milii. Сначала я выбрала именно его, но
программа отказалась работать корректно, выдав всего 3 файла из 6, причем с пустым содержимым. Тогда я взяла
наиболее близкий организм из оставшихся - Danio rerio.
Предсказание UTR аннонсирует нетранслируемые участки, а также, если верить мануалу AUGUSTUS,
делает предсказание кодирующих участков чуть более точным. Однако эта опция доступна не для всех организмов.
Для Danio rerio такой возможности пока не предусмотрено, поэтому значением данного параметра было
выбрано false, чтобы ускорить работу программы.
Так как по данным предыдущего задания в анализируемом файле есть альтернативные транскрипты, то была включена
учитывающая их опция на значение medium.
Остальные параметры были взяты по умолчанию (предсказывать гены на обеих цепях, предсказывать любое число полных/частичных
генов, не игнорировать конфликты с комплементарной цепью).
В результате AUGUSTUS выдал [tar.gz]-архив с 6 файлами:
| Расширение | Содержимое |
|---|---|
| *.gff | предсказание генов в формате gff - General Feature Format |
| *.gtf | предсказание генов в формате gft - 2-ой версии gff |
| *.gbrowse | трек-файл предсказания для GBrowse |
| *.aa | предсказание генов в виде fasta-последовательности белков |
| *.cdsexons | предсказание экзонов в виде fasta-последовательности ДНК |
| *.codingseq | предсказание генов в виде fasta-последовательности кодирующих участков ДНК |
Ссылка на файл с предсказаниями генов: augustus.gff.
Оценка предсказания
AUGUSTUS предсказал 219 генов, в то время как в файле GenBank аннотировано всего 73 гена.
При помощи программ Python,
аналогичным тем, что использовались в предыдущем практикуме, я провела сравнение предсказания с действительностью.
Программа contig.py перевела последовательность контига в формате gff
(contig.gff) и предсказание augustus (augustus.gff) в
файлы contig1.txt и augustus1.txt, содержащие
только координаты и ориентацию генов, которые затем были сопоставлены программой
augustus.py.
Выдача программы:
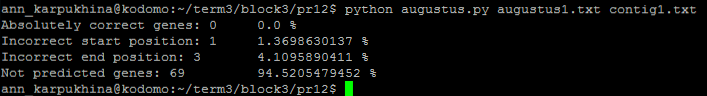
В результате выяснилось, что предсказание AUGUSTUS получилось совсем
некачественным. Ни одного гена не было предсказано полностью правильно, 1 ген был предсказан с ошибкой в координате начала,
и три - с ошибкой в координате конца. Также было предсказано 46 лишних генов. Очевидно, что данный результат совсем не
удовлетворительный. Возможной причиной такого несовпадения мне видится то, что Callorhinchus milii сам является
модельным организмом, на сравнение с которым натренирован AUGUSTUS, и проводить предсказание для него на основании
другого организма не совсем корректно. Вероятно также, что Danio rerio и Callorhinchus milii недостаточно
близки.
Так как ни одного гена не было предсказано правильно, то понятно, что и для исследуемого в задании 1 гена slc37a1
предсказание оказалось неверным. Среди предсказанных наиболее "похожим" на наш оказался ген с координатами
complement(761504 - 771797), однако это, на мой взгляд, вообще нельзя считать предсказанием, так как ошибка слишком
велика. "Предсказанный" ген короче на 24674 bp, и гены окружения тоже предсказаны неверно. Поэтому все печально и
сравнивать нечего =(

Источники