Банки нуклеотидных последовательностей
Качество сборки генома эукариотического организма
 Я выбрала эукариотический организм Calypte anna (Anna's hummingbird).
Я выбрала эукариотический организм Calypte anna (Anna's hummingbird).
Калипта Анны, или колибри Анны — вид птиц семейства колибри, получил название
в честь герцогини Анны Массена.
Калипта Анны имеет длину примерно 10 см, размах крыльев 11,4 - 12,1 см,
вес 4 - 4,5 г.
Живёт на западе Северной Америки на тихоокеанском побережье.
Питается преимущественно нектаром цветов и пыльцы,
которые собирает из цветков, зависая в воздухе, а также насекомыми.
Относительно длины своего тела калипта Анны является самым быстрым
позвоночным животным мира. Bо время брачных полётов птицы достигают скорости
385 размеров тела в секунду (98,28 км/ч), испытывая перегрузки примерно в 10g.
[1], [2].
Для данного организма пока есть только одна сборка генома - GCA_000699085.1 (
ASM69908v1).
Проектов по секвенированию генома 2 (PRJNA253828 и PRJNA212866), но они связаны между собой.
Оба были зарегистрированы 6 мая 2014 года. Сборка RefSeq (проект PRJNA253828)
была заимствована из загруженной сборки GeneBank (проект PRJNA212866). Аннотирование было основано
на результатах NCBI annotation pipeline. Секвенирование генома было произведено
организацией BGI в целях иузучения
эволюции.
Количество образцов - 1 (BioSample:
SAMN02265252; Sample name: BGI_N300; SRA: SRS464260). Образец
был взят у самки калипты Анны в США.
 Для единственной имеющейся сборки ASM69908v1:
Для единственной имеющейся сборки ASM69908v1:
- Длина полследовательности - 1,105,676,412
- Гэпов между скэффолдами - 0
- Число скэффолдов - 54,736
- Scaffold N50 - 4,052,191
- Scaffold L50 - 81
- Число контигов - 124,820
- Contig N50 - 26,738
- Contig L50 - 11,601
- Общее число хромосом и плазмид - 0
- Таблица контигов
- Самый длинный контиг - 303710 ( JJRV01046186)
- Самый короткий контиг - 200 ( JJRV01000111)
- Последовательность контига JJRV01000007 (contig7)
Ключи в таблицах особенностей
Последовательности нуклеиновых кислот предоставляют фундаментальную стартовую возможность для описания и понимания структуры, функций и развития генетически разнообразных организмов. Нуклеотидные банки GenBank, EMBL, and DDBJ с самого своего основания использовали таблицы сайтов и особенностей для описания роли и расположения наиболее высокоорганизованных доменов последовательностей и элементов генома организма. В феврале 1986 года GenBank и EMBL начали совместную работу по созданию общего формата таблиц особенностей и универсальных стандартов аннотирования последовательностей. В 1987 году к ним присоединился DDBJ.
Глобальная цель языка таблиц Feature Table - предоставить обширный запас обозначений для описания
включает общие для трех вышеперечисленных баз данных правила, которые позволяют им
безпрепятсвенно обмениваться данными на регулярной основе.
Список представленных особенностей крайне разнообразен. Описываются участки, которые:
- выполняют определенную биологическую функцию
- влияют на определенную биологическую функцию или являются результатом ее экспрессии
- взаимодействуют с другими молекулами
- влияют на репликацию последовательности
- являются результатом рекомбинации различных пследовательностей или влияют на рекомбинацию
- являются типичным повторяющимся фрагментом
- имеют вторичную или третичную структуру
- демонстрируют вариабельностьили были отредактированы/исправлены
Формат записей основывается на табличном подходе и включает следующие пункты:
- Feature key (ключ особенности) - одно слово или аббревиатура, указывающие на функциональную группу
- Location (расположение) - инструкции, указыващие где найти данную особенность
- Qualifiers (спецификатор) - вспомогательная информация
Во всех возможных для этого случаях в таблицах особенностей используется общепринятая биологическая
терминология. К примеру, такой пункт из таблицы особенностей

может быть прочтен следующим образом: Ообенность CDS - кодирующая последовательность, начинающаяся
с основания 23 и заканчивающаяся на основании 400, имеет продукт "алкогольдегидрогеназа" и
закодирована в гене "adhI".
Далее приведено описание еще 10 произвольно выбранных ключей таблиц особенностей с примерами.
Feature Key source.
Указывает биологический источник для обозначенного участка последовательности. Является
обязательным. Возможно существование нескольких таких ключей для одной и той же
последовательности. Каждая запись должна иметь либо один source key, верный для всей
последовательности, либо несколько, которые при объединении своих участков дают целую
последовательность.

Feature Key regulatory
Указывает на любой участок, принимающий участие в регуляции трансляции или транскрипции.
Обязательным спецификатором для этого ключа является регуляторный класс (/regulatory_class="TYPE").
15 декабря 2014 года этот ключ заменил следующие ключи: enhancer, promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal,
RBS, GC_signal, polyA_signal, attenuator, terminator, misc_signal.

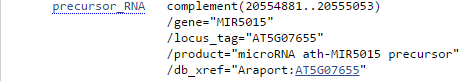
Feature Key precursor_RNA
Любой образец РНК, который еще не является зрелым РНК-продуктом. Может включать мРНК, рРНК,
некодирующие РНК, 5′-нетранслируемые области (5'UTR), кодирующие последовательности (CDS, exon),
интроны, 3′-нетранслируемые области(3'UTR). Ключ используется для РНК, которые могут являться
результатом посттранскрипционных модификаций. Если известно, что данная РНК не была модифицирована,
то используется ключ prim_transcript.
Feature Key V_region
Вариабельные участки легких и тяжелых цепей иммуноглобулинов, α-, β- и γ-цепей Т-клеточных рецепторов.
Кодирует вариабельные N-концы. Может состоять из V_segments, D_segments, N_regions и J_segments.
Ключ используется только для последовательностей эукариотических организмов.

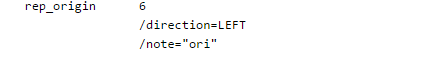
Feature Key rep_origin
Указывает на ориджин репликации. В спецификаторе может указываться направление репликаци
от точки ее начала (RIGHT, LEFT, or BOTH).
Feature Key D-loop
Указывает на наличие D-петли - области митохондриальной ДНК, в которой небольшой участок РНК
взаимодействует с одной из цепей ДНК, вытесняя исходную комплементарную цепь с образованием
петлеообразной структуры. Также используется для описания смещения участка одной из цепей
ДНК-дуплекса из-за внедрения одноцепоченой нуклеиновой кислоты в реакции, катализируемой
белком RecA. Ключ используется только для ДНК-последовательностей.

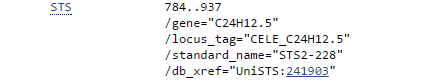
Feature Key STS
Указывает на STS (sequence tagged site) - короткие участки с известной нуклеотидной
последовательностью, которые встречаются в ДНК только один раз и могут быть легко обнаружены
при помощи ПЦР со специфическими праймерами. Используются как ориентиры при составлении
генетических карт. Участок генома может быть картирован в результате определения порядка
серии из нескольких STS. Ключ используется только для ДНК-последовательностей.
Feature Key polyA_site
Указывает на РНК-транскрипт, к которому будут добавлены остатки аденина в ходе посттранскрипционного
полиаденилирования. Используется толкько в последовательностях эукариот и эукариотических
вирусов.

Feature Key mat_peptide
Указывает на последовательность, кодирующую зрелый, проходящий посттрансляционную модификацию,
пептид или белковый продукт, не включая стоп-кодон (в отличие от соответсвующей CDS).

Feature Key introne
Указывает на сегмент ДНК, который удаляется из первичного транскрипта и отсутсвует в зрелой
РНК.

Описание геномного проекта
MSSNG - совместный проект Google и организации Autism Speaks, запущенный в 2014 году и
нацеленный на создание самой большой в мире геномной базы данных по аутизму. Планируется
отсеквенировать ДНК более 10000 семей в той или иной степени подверженных заболеванию.
Благодаря Google Cloud эти обширные массивы данных будут находиться в свободном доступе для
исследователей по всему миру.

Реализованная ранее пилотная программа по секвенированию 1000 полных геномов уже привела
к некоторым новым открытиям, важным для понимания патологии аутизма. Теперь, увеличив базу
до 10000 геномов и заручившись поддержкой глобального научного сообщества, Autism Speaks
надеется добиться еще более значительных результатов. В частности, планируется выделить
отдельные подтипы аутизма для получения возможности более персонифицированного и точного
лечения.
На данный момент уже доступны данные для 5194 индивидов (всего 5205 образцов геномов).
Из них 2621 - больные аутизмом (2063 мужчин и 558 женщин), 2573 (1282 мужчин и 1291 женщин) -
близжайшие родственники больных. Всего исследовано 2077 семей, преимущественно состоящих из
трех (здоровые родители и больной ребенок) или четырех (здоровые родители и двое больных детей)
человек. Более подробно о доступных данных, их местонахождении, формате и условиях доступа можно
прочитать на странице с официальной документацией.
Посдедняя из опубликованных статей, связанных с проектом:
Genome-wide characteristics of de novo mutations in autism.
 Организация Autism Speaks, явившаяся основным инициатором данного проекта, была основана в 2005 году супругами Райт,
внук которых страдал аутизмом. К настоящему времени она является ведущей в сфере научного изучения аутизма,
юридической, финансовой и психологической поддержки страдающих от данного недуга семей.
Ее главный офис находится в Нью-Йорке, США, однако представительства организации имеются во многих странах по
всему миру.
Организация Autism Speaks, явившаяся основным инициатором данного проекта, была основана в 2005 году супругами Райт,
внук которых страдал аутизмом. К настоящему времени она является ведущей в сфере научного изучения аутизма,
юридической, финансовой и психологической поддержки страдающих от данного недуга семей.
Ее главный офис находится в Нью-Йорке, США, однако представительства организации имеются во многих странах по
всему миру.
Таблица митохондриальных генов
Было необходимо составить таблицу митохондриальных генов для организма из таксона
Stramenopiles. Чтобы найти все полные митохондриальные геномы, в БД Nucleotide (NCBI) был
выполнен следующий поисковой запрос:
(((stramenopiles[Organism]) AND complete[Title]) AND mitochondrion[Title]) AND
genome[All Fields].
Найдено 155 результатов, из них 86 - GenBank, 69 - RefSeq.
 Я взяла организм Thalassiosira pseudonana - вид морских центрических диатомей. Этот организм
был первым среди морского эукариотического фитопланктона, для которого секвенировали полный геном.
Выбор был обусловлен его широкой распространенностью и относительно небольшим размером
генома. Thalassiosira pseudonana используется как модельный объект для изучения светопоглощения.
Модифицированная форма Thalassiosira pseudonana рассматривается как потенциальный посредник для доставки
плохо растворимых в воде лекарств для химиотерапии рака [5].
В своей клеточной стенке T. pseudonana имеет сложные кремнеземные структуры, интересные для нанотехнологии.
[6].
Я взяла организм Thalassiosira pseudonana - вид морских центрических диатомей. Этот организм
был первым среди морского эукариотического фитопланктона, для которого секвенировали полный геном.
Выбор был обусловлен его широкой распространенностью и относительно небольшим размером
генома. Thalassiosira pseudonana используется как модельный объект для изучения светопоглощения.
Модифицированная форма Thalassiosira pseudonana рассматривается как потенциальный посредник для доставки
плохо растворимых в воде лекарств для химиотерапии рака [5].
В своей клеточной стенке T. pseudonana имеет сложные кремнеземные структуры, интересные для нанотехнологии.
[6].
Для получения списка митохондриальных генов я перешла по ссылке gene в разделе
Related information. Cортировка списка была проведена в соответствии с порядком генов в геноме
(Sort by Chromosome). Всего был найден 61 ген, из них только 35 кодируют белки (Categories - Protein-coding).
В файле Excel представлены таблицы всех митохондриальных генов и отдельно
таблица только белок-кодирующих генов c идентификаторами белков.
Таблица размеров геномов
| Размеры геномов, п.н. | |||
|---|---|---|---|
| Минимальный | Типичный | Максимальный | |
| Вироиды [7] | 120 | ~350 | 475 |
| Вирусы [7] | 1759 (Porcine circovirus). | ~10^4 | 2400000 (Pandoravirus salinus) |
| Археи [8] | 0,49 млн (Nanoarchaeumeum equitans) | ~10^6 | 5,75 млн (Methanosarcina acetivorans) |
| Бактерии [8] | 0,58 млн (Mycoplasma genitalium) | ~10^6 | 9,2 млн (Bradhyrhizobium japonicum) |
| Эукариоты [9], [10] | 0,551 млн (Guillardia theta) | ~10^9, но очень сильно варьируется | 670 млрд (Amoeba dubia) |
Источники
- [1]. Anna's hummingbird, Wikipedia
- [2]. Digital Photography Review
- [3]. NCBI
- [4]. INSDC, The DDBJ/ENA/GenBank Feature Table Definition
- [5]. Thalassiosira pseudonana, Wikipedia
- [6]. Thalassiosira pseudonana, MicrobeWiki
- [7]. Virology Blog
- [8]. ГЕНОМ ПРОКАРИОТ, Н.В. Равин, С.В. Шестаков
- [9].Wikipedia: Smallest organisms
- [10]. Largest and Smallest Genome in the World