Прочтение последовательностей по Сэнгеру
В этом задании было необходимо прочитать ДНК последовательность, используя данные, полученные из капиллярного секвенатора по Сэнгеру. Капиллярный секвенатор по Сэнгеру выдает файлы с хроматограммой и автоматически прочтенной последовательностью в формате .ab1. Мне были даны два исходных файла:
Для просмотра хроматограмм и редактирования автоматического прочтения использовалась программа Chromas (Lite). В программе были открыты оба исходных файла, при этом файл с обратной цепью был переведен в комплементарную цепь с помощью команды Edit > Reverse+Complement . Затем для обоих последовательностей был настроен одинаковый масштаб по горизонтали и было произведено выравнивание двух последовательностей с использованием поиска подслов Find.| Границы не читаемых 5'- и 3'-участков (координаты по прямой последовательности): | ||
|---|---|---|
| Последовательность | 5'-участок | 3'-участок |
| Прямая | 1-139 | 672-713 |
| Обратная | 1-38 | 549-708 |
Важно заметить, что некоторые участки нечитаемы в одной последовательности, но при этом вполне приемлемо определяются в другой. Таким образом, объединяя результаты для двух цепей, можно получить наиболее подробное и точное определение нуклеотидной последовательности.
Оценка качества хроматограмм.
- Прямая цепь. В целом, хроматограмма достаточно приемлемого качества, в срединных участках сигнал в значительной степени (в десятки раз) превосходит шум, пики четкие. Средняя сила сигнала ≈ 1000. Разброс силы сигнала от нуклеотида к нуклеотиду относительно невелик, но можно заметить, что наиболее высокие пики наблюдаются для пуриновых оснований - гуанина и аденина. Силы сигнала и шума вдоль последовательности выражены неравномерно. Ближе к концам качество хроматограммы падает. Шум становится более выраженным, начинают появляться размытые и раздвоенные пики, особенно со стороны 3'-конца. Размеры нечитаемых конечных участков очень велики.
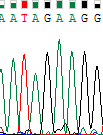
- Обратная цепь.Хроматограмма для обратной цепи показалась мне примерно такой же по качеству, как и для прямой. Пики в срединной части последовательности четкие, шум почти нигде не выходит на уровень сигнала. Средняя сила сигнала также около 1000, наиболее высокие пики дают пиримидиновые основания - цитозин и тимин. Качество краевых участков вновь значительно хуже. В начале и в конце наблюдается достаточно много размытых и раздвоенных пиков, более низкое соотношение сигнал/шум. Размеры нечитаемых концевых участков велики.
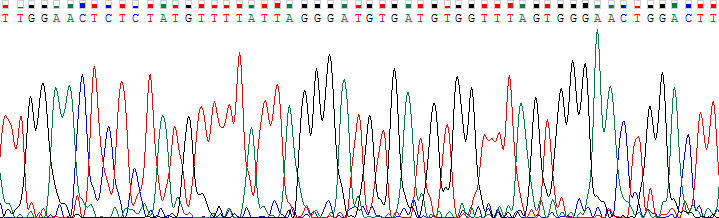 |
| Размытые и раздвоенные пики в начале хроматограммы для обратной цепи |
|---|
Далее было необходимо проанализировать последовательности и отредактировать их, сравнивая результаты двух хроматограмм и исправляя потенциально возможные ошибки программы. Все исправления внесены строчными буквами.
С помощью Jalview было получено выравнивание исправленных последовательностей (тех их частей, которые ранее были признаны читаемыми). На основании полученнных данных была составлена итоговая нуклеотидная последовательность.
- Исправленная прямая цепь
- Исправленная обратная цепь
- Итоговая последовательность в fasta-формате
- Jalview проект с выравниваним прочтений прямой и обратной последовательности
На рисунке можно увидеть изображение полученного выравнивания с раскраской по нуклеотидам. Проблемные нуклеотиды выделены строчными буквами. Явных полиморфизмов найдено не было.

Обоснование решений для проблемных нуклеотидов
На рисунках показаны некоторые из произведенных мною замен. В каждом из случаев верхняя хроматограмма
соответсвует прямой цепи, а нижняя - обратной.
| 1 | |
|---|---|
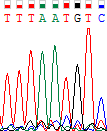
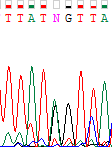 | Нуклеотид, обозначенный буквой N, был не определен программой в прямой цепи, так как в ней в этой позиции наблюдаются два достаточно четких и при этом почти одинаковых по высоте пика сразу для двух пуриновых оснований - аденина и гуанина. Здесь можно было бы предположить наличие полиморфизма. Однако в обратной последовательности ничего подобного не наблюдается, и присутсвует лишь один выраженный пик, соответствующий аденину. При этом количество шума минимально. Поэтому было принято решение поставить на эту позицию аденин. |
 | |
| 2 | |
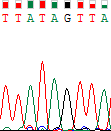
 | Программой не был определен один из нуклеотидов прямой цепи между аденином и тимином, так как наблюдаются сразу два сравнимых по высоте пика. Тем не менее пик для аденина в два раза выше, чем для гуанина, а в обратной цепи пик для гуанина вовсе отсутсвует и однозначно определяется аденин. Все это позволяет нам принять пик гуанина за шум и достаточно уверенно поставить аденин в прямую цепь. |
 | |
| 3 | |
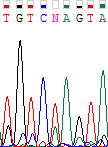 | Как и в предыдущем случае, нуклеотид прямой цепи не был определен из-за высокого, конкурирующего с сигналом, уровня шума. Наблюдаются сравнимые пики для аденина и тимина. Однако на обратной цепи уровень шума в этой позиции минимален и виден очень четкий сигнал для тимина. Поэтому в эту позицию был поставлен тимин. |
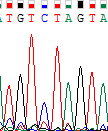 | |
| 4 | |
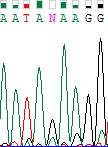 | Нуклеотид прямой цепи не был определен из-за слишком слабого сигнала, который почти не отличается от окружающего шума. Поэтому решение принималось на основании хроматограммы обратной цепи, в которой данный участок выглядит очень качественно. На интересующей нас позиции наблюдается очевидно выраженный пик для гуанина с сильным уровнем сигнала, соответственно и в прямую цепь был поставлен гуанин. |
 | |
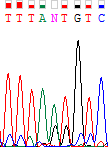
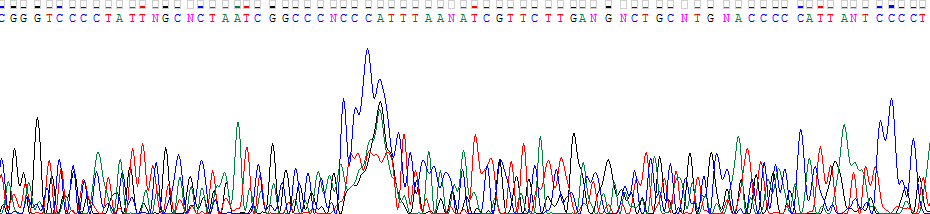
Пример плохой хроматограммы
На рисунке приведен фрагмент очень плохой хроматограммы. Уровень шума очень высок, практически в каждой
позиции наблюдается сразу несколько пиков, сравнимых по высоте, что свидетельствует о сильной
загрязненности образца. Вероятно, в нем было сразу несколько различных ДНК.
В центральной части - большие размытые пики, которые могут быть пятнами краски.
 |