Работа с KEGG ORTHOLOGY
Выбор пары ортологичных белков
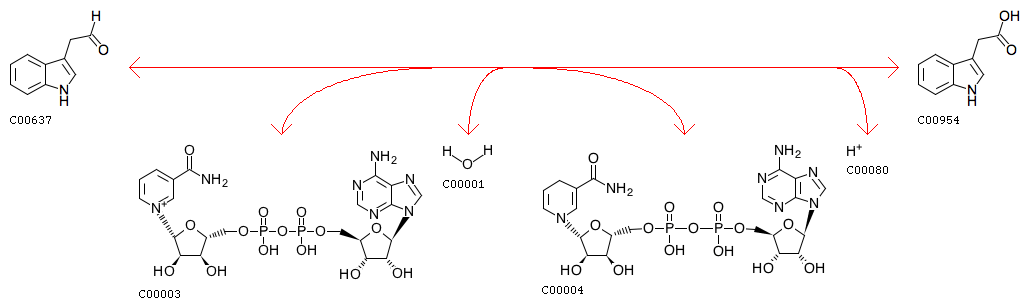 |
| Indole-3-acetaldehyde + NAD+ + H2O <=> Indole-3-acetate + NADH + H+ |
| Рис. 1. Выбранная реакция |
В нем была выбрана реакция EC 1.2.1.3 (Рис.1),катализируемая тремя ортологическими рядами белков:
- K00128 - aldehyde dehydrogenase (NAD+)
- K00149 - aldehyde dehydrogenase family 9 member A1
- K14085 - aldehyde
dehydrogenase family 7
member A1
| Таблица 1. Информация об ортологичных рядах | ||
| Идентификатор | Число белковых последовательностей | Число генов |
| K00149 | 57 (49 Uniprot) | 219 |
| K14085 | 134 (126 Uniprot) | 785 |
Необходимая информация о выбранных ортологичных рядах представлена в таблице 1.
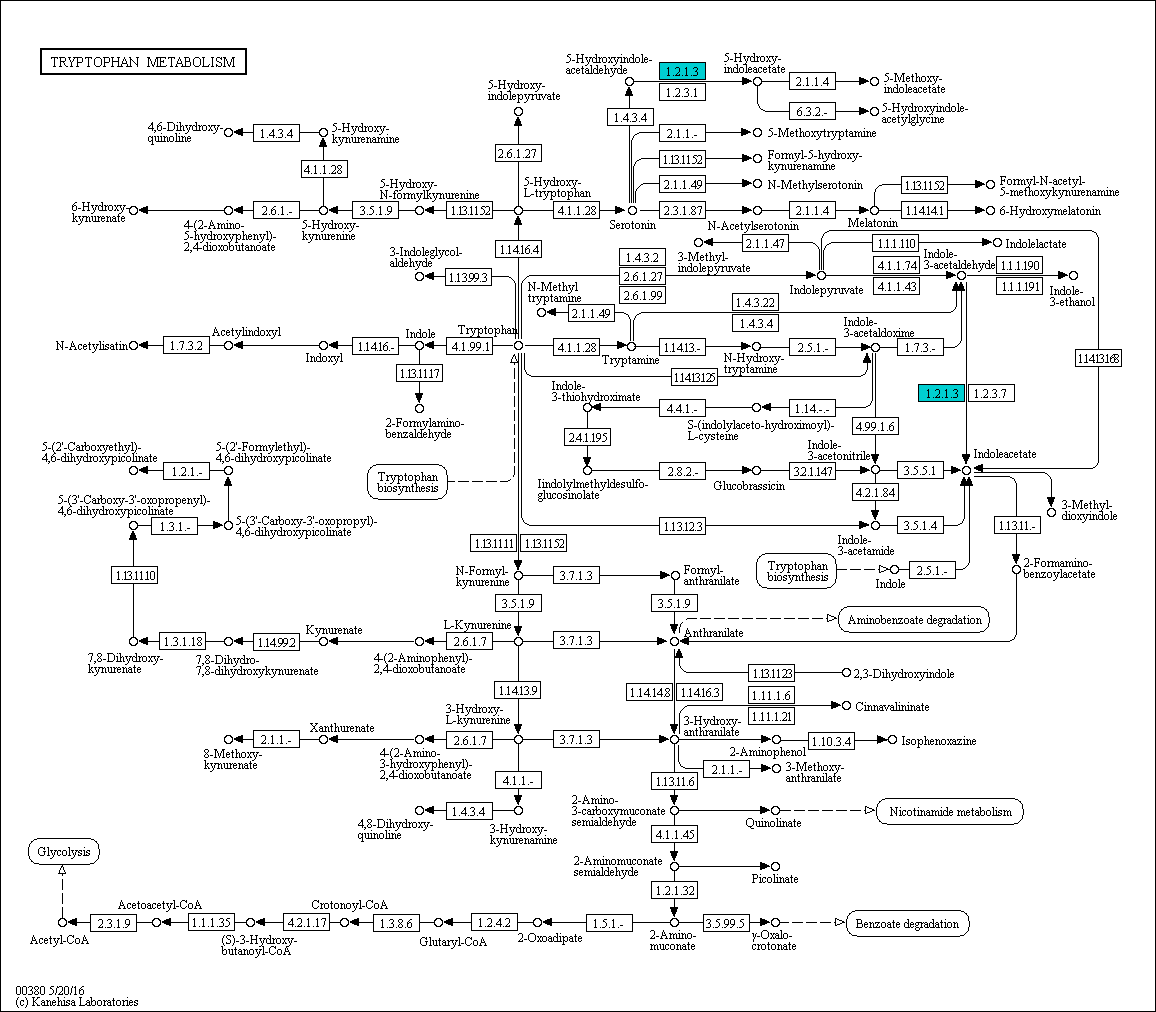 |
| Рис. 1. Путь метаболизма триптофана |
Получение совместного множественного выравнивания
Для каждого ортологического ряда KEGG были получены последовательности белков. По ссылке Uniprot со страницы
описания ортологического ряда была получена таблица их идентификаторов, которая была скопирована в Excel, а
затем вторая колонка этой таблицы была подана сервису "Retrieve/ID mapping" БД Uniprot.
С помощью небольшого скрипта на Python в названиях последовательностей были оставлены только идентификаторы и
информация об ортологическом ряде.
Полученнные файлы c белковыми последовательностями:
Проверка гомологичности белков в выравнивании
Выравнивание, полученное из всех имеющихся белковых последоваетльностей, не отличается хорошим
качеством. В нем имеются белки, значительно нарушающие общую картину.
Например, последовательность A0A158PZE7_BRUMA|K00149 гораздо длиннее прочих, и на всем протяжении
выравнивания имеет относительно мало совпадений с остальными. Поэтому она была удалена. Также встретились
и слишком короткие последовательности, содержащие много гэпов в тех участках, где в других
последовательностях выравнивания консервативные колонки, например B9TE04_RICCO|K14085, K7JC23_NASVI|K14085,
K1Q463_CRAGI|K00149, I7GA13_MACFA|K00149, I3L670_PIG|K14085 и
некоторые другие. Они также были удалены.
После удаления всех "подозрительных" последовательностей было получено следующее выравнивание
ali_before.fasta и JalView-проект
ali_after.jvp. Изображение итогового выравнивания можно представлено
на рисунке 3.
 |
| Рис. 3. Отредактированное выравнивание белковых последоваетльностей ортологических рядов K00149 и K14085, раскраска по ClustalX |
На мой взгляд, по полученному выравниванию можно с уверенностью говорить о гомологии белков, принадлежащих одному ортологическому ряду. Если смотреть только на последовательности в пределах одного ряда, можно увидеть достаточно крупные вертикальные блоки со значительным числом консервативных позиций. Однако сколько-нибудь больших консервативных блоков между последовательностями из разных ортологических рядов не обнаружено. Поэтому говорить о множественном выравнивании вообще в данном случае, скорее всего, ошибочно. Последовательности, принадлежащие разным ортологическим рядам, считать гомологами также нельзя.
Построение филогенетического дерева
Поскольку последовательности из разных ортологических рядов в моем случае получились негомологичны, то и строить
дерево, как мне кажется, нельзя, ибо оно не будет нести никакого биологического смысла.
P.S. Если выяснится, что я не права, то обязательно построю =)